1. 引言
在植物界中,十字花科是一种非常重要的科,其拥有375个属,3200个种,广泛分布于世界各地,其中在中国就有95个属,约411余种 [1] 。十字花科植物的种类繁多,既有富含食用价值的蔬菜,如白菜、甘蓝等植物,又有极具观赏价值的紫罗兰等植物,此外还有极高经济价值的油菜,亚麻荠等油料作物,是所有开花植物中最有经济价值的科之一 [2] 。更重要的是,由于十字花科植物拟南芥的生长周期短,结实多,广泛生长等优点,成为了植物界内研究最早的模式物种之一,在遗传育种等分子生物学研究中扮演着非常重要的角色 [3] 。近年来,在基因水平上通过一些分子标记来提高十字花科对人的营养利用价值,经济价值和科研价值的研究成为了一个热点 [4] [5] 。
简单重复序列(simple sequence repeats, SSR),又称为微卫星DNA序列,是广泛存在于整个基因组中的一类分子标记 [6] 。SSR一般定义为1~6个碱基串联重复组成几十bp的序列,其两端的序列是相对保守的单拷贝序列。SSR具有的广泛分布,共显性高,重复性好,多态性高,易于检测等生物学优点使得其成为一种常用的分子标记,并广泛应用于植物遗传多样性分析、遗传图谱构建、基因定位和分子标记辅助育种等领域 [7] [8] [9] 。但是据我们查询所知,现在对于十字花科植物SSR的研究仅局限于单个物种,并且仅利用表达序列标签(Express Sequence Tags,EST)序列检测的SSR并不完整 [10] [11] 。
随着现在第二代测序技术的发展成熟以及测序价格的降低,到目前为止,十字花科植物中阿拉伯岩芥菜 [12] ,玉山筷子芥 [13] ,拟南芥 [14] ,油菜 [15] ,甘蓝 [16] ,芜菁 [17] ,亚麻芥 [18] ,荠菜 [12] ,亚拉巴马水芹 [12] ,盐芥近缘种 [19] [20] ,水蒜芥 [12] ,小盐芥 [21] 等13个物种已经完成了全基因组测序并进行了基因的预测,这给本研究提供了丰富的数据基础。现在分子生物学技术以及生物信息学技术的发展,也给本研究提供了后续个性化分析技术和平台。本研究将基于十字花科物种的基因组信息,进行SSR标记标记的大规模开发和相关数据库的构建。
2. 材料与方法
2.1. 实验材料
从十字花科基因组数据库BRAD (http://brassicadb.org/brad/index.php) [22] 以及美国生物信息中心NCBI (https://www.ncbi.nlm.nih.gov/)等公共数据库中下载到十字花科物种的全基因组序列以及其基因序列,统计各个物种的详细信息(见表1)。
2.2. 方法技术
2.2.1. SSR位点扫描
利用Micro Satellite identification tool-MISA [23] 软件包中的Perl脚本进行批量扫描十字花科物种的SSR位点,本研究设定是否为SSR的标准为:1) 一单元至少十次重复,二单元至少六次重复,三单元至少五次重复,四单元至少五次重复,五单元至少五次重复,六单元至少五次重复。2) 两个SSR之间距离小于100 bp时组合为一个复合SSR。
2.2.2. SSR位点统计
利用Perl脚本对扫描得到的SSR做了以下分类统计:1) 各个基因组和基因序列SSR位点数目;2) 各个物种的1~6重复单元数目;3) 各个物种不同的重复单元前两位的重复片段数目。
2.2.3. 序列截取
计算每个SSR位点在基因组序列上的物理位置,利用SSR位置信息从每个十字花科物种的基因组上截取SSR起始终止位点前后各60 bp的序列,并将截取的序列中N值小于10%的序列筛选出来进行下一步引物的设计。

Table 1. The detail information of Brassicaceae species
表1. 十字花科物种详细信息表
2.2.4. 引物设计
将上述截取的序列利用Emboss (http://emboss.open-bio.org/)软件包中的e-primer3(http://emboss.bioinformatics. nl/cgi-bin/emboss/eprimer3)进行引物的设计,设置参数为:MinTM = 55,MaxTM = 80,MinGC = 40,MaxTM = 60,Product Size = 100~300,numberturn = 5。引物设计完成后将引物提取出并将重复的引物去除。
2.2.5. 引物验证
利用Electronic PCR-e-PCR (https://www.ncbi.nlm.nih.gov/tools/epcr/) [24] 将各个物种所有的引物在其各自的基因组上进行电子PCR模拟扩增,将能够在其物种上完整扩增的引物提取出来作为十字花科SSR的储备引物。
2.2.6. 特异性扩增引物
利用Shell语言提取在各个基因组中能够进行特异性扩增的引物,对所有特异性扩增引物进行数目和引物在基因组中的分布统计。
2.2.7. 通用扩增引物
将所有已验证过的SSR扩增的引物与所有的物种的基因组进行电子模拟PCR扩增,通过Perl以及Shell语言将在所有物种中均能够扩增的引物提取出来作为十字花科物种的通用SSR引物并提取出。
2.2.8. BSSRD数据库的构建
使用LAMP (Linux, Apache, MySQL, PHP/Perl)框架进行数据库的构建。首先通过Dreamweaver 2014CC软件构建用户使用的前台界面,将所有的SSR位点和引物信息导入MySQL后台数据库中,通过编写PHP和Perl脚本实现前台界面和后台数据的交互构建出十字花科SSR数据库BSSRD (http://biodb.sdau.edu.cn/BSSRD)。SSR开发以及引物设计技术路线见图1。
3. 结果与分析
3.1. SSR位点数目及特征
分别对十字花科物种进行了全基因组序列和基因序列的SSR扫描,在全基因组中共发现了1,786,619个SSR位点,在基因序列中共发现了63,915个SSR位点,占总基因组序列SSR的3.58%。在十字花科物种的全基因组序列中,平均每100,000,000个碱基中都会存在324~596.8个SSR。拥有两个较大基因组的物种B. napus和C. sativa其Genome-SSR和Gene-SSR也居于前两位。各物种Gene-SSR占Genome-SSR的比值在(B. oleracea) 1.86%~(A. lyrata) 6.14%之间趋于相对的平稳(见表2)。
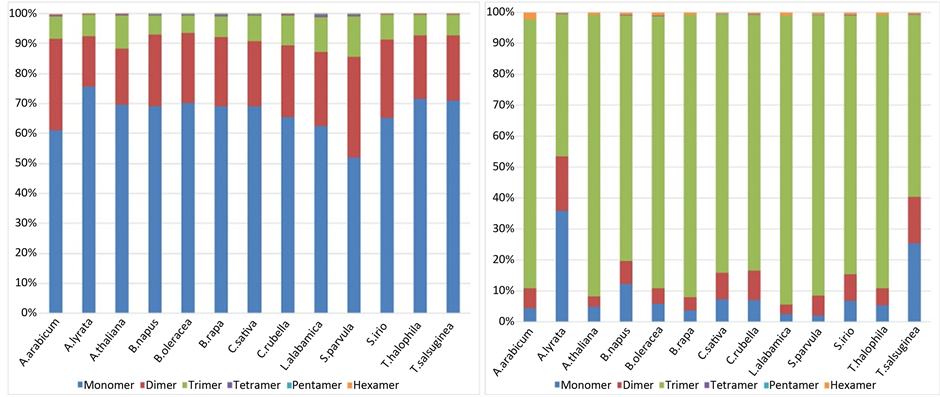
不同SSR重复单元具有较大的偏向性,在全基因组序列和基因序列中的SSR重复单元也存在差异。在全基因组序列中一单元重复的SSR数目占了总数目的一半以上为68.4%,其次是二单元重复和三单元重复的23.32%和7.59% (见图2(a)),然而在基因序列中的三单元重复的SSR数目高达79.47%,其次为一单元重复和二单元重复的11.12%和8.47%(见图2(b))。
对不同十字花科物种基因组序列的不同SSR重复片段分布频率做了一个统计,将每个物种每个重复单元数目前两位的重复片段提取出来。各个物种各个重复单元类型的前两位SSR片段基本上是相同的,其中二碱基重复的AT/TA,三碱基重复的GAA及TCT和四碱基类型的AAAT/TAAA在每个物种中数目都是非常高。在三碱基重复单元中大部分物种数目前两位为GAA和TCT,然而S. parvula物种为GAA和TTC (见表3)。
3.2. SSR引物设计及验证
利用十字花科物种的全基因组序列以及基因序列扫描到的SSR进行批量的设计了引物并进行了电子

Figure 1. The route of SSR development and primer design. 1) SSR development, primer design and confirm of universal primers; 2) Put all the data to MySQL; 3) The connection by PHP and Perl scripts; 4) Foreground display by Apache
图1. SSR开发以及引物设计技术路线图。1) SSR开发,引物设计以及特意引物和通用引物的确定;2) 将所有数据导入MySQL数据库中;3) PHP和Perl语言连接前台和后台;4) Apache前台页面展示

Table 2. The statistics of Genome-SSR and Gene-SSR in Brassicaceae
表2. 十字花科物种Genome-SSR和Gene-SSR数目表
模拟PCR扩增验证,最终,在全基因组序列中共有1,919,464对引物被确定,同时在基因序列中有54,619对引物被确定。在不同的物种的全基因组序列中引物的密度从(L. alabamica) 112.94对/Mb到(T. halophila) 1177.68对/Mb不等,在基因序列中为(A. arabicum) 42.65对/Mb到(C. sativa) 139.69对/Mb不等(见表4)。
3.3. 特异性引物和通用引物
在全基因组序列和基因序列中分别找到435,414和30,141对特异性扩增引物,同时11对能在十字花科物种中扩增的SSR引物被确定(Universal-primer-01~ Universal-primer-01),其中有三条来自于C. rubella,
 (a) (b)
(a) (b)
Figure 2. The distribution of different repeat unit in Genome-SSR and Gene-SSR
图2. Genome-SSR和Gene-SSR不同单元重复数目分布图

Table 3. The statistics of different SSR top two repeat units
表3. 不同SSR重复单元数目前两位的重复片段统计
S. irio和T. halophila分别占用两条,其余来自于B. oleracea,A. arabicum,S. parvula ,T. salsuginea分别占一条(见表5)。
3.4. BSSRD数据库的构建和应用
基于实验中的所有的数据包括物种的序列信息,SSR位点信息,e-PCR信息以及各类引物信息,构建了十字花科SSR数据库(BSSRD, Brassicaceae Simple Sequence Repeats Database http://biodb.sdau.edu.cn/BSSRD),该数据库包括SSR,Primer,Tools三个部分,用户可以在SSR和Primer界面中利用SSR ID和Primer ID等个性化搜索有目的的进行SSR及其引物的检索,同时在Tools界面能够使用一些简单的工具,包括界面化的

Table 4. Statistics of the SSR primers in Brassicaceae
表4. 十字花科物种中SSR引物的统计

Table 5. The detail information of 11 pair of universal primers
表5. 11对通用引物信息表
SSR的检测和e-primer3的在线使用(见图3)。
4. 讨论
由SSR检测结果可以看出,SSR广泛分布于各个十字花科物种基因组序列中,相比较其他分子标记来说,SSR是一种研究基因组的理想的分子标记 [25] 。从每个物种的Gene-SSR和Genome-SSR的比值来看,我们发现十字花科的物种拥有相对比较接近的SSR比例,这也从侧面揭示了十字花科物种SSR分布的广泛性和统一性。

Figure 3. The construction and use of BSSRD
图3. BSSRD的构建以及使用
通过对SSR各个碱基重复的比例以及SSR片段统计可以看出,一单元,二单元和三单元重复占了基因组和基因序列的大部分,并且AT/TA以及GAA/TCT所占比例非常高,在以后的十字花科遗传图谱的构建过程中,我们可以利用相对保守稳定的二单元和三单元重复 [26] 。
在各个十字花科物种中特异性扩增的引物对以后十字花科植物的基因的定位,遗传育种以及遗传图谱的构建能得到广泛的参考和应用 [8] 。同时11对通用引物被发掘出来也为以后十字花科同源物种和同源基因的鉴定提供了数据基础。
最后,基于本研究的所有数据,我们构建了十字花科SSR数据库BSSRD储存我们所得到的所有SSR位点以及设计的各种引物信息来用作非商业性质的科学研究。在该数据库中在线的SSR扫描工具以及引物设计的交互式界面也给科研工作者提供了很大的便利。当有新的基因组以及基因数据产生时我们会及时的去更新该数据库。我们相信这个数据库在以后的关于十字花科的遗传图谱的构建,基因定位,基因组组装和遗传育种等研究中将发挥重要的作用。
5. 结论
利用现有的十字花科物种的全基因组序列和基因序列,批量扫描了其中的SSR位点,进行了各项统计分析,对这些位点进行了引物设计以及特异性引物和通用引物的查找,最后我们构建了BSSRD十字花科数据库来储存这些数据。
基金项目
本研究得到了山东省自然基金(ZR2015CL006)支持。