1. 前言
在生物医疗研究领域中经常需要测量端粒长度 [1] [2] [3]。由于在疾病机理研究、用药需求以及健康人群中,对端粒长度和端粒酶活性检测的需求越来越多 [1] [2] [3],我们认为需要开发一种可提高准确性、提高通量及降低成本的方法,为提高检测服务精准度。到目前为止,有多种方法可以测量端粒长度,如Q-FISH、STELA、TeSLA和传统的Southern印迹法等均可测量端粒的末端限制性片段(Terminal Restriction Fragment: TRF) [4] [5],但这些方法都是低通量且繁琐、耗时耗力。荧光定量PCR方法,即单色双管定量PCR方法可检测端粒相对平均长度,这种方法省时、操作简单,而且是小样本量检测,通量高。然而,这种方法也有缺陷,主要是端粒和内参在不同管中反应,从而产生加样误差和不同反应孔的差异 [2]。为了减少人为产生的误差,在此我们比较在单管内同时检测端粒和内参是否会减少这种差异,从而提高检测的准确性。
2. 材料与方法
2.1. 一般资料
参加本次研究的志愿者共22例,均签署知情同意书,年龄在50 ± 2岁。
2.2. 实验方法
2.2.1. 标本的采集
抽取22位病人静脉血,置无菌EDTA抗凝管中,颠倒抗凝管使全血和抗凝剂充分混匀。
2.2.2. 全血基因组DNA的提取
取200 μL全血,按照血液提取试剂盒进行基因组DNA提取(天根DP348),提取好的DNA用NanoDrop 2000 (美国Thermo)测定浓度和纯度,基因组DNA保存于−20℃备用。
2.2.3. 单色单管SYBR Green荧光定量PCR方法建立
1) 引物设计:用Primer5软件,根据端粒的序列(参考NCBI 的NG_009265)设计端粒引物,并由通用公司合成引物如下:T-F:ACACTAAGGTTTGGGTTTGGGTTTGGGTTTGGGTTAGTGT; T-R:TGTTAGG TATCCCTATCCCTATCCCTATCCCTATCCCTAACA。作为内参的单拷贝基因(参考NCBI的白蛋白序列NG_009291)的引物设计,为了提高产物的溶解温度,F和R共设计了50个GC夹钳结构,具体引物序列如下:S-F: CGGCGGCGGGCGGCGCGGGCTGGGCGGaaatgctgcacagaatccttg; S-R:GCCCGGCCCGCCGCG CCCGTCCCGCCGgaaaagcatggtcgcctgtt [2];2) 标准品设置:标准品DNA最高浓度210 ng/μL,按1:3倍比稀释成210、70、23.33、7.78、2.59 ng/μL;3) 荧光定量PCR反应体系:反应总体积为20 μL,包括1×SYBR Green Realtime PCR Master Mix (TOYOBO公司) 10 μL,DNA (样本和标准品) 2 μL,端粒引物T-F、T-R终浓度均为400 nmol/L;单拷贝基因引物al-F、al-R终浓度均为400 nmol/L。反应在同一块96孔板上进行,标准品和待测样本均设置3复孔。另外设置以相同体积的超纯水作为DNA的阴性对照,亦为3个复孔。在ABI 7500实时荧光定量PCR仪(赛默飞)上检测。4) 反应条件为I:95℃ 15 min;II:94℃ 15 s,49℃ 15 S,2个循环;III:94℃ 15 S,62℃ 10 S,74℃ 32 s (获取端粒信号),84℃ 10 S,88℃ 32 s (获取内参基因信号),32个循环。反应结束后建立熔解曲线。
2.2.4. 双管法SYBR Green荧光定量PCR方法建立
1) 引物:应用单管法的引物,包括T-F,T-R和S-F,S-R;2) 标准品设置:标准品DNA最高浓度210 ng/μL,按1:3倍比稀释成210、70、23.33、7.78、2.59 ng/μL;3) 荧光定量PCR反应体系:端粒管反应总体积为20 μL,包括1×SYBR Green Realtime PCR Master Mix (TOYOBO公司) 10 μL,DNA (样本和标准品) 2 μL,端粒引物T-F、T-R终浓度均为400 nmol/L;内参管反应总体积为20 μL,包括1×SYBR Green Realtime PCR Master Mix (TOYOBO公司) 10 μL,DNA (样本和标准品) 2 μL,单拷贝基因引物S-F、S-R终浓度均为400 nmol/L。反应在同一块96孔板上进行,标准品和待测样本均设置3复孔。另外设置以相同体积的超纯水作为DNA的阴性对照,亦为3个复孔。在ABI 7500实时荧光定量PCR仪(赛默飞)上检测。4) 反应条件为I:95℃ 1 min;II:95℃ 15 s,60℃ 40 S,40个循环。反应结束后建立熔解曲线。
2.3. 分析方法
本研究采用SPSS17.0软件的one way ANOVA得到均值,方差和差异系数。端粒的相对长度是由T/S值决定的,T/S = 端粒信号(T)/内参单拷贝基因信号(S),T/S相对值是和端粒的平均长度成正比的,从而决定端粒的相对长度。
3. 结果
3.1. 单色单管和双管法SYBR Green荧光定量PCR检测端粒 相对长度的标准曲线
两种方法分别对标准DNA样品(210 ng/μL)按1:3稀释成210、70、23.33、7.78.2.59 ng/μL的5个点进行检测,结果见表1:
从表上可以看出,把两对引物放入同一管中检测并没有影响扩增效率以及标准曲线的线性关系。

Table 1. Comparison of the parameters for standard curves between two methods
表1. 两种方法标准曲线的参数比较
3.2. 单色单管和双管法SYBR Green荧光定量PCR检测端粒相对长度的熔解曲线
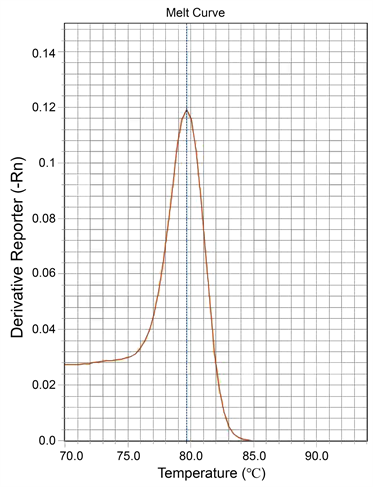
溶解曲线见图1,A单色双管的T峰,由于端粒和内参在不同的管中反应,所以不能同时出现T和S,从图可以看出双管T峰在80℃左右;B单色单管由于T和S在同一管里反应,所以溶解曲线出现两个峰,T峰还是在80℃左右,峰形单一正常没有杂峰,说明两对引物放在一管反应没有改变端粒引物的特异性,T峰在同样的温度溶解并峰形单一无杂峰。
 (a)
(a)  (b)
(b)
Figure 1. (a) Telomere dissociation curve of reactions in two separate tubes; (b) Telomere dissociation curve of reactions in single tube
图1. (a) 双管法的端粒溶解曲线;(b) 单管法的端粒扩增溶解曲线
3.3. 单色单管和双管法SYBR Green荧光定量PCR检测22例样本的端粒相对长度情况
比较了两种方法的标准曲线和溶解曲线都没有差异的情况下,用这两种方法检测了22例样本,每个样本每种方法都平行检测3次,通过计算得到每个样本的T平均拷贝数和S平均拷贝数,两种方法得到的T/S值进行比较P < 0.05,T/S结果分布见图2,从图直观可见两种方法检测同一样本的结果差异很大。
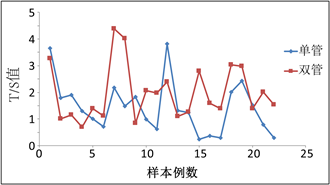
Figure 2. Distribution of T/S values of 22 samples detected with two methods
图2. 两种方法检测22例样本的T/S值的分布情况
3.4. 单色单管和双管SYBR Green荧光定量PCR法检测样本的重复性比较
为了确定两种方法对同一样本检测的差异,我们对同一样本分别用两种方法检测10次,看看哪种方法的重复性更好,结果见表2。

Table 2. Comparison of the reproducibility between the two methods
表2. 两种方法重复性比较
两种方法10次检测同一样本的T/S值的平均值进行比较发现,P < 0.05,即不同方法得到的结果差异很大,并且从表上的CV%值看,单管法重复性好,而双管法重复性很差。重复性高,说明检测结果准确,可靠。
4. 讨论
单管法实时荧光定量PCR测定端粒长度,将端粒引物和内参引物置于同一个PCR管中扩增,利用端粒和内参在扩增时Tm值的不同,可以在同一反应中,不同的温度情况下分别收集端粒荧光信号和内参荧光信号,从而能同时得到T和S值 [1] [2]。
该方法与传统多重PCR不同的是两个目标基因的扩增并不是同时进行。即理论上,当两个目标序列在同一扩增体系中,合成所需温度有很大差异时,在同一个qPCR系统中可以分别收集来自两个具有非常不同拷贝数的成对核酸目标产物的信号。在低拷贝的目标序列的信号仍处于基线时,高拷贝的目标序列荧光信号可以较早收集到信号。本方法通过引物设计,端粒首先扩增并在74℃收集信号。由于端粒的拷贝数比单拷贝基因高,此时单拷贝基因的荧光值仍在基线水平以下,因此74℃收集到的信号仅代表端粒。温度继续升高到85℃,此时端粒产物已完全解链,聚合酶只用于单拷贝基因的扩增,因此此时收集的荧光信号仅代表单拷贝基因产物的信号 [2]。
单管和双管方法比较,消除了加样误差和批内误差对结果产生的影响,同时试剂和模板用量以及时间均减少了一半,节约了经济成本和时间成本;进一步提高了QPCR端粒长度测量的准确性和通量,降低了成本。通过对两种方法的对比分析,单管法获得的T/S结果在独立运行中具有更高的可重复性,对端粒长度测定以及端粒疾病相关的药物研究都具有重要参考价值 [6] [7] [8]。另外,这种方法可以很容易地适用于许多自然发生在不同拷贝数的DNA模板的研究,例如mtDNA与单拷贝基因,rDNA与单拷贝基因,Alu-DNA与单拷贝基因。类似地,在反转录成cDNA之后,具有非常不同拷贝数的成对RNA物种可以通过这种方法进行量化 [7] [8]。
参考文献