1. 引言
癌症的致死率非常高,是世界范围内的主要死因之一。而癌症预后可以帮助医生对患者进行诊断,针对不同癌症患者制定相应的治疗方案。近年来,利用深度学习进行癌症预后取得了较大进展。癌症预后是一项极具挑战性的工作,不仅要考虑肿瘤组织的特征,还需要考虑肿瘤组织周围的特征,以评估患者的死亡风险 [1] [2] 。
近年来,很多深度学习的方法被用来用于全视野数字切片(WSI)的生存分析。比如,在神经网络的最后一层使用COX比例风险函数作为输出,进行预测 [3] ;或者使用K-Means方法,在WSI水平上进行聚类,作为卷积神经网络的输入进行预测 [4] 。
本论文使用了图卷积神经网络进行主要模型,用于预测胃癌和结直肠癌患者的生存预测,将WSI切片抽象为图,作为模型的输入。与其他深度学习的方法相比,图卷积神经网络不仅学习WSI中每个切片的特征,还将此切片及相邻切片的特征进行组合学习,具有较好的周围环境感知能力 [5] 。
2. 数据选择与预处理
2.1. 数据选择
本实验用到的数据集为胃癌(STAD)和结直肠癌(COAD)的WSI数据集。以上数据均来自 https://portal.gdc.cancer.gov/网站。
WSI的全称为Whole Slide Image,名称为全视野数字切片。这种图像的像素很大,通常以万为级别。本实验所用到的数据是胃癌和结直肠癌的WSI图片。由于本文研究的问题是胃癌和结直肠癌的1年期生存率,所以用到了癌症病人的WSI图片以及生存信息。每个癌症病人可能具有多个WSI图片,而每个癌症病人具有各自的生存信息,信息包括:样本生存时间(OS.time),是指从癌症病人确诊到最后一次随访的时间;生存状态(OS),这里的OS取1或0,1代表该病人为死亡状态,0代表病人为存活状态;生存时间(survival_months)指的是病人从确诊到最后一次随访的时间,以月为单位。根据OS.time和OS规定该病人的最终标签(label),判断标准见图1所示:
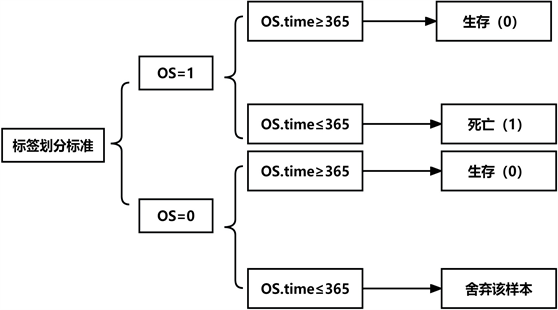
Figure 1. Criteria for dividing patient survival
图1. 病人生存划分标准
2.2. 数据预处理
对于胃癌和结直肠癌的WSI图片,如图2所示,我们采用以下流程进行预处理:
2.2.1. WSI图像切割及特征提取
WSI (whole slide images),名称为全视野数字切片,一般是通过对癌肿病理图像进行扫描得到。WSI图像的像素非常大,通常以万为单位,一张WSI图像通常会占用几十MB到几G不等。一张WSI图像包含了很多病理学信息。如图3所示,这是一张结直肠癌的WSI图像。

Figure 3. WSI image of colorectal cancer
图3. 结直肠癌WSI图像
对于像素巨大的WSI图像,我们采用OTSU算法进行切割。OTSU算法主要是通过将上述类型的WSI图像进行灰度级划分,再采取划定阈值的方法将图像中病例部分分割出来,以实现图像的分割,分割之后的切片用来进行特征提取。
将WSI进行分割之后,我们使用在ImageNet数据集上预训练好的Resnet50网络对WSI图像切片提取出了1024维的特征。
2.2.2. Graph的构造
我们在患者层面上构造WSI的Graph。对于一张WSI的若干切片,将这些切片定义为在WSI二维坐标上的点,这些点构成了WSI图的节点;Resnet网络提取的1024维特征作为每个节点的特征;将每个切片与其在二维坐标平面上周围8个点进行连接,作为WSI图的边。
如下图所示,以红色节点为例,将周围的8个节点进行连接,作为整张WSI图片的边。一个患者可能具有多张WSI图片,将患者的多个WSI图片分别进行如下图的构造,之后再将这几张WSI图片构成的图作为子图,形成该患者的Graph。图4为WSI图片节点和边的构造示例。

Figure 4. Illustration of the WSI diagram
图4. WSI图示意
2.2.3. 网络训练
我们采用Patch-GCN [5] 网络进行训练。由于本文构造的患者的每张WSI图片上的每个节点与其周围的8个节点相连,连边密度比较大,网络每增加一层,每个节点之间的邻居会增加很多,从而加重训练负担。另外,由于GCN的传播机制,每个节点在网络层数增加的过程中,聚合道德信息也会重复性增加,从而导致训练资源浪费。根据以上考虑,我们将图卷积神经网络的层数设置为4层,并在网络的最后一层使用Cox函数进行回归,进行生存分析。
3. 实验结果
我们使用了胃癌和结直肠癌的数据进行Graph的构造和图卷积神经网络的训练。使用数据为胃癌和结直肠癌的WSI图像数据以及两种癌症患者5年期生存数据,将患者层面的数据按照训练集和测试集4:1的比例进行5折交叉验证,训练的到的5个癌肿C-index值进行平均,作为该癌肿最后的C-index值,并绘制了两个癌肿的AUC曲线,如图1所示。
另外,为了进一步说明本文使用的图卷积神经网络(GCN)的性能,我们将原始数据(WSI)同样进行卷积神经网络的训练,模型为Ruoyu Li等人提出的graph CNN [6] 网络。如表1所示,最终得出使用图卷积神经网络进行预测的胃癌和结直肠癌的C-index值分别为0.65和0.58。实验结果证明本文使用的图卷积神经网络(GCN)在预测胃癌和结直肠癌的生存概率的性能上的确优于卷积神经网络。

Table 1. County level planning schedule
表1. 县域等级规划一览表
此外,我们将两个癌肿的预测结果绘制了AUC曲线,如图5和图6所示。
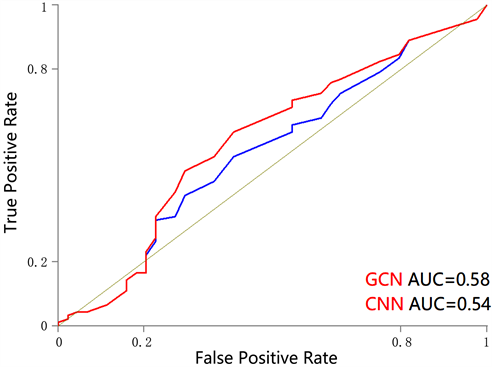
Figure 5. AUC curve for predicting survival probability of colorectal cancer (COAD)
图5. 结直肠癌(COAD)生存概率预测AUC曲线
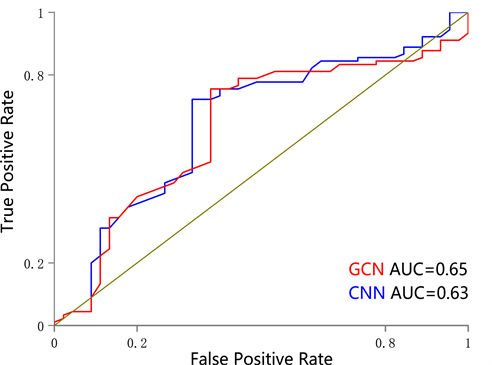
Figure 6. AUC curve for predicting survival probability of gastric cancer (STAD)
图6. 胃癌(STAD)生存概率预测AUC曲线
4. 方法原理
4.1. 图卷积神经网络
在图卷积神经网络(GCN)提出之前,深度学习一直都是以卷积神经网络(CNN)为主。CNN的核心在于卷积核通过在输入图片上平移进行类似点乘的操作来进行图片的特征提取。而GCN的核心则在于通过图上的节点和边的连接来进行节点之间的特征融合。GCN在一定程度上达到了各个节点特征之间的特征融合。
GCN模型的输入是图数据,假设每张图片具有N个节点和M条边,每个节点都有D维特征,那么每个节点都会有D × D维的特征矩阵X,N个节点构成的邻接矩阵A和特征矩阵X就是GCN的输入。本文使用的GCN网络共有4层,每一层都相当于一轮特征学习过程。
4.2. C-index
C- index全称为一致性指数(Concordance Index) [7] ,最早于1996年在范德堡大学的Frank E Harrell Jr.教授提出,通常被用来评估生存模型的预测结果。C-index的计算方法如下:将n个病人两两结对,数量为C(n, 2),将预测生存概率高低与病人实际生存状态相一致的对数除以C(n, 2),得到的比例就是一致性指数,它实质上计算出了预测结果与实际状态相一致的概率。根据以上计算方法,一致性指数的值应该在0.5至1之间。
5. 结论
本文对胃癌(STAD)和结直肠癌(COAD)的全视野组织图像(WSI)进行了切割、特征提取等预处理操作;预处理结束后,对每一张WSI进行了图的节点和边的构造;最后采用图卷积神经网络,对两个癌肿(胃癌和结直肠癌)的五年生存信息进行了生存分析,最终得到胃癌的C-index值为0.65,结直肠癌的C-index值为0.58,较卷积神经网络的结果有所提升。
6. 讨论
在以往关于WSI图像的生存分析中,大多数都是使用卷积神经网络(CNN)进行训练,从而得到结果。比较常见的有多实例学习(MIL)方法、弱监督方法,这次方法虽然能解决WSI上很多分类、回归任务,但并未达到WSI“全局”学习。也就是说,之前的方法并未将WSI中不同切块之间的特征进行整合学习;而图卷积神经网络(GCN)却很好地改进了这一点,其利用WSI层面的图结构,将每个切片看作图中的节点,通过节点之间的边的连接,完成了节点之间特征学习,从而具有全局性,在一定程度上也可以看作“多尺度”的学习。