1. RNA修饰与tRNAIle反密码子胞嘧啶Cyt34的修饰
目前已经发现自然界中有百余种RNA的修饰,在三界生物系统中均有存在。其中tRNA的修饰种类最多样 [1] [2],对于正常生命活动过程有很重要的作用。有些tRNA的修饰,改变了tRNA的性状,使其更加稳定不易被RNA酶降解。有一些修饰改变了tRNA的身份,使其可以被其它的氨酰tRNA合成酶识别,携带另一种氨基酸。真核生物用tRNAIle第34位的假尿嘧啶(pseudouridine)或者肌苷酸碱基(inosine),来识别AUA密码子的腺嘌呤碱基 [3]。tRNAIle反密码子中第34位胞嘧啶Cyt34上的修饰对于细菌和古菌AUA密码子的解码都是必需的。细菌中TilS蛋白使用赖氨酸来修饰tRNAIle前体的Cyt34,从而改变了tRNAIle对于氨酰tRNA合成酶以及密码子的特异性识别 [4] - [9]。
细菌的TilS蛋白催化tRNAIle前体的Cyt34修饰后,形成包含新碱基Lysidine34的新型反密码子LAU,可以识别AUA密码子,而不修饰的此tRNAIle前体可以被甲硫氨酰tRNA合成酶(MetRS)加上甲硫氨酸,并识别AUG密码子 [4] [10]。TilS识别tRNA的接受臂位置,包括核苷酸Cyt29-Gua41,还识别反密码子环附近的Ade35、Ura36和Ade37等核苷酸 [8]。更有趣的是,如果将tRNAMet的接受臂替换成为tRNAIle的接受臂,此tRNA可以被TilS进行修饰,生成可以与Ade配对的Lysidine碱基。所以通过逻辑推理可以想象,细菌内必然有一定的机制来保证TilS对tRNAIle前体的修饰及时快速的进行,以避免MetRS错误的将甲硫氨酸连接到未修饰的tRNAIle前体上面,造成遗传信息表达的错误。将大肠杆菌的RNaseE敲除掉,抽提其中的tRNA,分离纯化出来其中的tRNAIle,使用RNaseT1进行水解,质谱分析,发现所有成熟的tRNAIle都已经被修饰成Lysidine34,而未成熟的tRNAIle多数也已经被lysine修饰,只有少量的尚未被修饰(第34位仍然以胞嘧啶Cyt34的形式存在) [8]。所以推测,细菌内对于tRNAIle的修饰是在其刚被转录出来的前体时期就开始进行的,在其成熟时保证被修饰,从而保证遗传信息的高保真表达 [8]。
依据密码子表(附表1),AUA密码子编码异亮氨酸。在以Archeoglobus fulgidus为代表的很多古菌中,前人通过一系列基因组序列分析与实验只发现了反密码子为CAU/CAT的tRNA基因,并没有发现反密码子是UAU/TAT的tRNAIle基因。以古菌Archeoglobus fulgidus为例,2009年以前已确知的唯一的Af-tRNAIle是tRNA数据库中编号为tdbD00005900的Af-tRNAIle1 (http://trna.bioinf.uni-leipzig.de/DataOutput/Search),反密码子是GAU,解码AUC并编码异亮氨酸。根据已知,存在编码异亮氨酸的AUA密码子,可以推测存在解码AUA密码子的Af-tRNAIle,在A. fulgidus基因组里并没有发现包含反密码子UAU/TAT的tRNA基因,只发现了包含反密码子为CAU的tRNA基因,推测存在一定的修饰或者编辑等生物学过程使得Cyt与Ade能够很好的配对,从而解码AUA密码子。1984年科学家在Halobacterium volcanii古菌中发现了结构未知的tRNAIle胞嘧啶修饰 [11]。这是发现新型tRNA反密码子修饰的其中一种方法。Haloarcula marismortui,Methanococcus maripaludis,Sulfolobus solfataricus,Sulfolobus tokodaii,Archaeoglobus fulgidus等古菌细胞内分离获得转录出来的包含反密码子CAU的tRNA,对其进行质谱分析,发现了新型的tRNA修饰 [12] [13],经过质谱等实验鉴定Af-tRNA,这就发现了胞嘧啶的胍基丁胺修饰。这些古菌中没有类似于细菌TilS的同源蛋白 [4]。
2. tRNAIle2修饰酶TiaS蛋白
TiaS蛋白(tRNAIle2 agmatidine synthetase)是来自于古菌中的酶,功能是在tRNAIle2前体反密码子CAU的Cyt34上面加上胍基丁胺Agmatine修饰,从而使其不识别AUG,而识别AUA密码子。2010年Tsutomu Suzuki及Uttam RajBhandary等,在Euryoarchaeota,Crenarchaeota和Thaumarchaeota等中,发现了古菌的AUA解码系统 [12] [13] [14]。该系统使用Agmatine这一个在哺乳动物神经系统中也存在的多氨基化
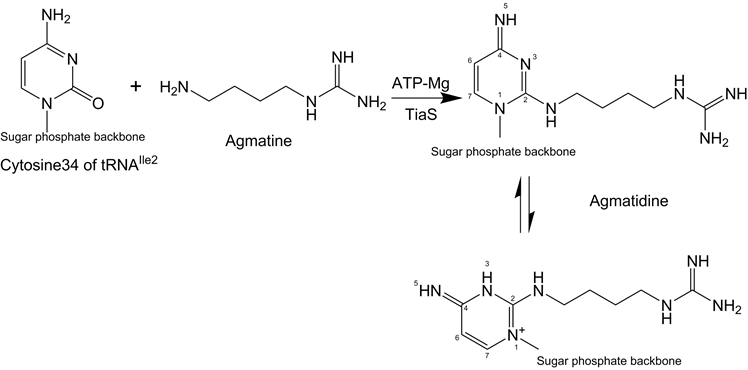
Figure 1. TiaS enzyme catalyzes the Agmatination of Cyt34 of tRNAIle2 with the presence of ATP
图1. TiaS酶利用ATP催化Agmatine修饰到tRNAIle2的Cyt34
合物 [15] [16] 来修饰tRNAIle2前体,将Agmatine的氨基共价连接到Cyt34嘧啶环的2’碳原子上,生成的碱基被称为Agmatidine或者Agm2C。此共价修饰使嘧啶环的3’氮原子发生质子化,这样在碱基配对时就能够与带负电荷的原子形成氢键,而发生质子化前形成氢键的话是通过接受质子来形成的。C4原子上面连接的氨基转变成为亚氨基,从而改变了氢键配对的形成方式,如图1。进一步实验发现,此反密码子为CAU的tRNAIle2前体经过TiaS蛋白的修饰后,生成可以解码AUA密码子的成熟tRNAIle2 [13]。没有修饰的tRNAIle2前体会被连接上甲硫氨酸,而不是异亮氨酸。催化这个反应的酶就被命名为TiaS。在这些微生物细胞中,刚转录生成的tRNAIle2前体需要迅速被修饰成熟,以保证在合成蛋白质时遗传信息的高保真性传递。确实,TiaS蛋白可以修饰未经剪切的含有5’与3’冗余序列的tRNAIle2前体(precursor tRNAIle2) [13] [17] [18]。TiaS蛋白的活性存在自然选择压力,会向催化速率更高的方向进化,所以,经过自然界长期的自然选择,野生型的TiaS蛋白,在保证底物特异性、蛋白高温稳定性等要求的条件下,应该是朝着其能够达到的最高酶综合活性趋近。在细胞中,酶(TiaS蛋白)、底物(tRNAIle2前体、Agm)、酶–底物复合物、辅因子ATP等的浓度可能与传统酶学方程中的假设有所不同。TiaS具有较高的底物选择性 [19],不像TilS可以将许多种不同底物类似物分子修饰到tRNA上面。
Af-TiaS蛋白的来源物种古菌A. fulgidus是嗜热菌,最适生长温度为83℃,可以生存的温度范围是约60℃~95℃。Af-TiaS蛋白有420个氨基酸残基,理论分子量约48 kDa,理论等电点5.7。目前有Archeoglobus fulgidus物种来源的TiaS蛋白的若干个晶体结构得到解析 [17],有力的推动了对于tRNAIle2修饰成熟机制的理解。TiaS蛋白有4个结构域,酶催化功能主要由N端3个结构域组成的酶核心完成,它们可以结合tRNAIle2的反密码子环。Tsutomu Suzuki等解析的TiaS蛋白结合tRNAIle2的两个晶体结构(3AMT.pdb,TiaS-tRNAIle2-ATP;3AMU.pdb,TiaS-tRNAIle2-AMPCPP-Agmatine),第4个结构域都是无序的,看不到半胱氨酸以及鳌合的金属原子 [17]。另外一个结构不结合tRNA (3AU7.pdb),去除了整个锌指结构域,是只有酶N端3个结构域的截短体 [17]。中国科学家解析了首个自由全长的单独的TiaS蛋白结构(3U02.pdb,5XOB.pdb),首次展示了TiaS蛋白完整的第4个结构域 [19] [20],如图2。TiaS蛋白C端的第4个结构
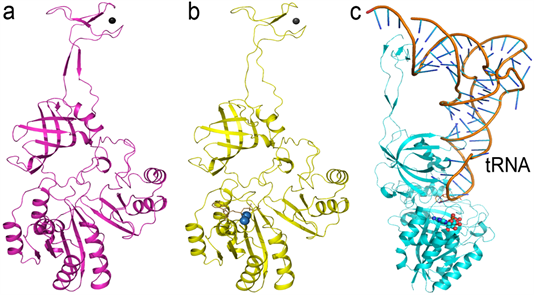
Figure 2. Typical 4 domain TiaS crystal structures solved until now. a. Free full-length TiaS (5XOB.pdb, 2.5Å). b. Crystal structure of TiaS complexed with Agmatine and ATP analogue AMPPCP (6AGG.pdb, 2.7Å). c. Crystal structure of TiaS-tRNAIle2-ATP complex (3AMT.pdb, 2.9Å). Both a and b show intact zinc ribbon, and the major difference between a/b and c is the conformation of zinc ribbon domain. There is minor difference between a, b and c in the conformation of ATP binding P2-loop (Reprinted with permission from ref. 20, Copyright 2018, Elsevier)
图2. 目前解析的典型的4个结构域TiaS蛋白的晶体结构,a. 自由单独的TiaS蛋白结构(5XOB.pdb,2.5埃分辨率),b. 结合了ATP类似物AMPPCP与Agmatine的TiaS蛋白结构(6AGG.pdb,2.7埃分辨率),c. TiaS-tRNAIle2-ATP复合物晶体结构(3AMT.pdb,2.9埃分辨率)。ab具有完整的锌指结构。a/b与c之间的主要区别在于锌指结构域的构象,abc之间P2-loop的构象也有区别(摘自ref. 20)
域是锌指结构域(zinc ribbon domain, ZRD),锌指通过一段长长的反向平行β折叠片与酶核心相连接,距离催化中心较远,锌指识别结合底物tRNAIle2的接受臂。不同古菌物种来源的TiaS蛋白序列比对发现,TiaS的锌指序列特征是CX2CX14CX2C。四个保守的负责鳌和锌原子的半胱氨酸残基是Cys352,Cys355,Cys370,Cys373。TiaS蛋白的4个结构域均可以找到同源结构,比如TiaS锌指结构域与RNA聚合酶II的锌指具有较高的三维结构相似性。但是这四种结构域的组合(TCKD,FLD,OBD,ZRD)是在TiaS蛋白家族中首次发现的,如图2。嗜热菌来源的TiaS蛋白有非常大范围的氢键网络和疏水相互作用,可能对于适应高温环境、稳定蛋白结构有重要作用。
3. TiaS蛋白的构象
依据锌指结构的差异,目前解析的TiaS蛋白晶体结构有两种主要的构象,一种主要构象是不结合底物tRNA的TiaS蛋白构象,硒代蛋白的结构与Native的结构相同,同时结合了AMPPCP与胍基丁胺Agmatine的TiaS蛋白晶体结构与自由的TiaS蛋白晶体结构基本相同,结合了另一种不同的ATP类似物AMPCPP的构象与此锌指构象也基本相同 [20],如图2。第二种主要构象是结合了底物tRNAIle2与ATP的构象(3AMT.pdb),结合了Agmatine形成的TiaS-tRNAIle2-AMPCPP-Agmatine四元复合物晶体结构(3AMU.pdb)与这一个三元复合物的结构基本相同 [17]。这两种主要构象之间的差别在于锌指结构的差异,如图3。引起这种锌指构象差异的主要原因是底物tRNAIle2的结合与否。除了这两种主要的构象之外,结合辅因子ATP或者ATP类似物与否,P2-loop会呈现不同的构象 [20]。不同古菌物种来源的TiaS蛋白序列比对发现,负责ATP结合的P2-loop比较保守。自由的TiaS蛋白,解析的结构中没有观察到负责ATP结合的P2-loop电子密度,表明了其构象的不均一性。结合了辅因子ATP或者ATP类似物以后,可以观察到P2-loop的主链,侧链不清晰 [20]。结合了底物tRNAIle2与ATP的构象显示,P2-loop主链侧链均可以清晰的观察到(3AMT.pdb)。tRNAIle2的反密码子环从一侧结合P2-loop,ATP从另一侧结合P2-loop。从这几个结构的不同之处,以及P2-loop的变化过程可以看出来,自由的P2-loop存在柔性,构象不均一,夹在中间的P2-loop随着两侧底物、辅因子的结合,构象逐步稳定下来。这几个结构的分辨率较为接近,如图2,图3。
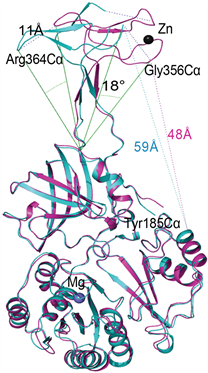
Figure 3. The difference between the two major conformations discovered by crystallography (Reprinted with permission from ref. 20, Copyright 2018, Elsevier)
图3. 蛋白质晶体学发现的TiaS蛋白的两种主要构象的区别(摘自ref. 20)
4. TiaS蛋白的分子动态特征
TiaS蛋白在不同状态下构象的差异,表明了TiaS蛋白分子很可能存在结构的动态变化现象。分子动力学模拟(Molecular dynamics simulation),对于运动轨迹的主成分分析(Principle component analysis),常规模态分析(Normal mode analysis) [21] 等方法都可以用来研究蛋白质分子的动力学特征。
研究表明,三种方法的研究结果基本一致 [20]。首先,TiaS蛋白分子结构动态变化的主要位置均显示是在锌指结构域,这与蛋白质晶体学实验观察到的两种主要不同构象的差异位置是一致的 [20]。其次,TiaS蛋白分子动态变化的模式,三种方法都显示,是TiaS蛋白分子锌指结构域与酶核心连接的linker扭动或者伸展收缩运动引起的锌指结构域的大幅度构象变化 [20]。第三,TiaS蛋白锌指结构运动的方向,结合底物tRNAIle2的构象显示,锌指结构域比自由的TiaS蛋白朝向OBD结构域方向摆动了一些,锌指距离酪氨酸Tyr185更远,同时,锌指结构域有一定角度的扭动。总体来说,结合底物tRNAIle2的构象其锌指朝向底物结合的方向进行了大幅度的运动,如果以自由的TiaS蛋白结构与底物tRNAIle2构建的复合物为起始模型,分子动力学模拟显示,锌指结构域也朝向底物tRNAIle2方向运动 [20]。下面总结常规模态分析给出的TiaS蛋白分子的运动特征 [22] [23]。
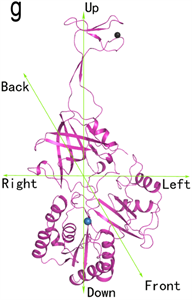
Figure 4. Normal mode analysis of free TiaS molecular dynamics. abc are views from the top of zinc ribbon along the long axis of Af-TiaS protein. ad, be, cf are molecular dynamics of the first, the second, and the third slow normal modes, respectively. def, side views of Af-TiaS protein, with the catalytic center facing outward. g, the artificially designated directions of Af-TiaS protein
图4. 常规模态分析自由TiaS蛋白的分子动力学特征。abc,从锌指一侧沿着Af-TiaS蛋白的长轴俯视图;ad,be,cf分别对应展示第一二三种模态的动力学特性。def,Af-TiaS蛋白侧视图,催化活性中心朝向纸面外。g,为方便讨论,人为给Af-TiaS蛋白指定的方位图
TiaS蛋白的第一种运动模态是锌指结构域向远离活性中心方向的摆动。如果以反密码子结合的口袋一面为腹面或前面,以对面为背面或后面,以锌指为头部,锌指运动的方向为上方,类似于抬头运动,朝上并略朝向后方的抬头运动。如图4。这个运动方向与结合底物tRNAIle2引起的运动方向是相反的。TiaS蛋白第二种运动模态是锌指朝向Tyr185残基方向的摆动,类似于锌指朝左侧下方的点头运动。自由TiaS蛋白锌指朝左下方侧向点头,与结合底物tRNAIle2引起的运动方向相反。第三种运动模态是绕着连接linker扭动或者称作转动,参照图4(g),扭动方向是向TiaS的左侧。这三个方向上的往复运动已经可以为锌指提供多位置多角度灵活定位的可能性。所以,连接linker反向平行β折叠片的柔性使得锌指具有了多种可选择的构象,来适配结合底物核酸。
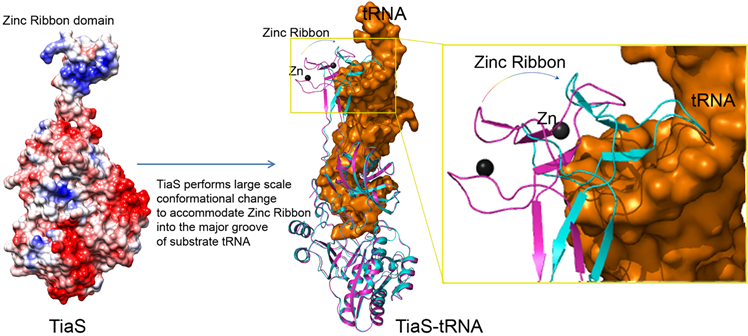
Figure 5. The binding of tRNA induces conformational change of TiaS. Left, the first free full-length 4 domain TiaS protein structure (5XOB.pdb, 2.5Å), large areas of electro-positive region of zinc ribbon domain is responsible for binding to the acceptor stem of substrate tRNAIle2. Right, the proposed models of tRNAIle2-TiaS complex, tRNAIle2 is colored brown, the binding of substrate tRNA probably induces conformational change of TiaS zinc ribbon, and TiaS protein are colored purple and cyan corresponding to configurations before and after conformational change, respectively (Reprinted with permission from ref. 20, Copyright 2018, Elsevier)
图5. 底物tRNA结合引发TiaS构象变化模式图。左图,解析的首个自由全长的4结构域TiaS蛋白结构(5XOB.pdb,2.5Å),锌指结构域有大面积的正电荷,是与底物tRNAIle2接受臂结合的区域。右图,推测的底物tRNAIle2 (棕色)与TiaS蛋白(紫色,无构象变化)结合的结构模型,底物tRNA的结合很可能引发TiaS锌指结构域发生构象变化,模拟的发生构象变化后的TiaS与tRNAIle2结合模式图中TiaS结构标记成了蓝色(摘自ref. 20)
5. TiaS蛋白催化tRNAIle2修饰的酶工作机制
Af-TiaS蛋白在有ATP-Mg存在的条件下,催化ATP水解,生成磷酸化修饰的Cyt34。活化的Cyt34与Agmatine反应生成产物Agm2C [18],TiaS完成修饰反应。这是生化实验的研究结果。依据解析的Af-TiaS蛋白全长的晶体结构(5XOB.pdb)、TiaS-AMPPCP-agmatine结构(6AGG.pdb)、TiaS-tRNAIle2-ATP结构(3AMT.pdb)等,TiaS的催化机制从生物物理学角度也得到解读 [20]。截至目前为止,人们已知的TiaS蛋白修饰tRNAIle2的分子机制,总结在了这两张图中,如图5、图6。从图中可以看出,锌指结构域在酶发挥催化修饰活性过程中发挥着重要的作用。
溶液中,通过自由扩散、静电吸引等过程,TiaS蛋白与底物tRNAIle2前体(precursor tRNAIle2)的距离被拉近,由于酶N端3个结构域与tRNAIle2结合面积与结合能量巨大,很可能首先与tRNAIle2的反密码子区域接触结合,这时候配体ATP、Agmatine可能与二元复合物结合,也可能稍后再与二元复合物结合。由于结合面外形基本互补,通过相互诱导契合等过程,TiaS酶核心与底物tRNAIle2前体的反密码子区域结合逐渐紧密,然后锌指结构域发生巨大的构象变化,移动达14Å,扭转达18º,从而与底物tRNAIle2
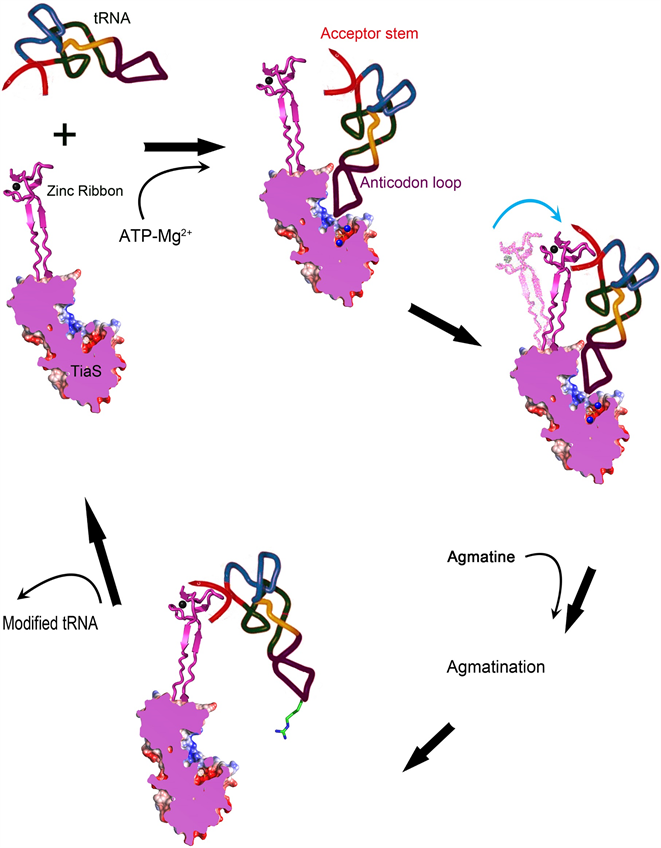
Figure 6. The molecular mechanism of tRNAIle2 modification by TiaS enzyme (Reprinted with permission from ref. 20, Copyright 2018, Elsevier)
图6. TiaS修饰tRNAIle2的分子机制(摘自ref. 20)
的接受臂建立起相互作用。至此,TiaS蛋白与底物tRNAIle2的识别结合过程完成,进入催化修饰过程。在镁离子存在条件下TiaS酶水解ATP,发挥针对Cyt34的Agmatine修饰活性。修饰后的tRNAIle2由于化学结构的改变,改变了反密码子区域的物理化学性质,与TiaS的亲和力低于未修饰的tRNAIle2前体,因此,未修饰的tRNAIle2前体的反密码子区域会首先竞争占据酶N端3个结构域。由于TiaS蛋白与底物tRNAIle2的结合能量与结合面积主要由酶与反密码子区域提供,所占总的结和能量与总的结合面积比例均约2/3,因此tRNAIle2前体占据酶N端3个结构域以后,会把修饰后的成熟tRNAIle2竞争走。每一次催化反应都会进行一轮这样的结合–催化修饰–解离的循环 [20]。
6. 远离催化中心的TiaS锌指结构域与酶的催化功能
TiaS锌指结构域影响酶催化活性的已有研究结果总结如下。去除锌指结构域的TiaS截短体,以及置换掉tRNAIle2接受臂的底物突变体,均不能够完成正常的催化修饰过程 [18]。锌指结构域的Cys352Ala-Cys355Ala双突变体酶催化修饰底物的活性显著下降 [18],这些结果都表明了锌指结构域对于酶修饰功能的重要性。实际上,从解析的结构可以清晰看到,Cys352、Cys355对于维持锌指结构域的三维空间结构非常重要 [20]。实验发现TiaS蛋白催化ATP水解活性可以不依赖于tRNAIle2修饰反应发生。只包含酶N端3个结构域的截短体仍然具有ATP水解酶活性,不具有tRNAIle2修饰活性 [18]。
锌指结构域远离催化活性中心,两者距离超过60Å (5XOB.pdb)。可以推测锌指结构域可能对于识别结合底物过程,在薅住底物放置于正确位置上发挥作用。TiaS蛋白使用酶催化核心和锌指结构域识别结合底物tRNAIle2。从静态的三维结构上看,锌指结构域结合底物tRNAIle2的接受臂。晶体结构(3AMT.pdb)中可以观察到TiaS蛋白的Glu360侧链羧基与tRNAIle2的Cyt70的N4原子存在氢键相互作用。
7. TiaS锌指与核酸相互作用的研究
锌指是典型的蛋白质结构单元之一,可以介导蛋白与蛋白,蛋白与核酸的相互作用。含有锌指结构的蛋白质参与了多种重要的生命过程,是全球酶学、生物化学等领域的典型研究对象 [24] [25] [26]。CRISPR/Cas系统和锌指都是靶向核酸的重要工具,靶向核酸酶在基因矫正、基因组编辑、基因治疗等新兴生命医药以及研究领域具有巨大的应用前景。核酸靶向蛋白的改造是其中的热点之一。设计改造锌指结构单元一直是核酸靶向蛋白组件研究的前沿阵地 [27]。与CRISPR/Cas系统相比,使用锌指作为核酸序列特异性结合元件的挑战之一,是锌指组件数目的增加并不一定达到预期的亲和力增加,因为存在空间位阻 [27]。也由于存在这样的难点,使用锌指靶向核酸的应用受到限制,将其成功利用仍需大量研究。
TiaS锌指以柔性来适配与核酸的结合。对于TiaS蛋白的研究发现,锌指与N端3个结构域的连接部位细长,可以通过伸缩扭动使锌指结构域空间位置与取向发生变化,具有较大的柔性和多种可选择的构象(4),来适配底物核酸。这是TiaS蛋白新型锌指的第一个优点。
TiaS锌指以序列特异性识别核酸。对于核酸靶向锌指的改造来说,实现靶向性的一个关键要求是,对于核酸碱基的识别是序列特异的。研究发现,TiaS锌指的若干氨基酸残基通过碱基特异性方式识别底物tRNAIle2。TiaS锌指的Glu360,Ser361,Ala362,Gln366,Arg369氨基酸残基识别底物tRNAIle2的一段特定碱基序列Cyt70Cyt71Cyt72Ade73 (其中Cyt71-Gua2,Cyt72-Gua1部分识别的是双链碱基),6对氢键是与4个连续的碱基一一形成的,不是与糖磷酸骨架形成的。晶体结构3AMT.pdb中,也观察到了Glu360、Ser361、Arg369与碱基的氢键,是相互吻合的。这是TiaS蛋白新型锌指的第二个优点。目前发现的锌指的这些开创性的新特征,有可能会拓展锌指作为靶向核酸工具的科学前沿。进一步探索TiaS蛋白识别结合底物tRNAIle2过程中锌指结构域的作用,深入发掘以前未知的锌指及其连接部位的新功能特性,将为锌指的工程改造提供依据。
目前,利用TiaS特异性识别结合核酸的特性,科研工作者进行了应用研究的开发。让TiaS使用具有点击反应活性的化合物对RNA进行靶向定点修饰,利用点击反应在体外和哺乳动物细胞中实现了目标RNA的特异性共价荧光标记 [19],这是一种进行RNA标记的新方法,而目前科学领域诸多重要的RNA研究依赖于优越的新型标记方法 [28]。
8. 结论
目前,对于TiaS蛋白的工作机制已经有了较多的实验证据和较为充分的理解,TiaS蛋白的新型锌指的新特性值得进一步研究,针对TiaS蛋白锌指关键氨基酸残基与核酸相互作用的深入研究,将为靶向核酸锌指的改造设计以及应用研究打下基础,也将拓宽加深人们对于酶催化机制的理解。近年来,锌指的一些新特性被发掘出来,包括通过人工设计与实验室进化获得的以锌指为核心的具有催化活性的小分子量的生物酶,该酶可以特异性水解酯键 [29]。另使用沙利多安及其类似物(Thalidomide,lenalidomide,pomalidomide)联合其它分子来靶向结合包含锌指的蛋白(很多是细胞内的转录因子),从而对一些以前认为是不可成药的蛋白质靶点通过其锌指进行了特异性的靶向降解 [30]。这些研究进展,使得锌指的应用范围被拓展的更加广阔。
基金项目
本研究得到国家自然科学基金青年项目资助,项目编号31900913;以及科技部重大专项2016YFA 0501800。
缩略词
Cyt34, cytosine 34; TiaS, tRNAIle2 agmatidine synthetase; ZRD, zinc ribbon domain; PCA, principal component analysis; NMA, normal mode analysis; tRNA, transfer RNA; RNA, ribonucleic acid; TilS, tRNAIle2 lysidine synthetase; Ade, adenine; Ura, uracil; Cyt, cytosine; Gua, guanine; MetRS, Methionyl-tRNA synthetase; TCKD, Thr18-Cyt34 kinase domain; FLD, ferredoxin-like fold domain; OBD, oligonucleotide binding domain.
附录