1. 引言
阿尔茨海默病(Alzheimer’s Disease, AD),俗称老年痴呆,其临床症状表现记忆力衰退,视听觉功能损害,逐渐变得生活不能自理,重度阶段患者出现昏迷且伴有多种并发症。美国国家卫生研究院(National Institutes of Health) [1] 将阿尔茨海默症评估为第三大老年人致死疾病,仅次于癌症和心脏病。据阿尔茨海默协会(Alzheimer Association) [2] 统计,2020年全球约有5500万人罹患阿尔茨海默症,预计到2030年患病人数将达到8200万,而2050年预计将攀升至1亿5200万人。阿尔茨海默症的广泛发生给患者家庭和政府带来了沉重的经济负担。然而,阿尔茨海默症的病理机制仍未完全揭示,其主要原因在于人脑是一个由脑区和神经元构成的高度复杂的系统,认知的完成需要多个脑区之间协调配合(即神经环路) [3]。另一方面,临床脑科学研究也受到人类伦理的限制。这些原因造成了阿尔茨海默症病理机制的研究进展缓慢。
过去十年中,随着计算机和人工智能的发展,机器学习、网络分析 [4] [5] [6] [7] [8] 等技术在许多领域获得了广泛的应用。医学图像分析逐渐走向智能化 [9] - [14],越来越多的机器学习方法开始介入阿尔茨海默症的研究 [15] [16] [17],基于图像形态学的机器学习方法 [18] [19] [20] [21] [22] 已经在阿尔茨海默症智能辅助诊疗相关的一些基本任务中取得了巨大成功,例如阿尔茨海默症早期检测。但是这些方法只关注了脑区是否萎缩等形态学变化,无法刻画脑区之间的相互作用关系,因此仍然难以帮助人们彻底理解阿尔茨海默症的发病机制。为了脑区之间的连接关系表征阿尔茨海默症相关特征,脑网络方法应运而生。
脑网络 [23] [24] 将脑区以及脑区之间的连接关系映射到一个图数据。图的节点代表大脑的各个脑区,图的边代表脑区之间的连接。脑网络可以分为两种基本类型:功能连接和结构连接。功能连接被定义为两个脑区的血氧水平依赖性信号之间的关联性强弱,其中血氧水平依赖性信号通常可以从功能磁共振(fMRI)中提取。结构连接被定义为两个脑区之间的神经纤维连接强度,通常可从弥散张量成像(DTI)中提取。研究表明 [25] 使用功能连接或结构连接能够获得一些传统图像学方法无法发现的阿尔茨海默症相关特征。这证明了脑网络方法在阿尔茨海默症的研究中更具优势。然而,现有的大多数脑网络研究都是基于单模态的,只关注结构连接和功能连接中的一种。当前研究者们开始注意到单模态数据可能只包含部分的疾病相关信息,这意味着仅用使用单模态数据会失去利用跨模态互补信息来更加准确且全方位地对阿尔茨海默症进行研究的机会。因此,多模态大脑网络计算在医学图像智能计算领域已受到越来越多的关注。多模态脑网络的结构信息–功能信息深度融合的关键在于如何有效利用跨模态互补信息 [26]。现有大多数的多模态脑网络计算方法利用加权求和、仿射变换、特征拼接等方式进行特征融合。然而,由于fMRI和DTI数据之间的强异质性,二者的特征向量最初位于不同子空间中且关联特征是非线性的 [27],因此现有大多数多模态脑网络计算方法并不能充分利用fMRI和DTI数据之间的互补信息。
为了解决上述问题,同时更加有效地利用脑网络中的高阶拓扑信息,已有研究者开始利用超图来表征脑网络 [28]。超图是图数据的推广,超图和图数据的最大区别在于超图中的每条超边可以包含多个节点,以此刻画高阶拓扑特征。超图的这一特点使得其在神经环路的高阶互补特征融合上具有其他数据形式所无法比拟的优势。另一方面,生成对抗策略 [29] - [35] 已成功用于医学图像分析的许多分支。生成对抗策略可以在无监督的情形下学习到未知复杂分布,基于这一特点,使得我们的融合算法中不需要加入额外的先验假设,从而更灵活高效的进行异质数据深度融合。在本项工作中,我们提出了交替式超边神经元算法来对fMRI和DTI进行深度特征融合,并且设计了最优超图同态算法增强超图建模的稳定性和鲁棒性。在生成对抗策略方面,我们设计了基于图随机游走的判别器来约束模型学习得到的多模态脑网络分布。与传统的多模态融合算法相比,我们提出的基于超图理论和生成对抗策略的fMRI、DTI融合算法的优势在于能够更加准确地利用疾病相关的互补信息以及脑网络高阶拓扑特征,为阿尔茨海默症相关生物标志物的精准识别奠定基础。
2. 相关工作
2.1. 超图算法
利用超图对脑网络进行研究,针对单模态脑网络的超图相关算法主要集中在超边表征学习的高阶疾病生物标志物检测以及功能连接计算方面。高跃等人 [36] 采用超边表征学习对自闭症患者的功能脑网络进行研究,得到了关于自闭症相关的生物标志物,并证明该结果和自闭症临床研究结果高度一致。刘明霞等人 [37] 采用超图对齐学习对阿尔茨海默症的早期诊断进行了研究,实验结果证明基于超图的方法相比于其他方法在分类性能上有明显优势。针对多模态数据以及多模态脑网络融合问题,邵伟 [38] 等人采用超图建模对多模态数据的阿尔茨海默症多标签信息进行学习,模型性能相比于传统多模态融合算法有所提升。Aviles-Rivero [39] 等人利用扩散学习研究了多模态数据的超图融合。
2.2. 生成对抗策略
生成对抗策略可以看作是一种基于变分推断 [40] [41] [42] [43] 的对抗式学习方法。生成对抗网络(Generative Adversarial Networks, GAN)是近年发展起来并引起广泛关注的深度学习模型,因其无需先验假设就能够高效学习复杂未知分布的能力,使得生成对抗网络已经成医学图像领域的研究热点之一。陈卓 [44] 等人利用一致感知型生成对抗网络实现了脑卒中病变区域的自动分割。胡圣烨等人 [45] 基于生成对抗网络实现了MRI数据到PET数据的跨模态生成,解决了阿尔茨海默症研究中PET数据难以获得的问题。冀俊忠等人 [46] 利用基于循环神经网络的生成对抗策略实现了阿尔茨海默症脑网络有效连接的精准计算。
3. 数据与方法
3.1. 数据
本项工作中使用的数据集是Alzheimer’s Disease Neuroimaging Initiative (ADNI),我们从该数据集中挑选了健康对照组(NC),轻度认知障碍(EMCI),重度认知障碍(LMCI),阿尔茨海默症(AD)四种疾病类别共计268个被试的数据,其中每个被试都包含fMRI数据和DTI数据,其数据描述和分布如表1所示:
对于fMRI数据,采用解剖自动标记模板(Anatomical Automatic Labeling, AAL90)和DPARSF工具包 [47] 进行预处理,处理步骤包括丢弃前20个时间序列片段、头部运动校正、手动位置校正、带通滤波和高斯平滑,最终提取得到90个脑区长度为180的血氧水平依赖性信号。采用GRETNA工具包 [48] 从血氧水平依赖性信号中计算得到脑网络功能连接。对于DTI数据,采用解剖自动标记模板和PANDA工具包 [49] 进行预处理,处理步骤包括脑掩模估计、涡流效应校正、脑区之间的白质纤维束追踪估计。
3.2. 方法
所提出的模型属于生成对抗学习,包含一个基于交替式超边神经元算法的生成器,以及一个基于图随机游走的判别器,模型结构总图如图1所示:
3.2.1. 超图
超图(Hypergraph)是一种新的数据结构,由于组合数学和计算机科学的发展,超图在数据科学中的应用也越来越丰富。超图主要包括了两个部分,即节点和超边。我们将超图记作
,其中V表示节点构成的集合,E表示超边构成的集合,其中每条超边是一组具有某种关联关系的节点所构成的子集,令
分别表示节点的个数以及超边的条数。记节点为
,超边为
。超图H所对应的关联矩阵
定义为:
超图数据的示意图如图2所示。

Figure 1. Network structure of proposed module
图1. 模型网络结构
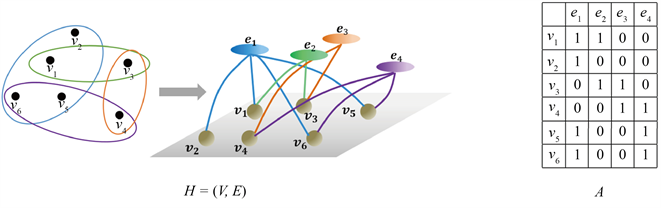
Figure 2. Left: A hypergraph H with
and
, where
,
,
, and
. Right: The incidence matrix A of the hypergraph H
图2. 左边:节点集合为
,超边集合为
的超图H,其中
,
,
,
。右边:超图H所对应的关联矩阵A
3.2.2. 最优超图同态算法
在本项工作中,我们将每个脑区作为一个节点,使用Jiangwen Jiang等人 [50] 提出的动态超图构造算法(Dynamic Hypergraph Construction Algorithm),以从fMRI中提取得到的血氧水平依赖性信号为节点特征,计算得到每个被试的初始超图数据
。然而,每个初始超图
侧重于刻画对应被试的同频激活脑区,因此当我们直接将初始超图
送入模型学习会导致模型训练不稳定、鲁棒性差等问题。为了克服这一缺陷,我们针对性的设计了最优超图同态算法,用以提取超图数据集
中的公共拓扑分布,以此增强模型的鲁棒性。最优超图同态算法基于超图相似度指标,我们首先给出超图相似度指标的概念。对于两个具有相同节点的超图
和
且条件超边条数相等,即
,H与
之间的相似度定义如下:
这里f是超边集合E到
的双射,
的定义如下:
其中
对于超图集合
,基于超图相似度,最优超图同态算法定义如下:
通过最优超图同态算法得到与
具有最高公共拓扑分布的超图H后,我们采用对
和H进行拼接的方式完成超图构建,得到超图
,即
。我们记第k个被试的超图数据
的关联矩阵为
。
3.2.3. 交替式超边神经元算法与生成器
利用解剖自动标记模板(Anatomical Automatic Labeling, AAL)将大脑划分为90个脑区。从fMRI数据中提取每个脑区的血氧水平依赖性信号,长度为180。这样我们就得到了维数为
的节点初始特征,记作
,这里下标k代表被试编号。同时,从DTI数据中提取各个脑区之间的白质纤维连接强度,得到维数为
的结构连接矩阵
,结合由最优超图同态算法得到的被试超图关联矩阵
(m为超边的条数),得到超边初始特征:
模型生成器G由L层交替式超边神经元模块构成,每个交替式超边神经元模块对上一层的节点特征和超边特征进行交替式更新,第
层节点及超边特征更新具体算法如下:
(1)
(2)
这里
和
分别表示第l层节点及超边特征,
和
是第
层交替式超边神经元模块中可学习的权重矩阵,
是预先设置的超参数,
是激活函数。交替式超边神经元算法深度融合了结点和超边的特征,由于节点初始特征由fMRI所提取功能信息得到,超边初始特征由DTI所提供的结构信息得到,结合超边刻画高阶拓扑信息的优势,因此经过L层交替式超边神经元模块,实现了功能–结构互补信息在高阶拓扑上的深度融合。最后,利用超图关联矩阵
与第L层超边特征
,计算得到多模态脑网络连接矩阵:
(3)
3.2.4. 生成对抗策略与随机游走判别器
模型判别器基于随机游走对模型生成的多模态脑网络进行功能分布与结构分布的双向约束。对于任意给定节点
,我们将其作为随机游走的起始点。随机游走的终止条件是在随机游走的某一步,点回到上一步的位置。我们以步长为
的一次随机游走来进行说明,假设此次随机游走所经过的路径为
,那么,根据随机游走的终止条件,上述游走路径满足
。由于随机游走是一个马尔可夫过程,因此经历这一条游走的路径的概率为:义如下:
我们分别以多模态脑连接矩阵
,由预处理软件从fMRI和DTI中得到的功能连接矩阵和结构连接矩阵
和
作为随机游走中的概率转移矩阵,并结合游走终止条件,带入上式概率公式得:
记
是所有以
为起点,v为终点的路径所构成的集合。那么,在多模态脑连接、功能连接、结构连接下的随机游走分布分别为:
(4)
(5)
(6)
判别器D的结构由多层感知机构成,最后通过一个全连接层输出节点v是以功能连接或结构连接为概率转移矩阵进行随机游走得到的,还是由多模态脑连接作为概率转移矩阵得到的。根据生成对抗策略,判别器的损失函数为:
生成器损失函数为
这里K表示被试的个数。
4. 实验与结果
4.1. 实验设置
本实验的环境设置如表2所示。对于生成器,它由8层交替式超边神经元模块(算法见公式(1)和公式(2))构成,得到最终更新后的超边特征后利用公式(3)计算得到生成器输出的多模态脑网络连接矩阵。对于判别器,根据公式(4) (5) (6)分别以多模态脑网络连接、功能连接、结构连接为概率转移矩阵进行随机游走采样,得到相对应的分布,然后通过多层感知机提取特征,最终通过全连接层输出该节点是否为游走终点的概率。我们使用Adam优化器对模型中参数进行更新,其中学习率、动量衰减因子、无穷大范数衰减因子分别设置为0.0001与0.9、0.999。训练轮次设置为500轮,训练过程中每次送入模型的批次数量设置为16。

Table 2. Experimental environment parameter configuration
表2. 实验环境参数配置
4.2. 实验结果分析
训练完成后,我们按照四个不同的疾病阶段,即健康对照组(NC)、轻度认知障碍(EMCI)、重度认知障碍(LMCI)、阿尔茨海默症(AD),计算每个组别的多模态脑网络连接的平均值。首先对各个脑区之间的多模态连接进行p检验,以检查其是否符合高斯分布。如果其p值小于0.05,则判定该脑连接不符合高斯分布。我们将舍弃那些不符合高斯分布的脑连接,将其连接强度设置为0。对于符合高斯分布的脑连接,我们取其连接强度的平均值。不同疾病状态下的平均多模态脑网络连接矩阵如图3所示:
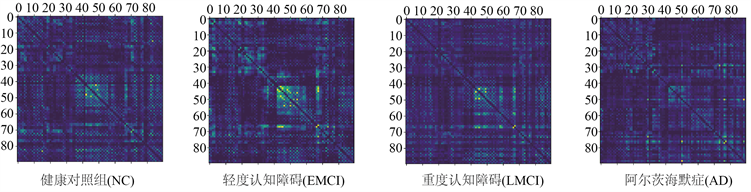
Figure 3. The multimodal brain network with respect to four groups of different disease stages
图3. 不同疾病状态下的多模态脑网络连接
由于在疾病发展过程中增加或者减少的脑连接是阿尔茨海默症重要的疾病标志物,在得到各个组别的多模态脑网络连接后,基于此我们对疾病变化过程中的脑连接变化情况进行了研究,结果如图4所示。
根据多模态脑网络在不同疾病状态下的连接变化情况,我们挑选出了8个连接变化最多的脑区:37-左侧海马体(Hippocampus_L),38-右侧海马体(Hippocampus_R),42-右侧杏仁核(Amygdala_R),56-右侧梭状回(Fusiform_R),59-左侧顶上回(Parietal_Sup_L),68-右侧楔前叶(Precuneus_R),73-左侧豆状壳核(Putamen_L),89-左侧颞下回(Temporal_Inf_L)。这里脑区名称前的数字为对应的解剖自动标记编号(ALL id)。我们分别对健康人和阿尔茨海默症患者在上述8个重点脑区内部的多模态连接进行了可视化,结果如图5所示。
结果显示,健康人和阿尔茨海默症患者在重点脑区内部的多模态连接具有明显的不同,因此说明我们模型计算得到的多模态脑网络能够有效刻画阿尔茨海默症疾病特征。
4.3. 对比实验
为了更好地验证模型表征阿尔茨海默症疾病相关特征的能力,我们在三个不同的分类器:支持向量

Figure 4. The changes of multimodal brain network connectivity on the different development processes of AD
图4. 不同阈值下,阿尔茨海默症发展过程中的多模态脑网络连接变化

Figure 5. Multimodal connectivity in crucial ROIs
图5. 重点脑区内的多模态连接
机(Support Vector Machine, SVM)、图卷积(Graph Convolutional Network, GCN)、深度神经网络(Deep Neural Networks, DNN)下设计了三组分类实验:健康对照组与轻度认知障碍、健康对照组与重度认知障碍、健康对照组与阿尔茨海默症,将我们所提出的多模态脑网络计算方法和其他多模态脑网络计算方法进行比较。参与比较的多模态脑网络计算方法包括:深度图卷积融合算法 [51]、多核学习算法 [52]、拓扑融合算法 [53]。实验结果的评价指标包括:精确度(Accuracy, ACC)、灵敏度(Sensitivity, SEN)、特异性(Specificity, SPE)、F1值。上述评价指标计算方法如下:
这里TP表示真阳性、FP表示假阳性、TN表示真阴性、FN是假阴性。结果如图6所示。

Figure 6. Under different classifiers, the prediction performance of multimodal brain network computed by different multimodal fusion methods
图6. 在不同分类器下,不同的多模态融合方法计算得到的多模态脑网络的预测性能
5. 总结
阿尔茨海默症病理机制复杂,传统的单模态形态学方法无法对其进行精准刻画,因此需要利用多模态脑网络方法进行研究。本文提出了基于交替式超边神经元算法的生成对抗策略可以有效地预测多模态脑网络的计算,为阿尔茨海默症的智能辅助诊疗和疾病标志物分析奠定基础。本文主要内容包括:
第一,提出了最优同态超图算法,改进了原有的超图建模方法,有效提高模型的鲁棒性和泛化能力。第二,提出了交替式超边神经元算法,对脑区活动特征和超边高阶拓扑特征进行交替式迭代更新,高效地利用了不同模态数据之间的高阶互补信息。第三,采取图随机游走的方式比较模型生成的多模态脑网络和软件模板计算得到的单模态功能连接、结构连接之间的分布差异,对多模态脑网络在功能分布与结构分布上进行双向约束,实现了异质–异构数据的高效融合。综合实验证明,本文所提出的多模态脑网络融合算法在阿尔茨海默症相关疾病检测分类任务中,精度明显高于其他参与比较的多模态脑网络融合算法。证明了我们所提出的融合算法能更为有效地提取疾病相关特征。
此外,本文所提出的多模态脑网络融合方法,可以无障碍地推广到其他神经退行性疾病的研究上,例如帕金森症、癫痫等。另一方面,本文也存在一些不足之处,如多模态标注数据不足、参与比较的其他融合方法有限等问题,为此,在未来的实验与研究中,计划采用更大规模的数据集进行实验,与更多的相关方法进行比较,与阿尔茨海默症临床研究方面的专家展开合作。
基金项目
本文工作受以下项目资助:深圳市自然科学基金重点项目(JCYJ20180507182506416)。
NOTES
*通讯作者。