摘要: 现如今医学领域和计算机领域的融合程度越来越深,越来越多的研究团队使用计算机先进的图像处理技术来完成医学影像的分类,分割,检测,配准和成像重建等工作,但是这仍是少数,绝大多数的医疗机构仍然是依靠医生来根据图像进行诊断和结果分析,这大大降低了医院的工作效率,诊断时间长,给患者无论是精神上还是身体上都带来了负担。因此,我们通过机器深度学习的预训练3D模型,3D建模和卷积神经网络技术来建立一个可以直接进行影像分类,切割和病灶检测的系统。本研究针对CT层面中的2D病灶检测问题提出了一种可以有效利用3D上下文信息的新框架,同时提出了一种预训练3D卷积神经网络的新思路,该研究在迄今规模最大的CT图像数据集NIH DeepLesion上进行了实验,并取得了SOTA的病灶检测结果。有监督预训练方法可以有效提升3D模型训练的收敛速度,以及在小规模数据集上的模型精度,用于提升病症分析的速度和准确率,提高医疗效率。论文的主要研究成果包括:1) 提出了3D卷积模型病灶检测预处理方法。该方法是通过数据的预处理将CT影像转换为三通道的伪彩色图像,从而将图像中的像素值归一化到相同的范围,减小了不同类型病灶形态差异。其次,通过预处理,结合多类型病灶框大小的先验知识,对锚框宽高比进行了优化。2) 开发了一个通用和高效的能够增强3D上下文信息建模的网络框架。首先提出一种改进的伪3D框架来对连续多层输入进行高效的3D上下文特征提取,同时配合一个组卷积变换模块,在该特征输入到检测头之前可以将3D特征转换为2D特征,来适配我们的2D目标检测任务。以确保模型始终具备3D上下文建模能力。3) 研究设计了一种有监督的预训练方法来增强MP3D的训练以及收敛性能。本研究工作提出一种基于变维度转换的3D模型预训练方法:将2D空间中的channel维度转换为3D空间中的dept维度,将原始具有色彩信息的RGB三通道二维图像转化成三维空间中的三个连续层面图像,以此达到有效的利用2D自然图像处理进行3D模型的预训练。本论文在DeepLesion数据集上对提出的方法进行了定性和定量的分析对比。结果显示本论文方法可以作为CT影像中多类型病灶的辅助检测方法,从而促进CT技术在临床的应用。
Abstract:
Nowadays, the integration between the medical field and the computer field is getting deeper and deeper. More and more research teams are using advanced computer image processing technology to complete the classification, segmentation, detection, registration and imaging reconstruction of medical images. However, this is still a minority, and the vast majority of medical institutions still rely on doctors for diagnosis and result analysis based on images. This greatly reduces the working efficiency of hospitals, takes a long time to diagnose, and puts a burden on patients both mentally and physically. Therefore, we use the pretrained 3D model of machine deep learning, 3D modeling and convolutional neural network technology to build a system that can directly perform image classification, cutting and lesion detection. This study proposed a new framework for 2D lesion detection at the CT level that can effectively utilize 3D contextual information, and proposed a new idea of pre-training 3D convolutional neural network. This study conducted experiments on NIH DeepLesion, the largest CT image dataset to date, and obtained SOTA lesion detection results. The supervised pre-training method can effectively improve the convergence speed of 3D model training, as well as the model accuracy on small-scale data sets, so as to improve the speed and accuracy of disease analysis and medical efficiency. The main research achievements of this paper include: 1) A pretreatment method for lesion detection with 3D convolution model is proposed. This method converts CT images into three-channel false-color images through data preprocessing, so that the pixel values in the images are normalized to the same range, and the morphological differences between different types of lesions are reduced. Secondly, the aspect ratio of the anchor frame was optimized by preconditioning combined with the prior knowledge of the size of the multi-type focus frame. 2) Develop a general and efficient network framework that can enhance 3D contextual information modeling. Firstly, an improved pseudo-3D framework is proposed to extract 3D context features efficiently for the continuous multi-layer input. At the same time, a group convolution transform module is combined to convert 3D features into 2D features before the feature is input to the detection head, so as to adapt to our 2D target detection task. To ensure that the model always has 3D context modeling capability. 3) A supervised pretraining method was designed to enhance the MP3D training and convergence performance. This research proposes a 3D model pretraining method based on variable dimension transformation: The channel dimension in 2D space is converted to the dept dimension in 3D space, and the original RGB three-channel two-dimensional image with color information is converted into three continuous level images in 3D space, so as to effectively use 2D natural image processing to conduct 3D model pretraining. In this paper, we compare the proposed methods qualitatively and quantitatively on DeepLesion data sets. The results show that the method in this paper can be used as an auxiliary detection method for multiple types of lesions in CT images, so as to promote the clinical application of CT technology.
1. 引言
1.1. 医学影像分析背景
如今,现代的医学很大程度上被高速发展的计算机推动了 [1],应用图像存档和传输系统(也称PACS),也很大程度上为存储图像信息和运输图像信息提供了新方法、新模式。而医学影像的进一步融合,作为图像预后处理技术,必定会成为一个影像学研究领域新潮的热点,同样的,它也必定将是医学影像学一个崭新的发展道路。我们所说的影像的融合,其实融合的是影像信息。在医学范围内应用的其实是信息融合技术,同时也是计算机技术的应用,数字化研究各种各样的影像学检查获得的图片,把多源数据协同到一起应用,在配准了空间以后,会自然产生一种崭新的影像,与此同时,还利用全方位多角度筛检的特点,从而让计算机成功辅助了医学诊治。
1.2. 计算机与医学影像融合刻不容缓
首先,技术更新才是需要影响融合的对象 [2]。计算机技术在医学影像学中的使用越来越多,正如前文提到过的图像存档和PACS,证明了一种更完美的技术方法在图像信息保存运输等方面正式出现了 [3]。其次,某一个单一检查成像的缺点也因为影响融合的出现彻底消失了。当前,像传统老旧的X线、B超,亦或是CR、CT、MRI、SPECT等,可以说是百花齐放,但其实他们在成像中有分别拥有着许多缺点,都有一些局限的地方。举例子来说:CT检查,他有着很高的分辨率,但是他在某些事情上还是存在困难,比如分辨某些密度十分相近的组织,甚至在颅后窝等检查中,他很容易产生骨性伪影,大大地降低了检查的准确以及精密性。再者,临床最需要的影像诊断也是影像融合技术,他最根本上为临床的医治做了很大贡献。我们相信,全新的影像在融合了多种影像后,会让医疗诊治精密全面许多。
1.3. 计算机与医学影像融合可行性
影像学的多个检查存在的相同性和不同性都为影响融合技术奠定了深厚的基础。虽然我们都知道每个检查都应用了不一样的检查方法、成像的原理和特点,但是尽管如此,它们都存在共同相似的形态学基础,它们都一样是通过影像来展现组织形态、以及病例解剖、代谢变化等等,医学影像融合技术应用了多种技术一致性。
2. 研究模型介绍
2.1. 搭建模型思路
由于目前计算机进行深度学习通常都需要依赖于大量的已标注数据,对于医疗图像的样本标注又会涉及到大量的专业医学知识,标注人员需要对病灶的尺寸大小、整体形状和边缘等信息进行准确的判断分析,甚至需要资历较深的专家团队来进行两次及以上的评判,这便推进了机器深度学习在医疗领域中的应用 [4] [5]。由此,基于3D医学影像中病灶检测的研究,该研究针对CT层面中的2D病灶检测问题提出了一种可以有效利用3D上下文信息的新框架,提出了一种预训练3D卷积神经网络的新思路。该研究在迄今规模最大的CT图像数据集NIH DeepLesion上进行了实验,取得了SOTA的病灶检测结果。当Sensitivity@0.5FPs,相比当前SOTA方法提升3.48%,而相比2.5D的baseline方法,提升高达4.93% [6]。该实验表明,上文所提出的有监督预训练方法可以较为有效提高3D模型训练的收敛速度,并且在小规模数据集上的模型精度也同样有明显的提升。研究团队在四个基准的3D医学数据集上进行了大规模的实验验证,其结果表明对我们的预训练3D模型进行调优(finetune)不仅可以显著优于从头训练(training from scratch)的3D模型,而且与目前现有的最先进、最尖端的自监督和全监督预训练模型相比,该实验团队模型效果在多数任务上都具有显著的优势。
2.2. 开发技术和算法
2.2.1. CT关键层面中病灶检测中的3D上下文建模
对于自然图像处理,普通的机器深度学习技术都是基于2D CNN解决相关问题。然而 [7],对于医学影像而言,尤其是3D医学影像(如CT,MRI等),不同层面不同精度的影像本质上是提供同一个病灶点或者器官的不同切面图像,这些不同层面精度上的信息具有高度的互补性和相关性。如果只是在单一、单个的层面上进行图像检测,那么该图像中所包含的极大一部分信息没有达到有效利用,这样不仅会造成信息资源的浪费,而且会导致诊断结果出现相对的偏差。因此基于CT影像的关键层面中的2D病灶检测的问题 [8] [9],一个相对比较直接的解决方案便是将得到的连续的三层CT影像拼接成一个三通道的二维图像,对连续多层CT图像使用3D卷积进行特征提取,这样能更好地提取连续层面之间的结构和纹理特征并将该连接图像输入到2D网络中进行病灶检测。
同时,为了解决3D卷积计算量大和训练收敛速度慢的问题,提出了针对性的模型结构改进和3D预训练方法。该方法可以较为有效的利用2D自然图像处理中的预训练模型提升其特征表达能力,但是仍存在技术上尚未攻克的难题。当前很多研究也意识到3D上下文建模的重要性,比如MVP-Net等工作采用2.5D的方式来提升3D上下文建模能力,他们通过构造多通道的2D网络来融合更多连续层面(9层或者27层)的2D特征,实验结果表明这种方法对比单纯的2D方法相对大的性能提升。
2.2.2. 3D上下文信息增强网络(MP3D)
针对CT图像关键层面中的病灶检测问题,开发了一个通用和高效的能够增强3D上下文信息建模的网络框架 [10]。首先提出一种改进的伪3D框架来对连续多层输入进行高效的3D上下文特征提取,同时配合一个组卷积变换模块,在该特征输入到检测头之前可以将3D特征转换为2D特征,来适配我们的2D目标检测任务,见图1。为了提高普通三维ResNet的计算和参数存储效率,我们采用伪3d残差网络(P3D ResNet)作为我们的雏形骨干网络。伪3d卷积模拟的是3 × 3 × 3卷积在轴方向视图切片上使用1 × 3 × 3卷积核并加上3 × 1 × 1卷积核来构建相邻CT上的上下文信息(如图1所示)。除此之外,在关键层面病灶检测设定上 [11],我们通常输入的图像层数(文中n = 9)远小于轴方位上的图像尺寸(通常是512 * 512)。在整个特征提取的过程中,只对X轴和Y轴方向进行降采样,保持Z轴方向的尺寸不变,以此来确保模型始终具备3D上下文建模的能力,见图2。
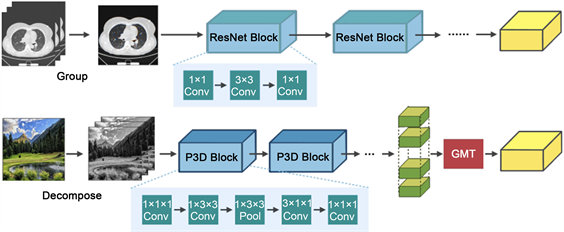
Figure 2. 1) Comparison of multiple sections using 2D Image-Net pre-training weights; 2) Hierarchical simulation of 3D network pre-training of 2D natural images
图2. 1) 利用2D Image-Net预训练权值进行多切片的比较;2) 对二维自然图像进行分层模拟医学图像三维网络预训练
2.2.3. 基于变维度转换的3D模型预训练
研究中发现在NIH DeepLesion这种拥有较大数据量的数据集中,在使用3D backbone进行建模时,虽然使用从头训练的方式可以达到较为可观的模型效果,但其收敛所花费的时间达到Finetune模式的3倍以上。而在数据量较小的情况下,即使训练轮次足够长,但其收敛的效果也始终难以和Finetune模式相衡。因此,研究设计了一种有监督的预训练方法来增强MP3D的训练以及收敛性能(图3) [12]。该研究工作时提出一种基于变维度转换的3D模型预训练方法:将2D空间中的channel维度转换为3D空间中的dept维度,将原始具有色彩信息的RGB三通道二维图像转化成三维空间中的三个连续层面图像。
通过这种变维度转换方法,将二维空间的丰富的色彩信息以3D结构信息的形式被保存下来。基于这些伪3D数据学习的3D卷积核由此便具备了可以分析表达存在于3D医学影像中的复杂3D结构和纹理信息的能力。使用变维度转换模块所得到的伪3D图像进行3D模型预训练,和我们上文中所提到的病灶检测模型相类似,需要注意的时不要在Z轴方向进行降采样操作,要始终确保Z轴方向保持在depth=3,从而可以不断的学习3D上下文信息。预训练的代理任务,根据其使用的数据集不同可以是基于ImageNet的分类任务、基于COCO的检测任务或者时基于分割数据集的分割任务等等。
最后,通过该变维度转换方法学习到的3D网络参数可以用于下游医学任务的微调及其优化,其迁移学习能力远超于在下游3D医学数据上的从头训练的实验效果。此外,因为是基于现有2D数据集来进行的3D模型预训练,和其他预训练方法相比较,此方法可以有效避免大规模医疗数据的采集和更新数据标注。同时,我们了解到,在本文之前,将连续层面的3D医学图像合并成RGB图像,并应用在自然图像处理领域预训练完好的2D模型进行特征表达已经成为处理医学影像的一个标准化流程。本文所提到的变维度转换可以理解为上述过程的一个逆向变换。基于该逆向变换,我们则可以有效的利用2D自然图像处理进行3D模型的预训练,见图3。
2.3. 面临的问题和解决方法
截至目前,虽然有一些公开可用的数据集(例如LIDC-IDRI、LUNA等),但是这些公开数据集仍然存在问题不够完美,图像数量较少,偏倚度较高的问题,这些都会导致模型不可避免地出现过拟合。为了攻克这一问题,通常会借助迁移学习使用ImageNet等大规模数据集上的预训练参数加快模型的收敛;然而,对于3D医学图像(例如CT、MRI等),目前仍然还没有很好的3D模型预训练参数 [13]。同时,对于连续多层CT图像使用3D卷积进行特征提取的方法仍然缺乏对多层面之间相关信息的处理和解释能力,因此也很难对3D上下文建模,导致其效果有限。

Figure 3. 3D model pre-training based on variable dimension transformation
图3. 基于变维度转换的3D模型预训练
3. 基于四个训练集下全监督3D模型较传统方法的优势与结果说明
注释在很大程度上限制了用于3D医学成像应用的训练数据集的样本量。因此,从零开始构建高性能的三维卷积神经网络仍然是当今世界的一项艰巨任务。以前在3D预训练方面的自监督方法,无法获得大规模监管信息,这种学习框架仍然存在问题。本项目重新审视一个创新而简单的全监督3D网络,在四个基准数据集上的实验证明了这一点,提出的预训练模型可以有效加速收敛的同时也提高了各种精度三维医学影像的分类、分割、检测等任务。此外,与培训相比从头开始,它可以节省高达60%的注释工作。在NIH的DeepLesion数据集中,它同样实现了最先进的检测性能,优于早期的自我监督和完全监督的训练前方法。
首先我们对自然图像数据集进行预训练,见图4,预训练的数据集是数据集ImageNet提供的分类数据集,这些图片是从互联网上收集的,并经过人工处理的,它由118千张自然图像和总共90万个边界框80个类别的注释构成 [14]。对于3D网络预训练,我们采用具有三维卷积的ResNet,二维图像转换为伪3D格式,使用变维变换作为输入。对于ImageNet分类任务的预训练,我们首先调整图像的大小为3 × 1 × 256 × 256,然后随机裁剪它们的大小为3 × 1 × 224 × 224。模型经过训练100代的交叉熵损失,学习率为初始设置为1e−1。此外,我们采用了更好的标签平滑泛化能力。对于MS-COCO的预训练,我们采用了改进的FPN架构。我们进行水平翻转以及多尺度训练(384, 448, 512, 576, 640)作为数据扩充。通过ImageNet或MS-COCO的预先训练,我们能够获得用于迁移学习的三维骨干网。预训练的3D骨干可以方便地适应新的成像任务。为了在目标任务上训练新生成的模型,我们可以微调网络中的所有层,或者保留其中一些较低级别的层固定并只微调后续的层。在我们所有实现的任务中,我们对所有层进行了微调。在MS-COCO的预训练中,我们的探测ResNet3D-18模型的平均精度为中等(MAP)为34.2,而我们的检测模型与ResNet3D-50和P3D-63的检测性能相当(37.0 vs 36.5)。
在肺结节的分类和分割数据集中我们应用相同的数据对ACS的模型训练和评估。肺结节分割,共2142个样本用于培训,526个样本用于测试 [15] [16] [17]。报告系数得分用于模型比较,不确定的注释(第3级)被进一步忽略,以减少恶性肿瘤评估的模糊性。按分组级别进行良、恶性二元分类,良性为1、2级恶性为4、5级。最后,与ACS一样,我们收集了1633个肺结节进行分类,包括1156个良性结节和556个恶性结节结节。我们使用一系列指标来评估两个类分类问题,包括AUC,准确性,灵敏度,评价的特异性、精密度和f得分,其中,AUC是主要的比较指标。为了进一步研究区域差异对测量结果的影响迁移学习的表现,我们进行了消融研究。对于结节分类,我们使用Adam 优化器以5e−4的初始学习率(lr)来训练所有的比较100个代的模型。学习率降低了在30、60和90期之后分别是10倍。初始lr的和最小lr的1e−6用于所有的结节分割除了具有U-Net结构 [18] [19] 的Genesis以外的型号Lr设置为1e−4以获得最佳性能 [20] [21]。具体来说,通过保持茎层上的预处理训练权值和第一个ResBlock固定,在对目标任务(进行微调时,我们获得95.82%的骰子分数。
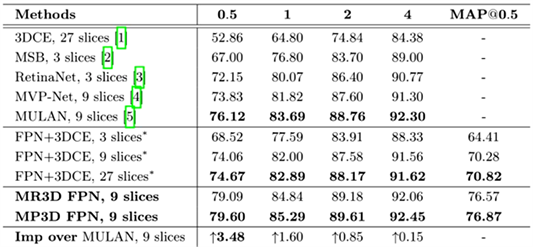
Figure 4. Sensitivity of model method under different FPs
图4. 模型方法在不同FPs下的灵敏度
之后使用的训练集是NIH DeepLesion,见图5,是病灶通用检测数据集,一个用于通用病变检测的大规模数据集,包含32,735个病变,分布在32,120个轴向CT切片上,并且在键片上提供了边界框。DeepLesion的官方拆分是使用训练集(70%)、验证集(15%)和测试集(15%),在我们的实验中。我们对所有比较的方法进行了评价,该测试通过报告不同假阳性时的敏感性设定(FPs)和它们的平均值(mFROC)。至于前处理,Hounsfield单元(HU)被夹进[1024, 1050]。我们还实现插值z轴归一化,所有CT切片的间隔为2.5mm。训练结果与之前的SOTA方法相比较可以看出在不同误报下的灵敏度和MAP@0.5都比之前的SOTA方法要改进很多,展现了3D模型的优势,以及MP3D网络对于上下文信息的建模能力。普通ResNet3D-18采用7 × 7 × 7,然而当预先训练时,我们提出的伪三维输入,其深度只有3。大卷积核的部分将不会被更新再训练。为了解决这个问题,我们采用了一种网络架构,即ResNet3DV1c-18的训练。ResNet3DV1c-18型号取代了7 × 7 × 7核配3 × 3 × 3核,这样所有这些参数将能够在训练前更新。实验结果表明,这种改变会大大改善病变模型的性能检测任务,适当的训练3D内核对于目标的高级性能是必不可少的任务。
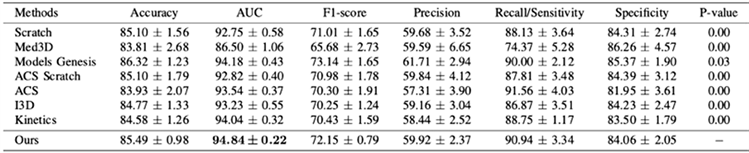
Figure 5. LIDC and LITS segmentation data set effect
图5. LIDC以及LITS分割数据集效果
目前在医学图像领域相关的训练任务中,生成预训练的任务中往往需要大量的标注数据集,一般会采用自监督的学习方法对未进行标注的数据通过对比学习的方法来训练预训练参数,但是对于一些普通的医学图像任务,会因其缺少一些监督信息,从而导致难以分辨其特征而不能进行信息挖掘。本项目为了验证程序在不同数据场景的有效性,在不同数据集上测试训练效果。分别在LIDC-IDRI数据集,LITS肝脏数据集,NIH DeepLesion数据集上进行分类,见图6,分割以及检测任务。并且用之前的SOTA方法同样对这些数据集进行训练,比较训练效果。比较结果是本项目的训练方法在这些训练集上均比原有SOTA方法有一定的提升。此外,我们在肝脏数据集的训练中,对分割图像的结果进行了可视化处理,在可视化处理中,本想能够完整分割出对应区域,进而证明了本项目方法的可实际应用性。为了验证本项目方法对于不同数据量的效果,我们基于NIH DeepLesion的数据集对预训练模型进行了微调,训练不同百分比下的此数据集,可视化中的每一幅图片都展示了在某一种具体指标上的不同方法之间的效果上的对比,其中最后一幅图片展示的meanFROC是对检测模型整体效果的一个描述。可以看出在同样的训练轮次,我们的预训练模型在每一种数据量下都可以取得比从头训练更好的效果,而且随着数据量不断的缩小,这个效果的差距也会被不断地放大。可以观察到,我们提出的预训练ResNet3D的模型明显优于竞争方法。我们的预训练的模型优于从零开始训练的模型2.09%,证明了这种模型的有效性。
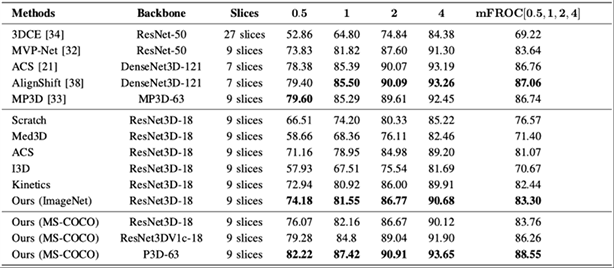
Figure 6. LIDC classification data set effect
图6. LIDC分类数据集效果
4. 结语
目前,基于医疗影像的辅助诊断技术正处于快速发展阶段,但是受医学影像数据量的制约,使得基于深度学习的建模方法无法向更复杂的模型进行探索。本文从3D监督预训练的全身病灶检测出发,概述了医疗影像病灶图像的成像特点,针对病灶检测及分割任务对现有方法进行了归类总结,并阐述了当前医学影像检测和分割的难点。分别从医学病灶检测搭建模型思路、开发技术和算法、面临的问题和解决方法、基于四个训练集下全监督3D模型较传统方法的优势与结果说明等方面进行了总结。最后,针对医学领域内运用计算机技术增强方法对于3D监督预训练进行了对比分析。
基于3D监督预训练的全身病灶检测可以大大提升3D模型训练的收敛速度,不仅有着十分高效的速度,同时在显著提升了小规模数据集上的模型精度。该模型效果在大多任务中都表现优异,有着明显的优势,而且可以以生成高质量清晰的图像,具有拟合高维数据的能力,被广泛应用于医学影像处理领域,哪怕与目前最先进、最尖端的自监督和全监督预训练模型相比,该模型都毫不逊色。虽然当前仍有一些公开可用数据集存在缺陷,例如图像数量过少、偏倚度过高等问题尚未解决,对于3D医学影像的问题也并未找出很好地预训练参数,但是相信随着医学和计算机技术的逐步发展,随着两者技术融合的不断深入,基于3D监督预训练的全身病灶检测技术会逐步趋于完美,为全人类提供便利。此外,针对医学病灶的成像特点以及数据量的大小构造合适的网络结构,也将是未来医学病灶检测研究的一大趋势。