1. 引言
染色体是重要的遗传信息载体,每个健康的人类细胞中存在23对染色体,其中包括22对常染色体和一对性染色体,在相同的生长阶段呈现各自固定的形态 [1] 。染色体异常通常表现为数量异常或结构异常,会导致许多先天性疾病,如:Edwards综合征,唐氏综合征等 [2] 。因此,染色体异常的识别已成为产前诊断和遗传疾病早期筛查的关键 [3] 。染色体核型分析,如图1,是从给定的细胞图像中分割出染色体实例,并根据它们的类别将这些实例排列到相应的核型中,通过提取染色体长度、着丝粒位置、长短臂比例以及是否有随体等信息来分析、比较、分类和编号染色体,从而识别染色体异常,如缺失、增加和突变 [4] 。在医学上,染色体核型分析已成为遗传疾病筛查、产前诊断和肿瘤遗传学研究中不可或缺的重要手段。

Figure 1. (a) An microscopic image of human metaphase cell; (b) Chromosomes karyotype image
图1. (a) 人中期细胞显微图像;(b) 染色体核型图
然而,染色体作为一种非刚性物质,在细胞核中经常会发生接触和重叠 [5] ,这使得目前大多数染色体核型分析都需要经验丰富的专家从给定的细胞图像中手动分割出所有染色体实例,会受到疲劳、注意力下降等主观因素的影响,耗时、昂贵且容易出错。因此,重叠染色体的自动分割已成为计算机辅助核型分析中亟待解决的问题。
在染色体分割任务中,基于几何特征的分割方法 [6] [7] [8] 已经取得了一些进展,大多是确定重叠部分的凹点后进行分割,但是由于染色体固有的非刚性性质使得染色体簇的形态具有多样性和不确定性,导致有效凹点可能会误判,因此基于几何特征的重叠染色体分割方法往往性能较差。
近年来,深度学习已被广泛应用于图像处理领域,目前,基于卷积神经网络的图像语义分割方法陆续出现,如FCN [9] 、SegNet [10] 、DeepLab [11] 、CE-Net [12] 等。然而,由于医学图像结构复杂,目标小,单个目标的边界模糊且常与正常组织重叠,导致医学图像的语义分割仍具有很大的挑战性。U-Net [13] 作为一种高效实用的卷积神经网络,在医学图像分割任务中表现出显著优势,一些改进的U-Net方法被用于重叠染色体分割任务中,Hu等人 [14] 构造了两层池化的Sim-U-Net来分割重叠染色体,减少三分之一的层数和块数来降低计算和存储成本,但是由于该方法的输入图像较小,经过简化模型的操作后丢失了较多的特征细节;Saleh等人 [15] 认为网络中卷积操作和池化的增加有利于提取更多的输入特征信息,因此,为提高分割精度,他们在UNet中建立了三层池化,并使用测试时间增强(TTA)方法用于重叠染色体分割任务,通过保留前三个下采样和相应的上采样块,分割精度和IoU略有提高。Song等人 [16] 提出了一种Compact Seg-UNet网络,结合了SegNet和U-Net的优点,增加网络层数,增强了深度特征学习能力和训练效能,在重叠染色体的分割性能上,该模型的IoU分数高于以上两个网络模型。
然而研究发现,现有的重叠染色体分割方法中使用的标准卷积大多只进行特征提取,不关注通道间信息的依赖相关性,且随着网络层数的增加,网络可能会出现梯度消失和网络退化的现象;网络中的池化层在降低图像分辨率的同时,往往会丢失很多对图像分割具有重要作用的位置信息;同时直接将编码器与解码器中对应特征进行拼接会导致语义差距过大的问题。针对上述问题,本文进行了一系列研究,提出了一种端到端的基于多尺度残差注意力的重叠染色体分割模型(RSE-UNet),用于对重叠染色体图像进行高精度分割,本文的主要贡献如下:
1) 采用残差注意力模块(Residual block with Squeeze and Excitation, ResSE)代替U-Net中原有的标准卷积,在残差块的基础上融合通道注意力机制,提高模型的全局感知能力;
2) 提出多尺度密集金字塔空洞卷积模块(Multi-scaled Dense Pyramid Atrous Convolution, MDPAC),扩大感受野,实现多尺度深层特征融合;
3) 设计了一种残差路径(Residual Path, ResPath)式的跳跃连接方式,解决特征拼接时出现的语义差距问题;
4) 使用focal loss损失函数,缓解数据类别失衡问题,优化训练目标。
2. 模型方法
2.1. 网络结构
针对重叠染色体图像的高精度分割,本文构建了RSE-UNet网络,整体采用了端到端的编码器–解码器结构,主要包括残差块(Resblock)、残差注意力(ResSE)模块、多尺度密集金字塔空洞卷积(MDPAC)模块和残差路径(ResPath),池化层数为4层,以平衡深度语义信息提取和高精度细节分割的要求,网络结构图如图2所示。对于编码器结构,通过一个Resblock和四个ResSE模块进行特征提取,使用2 × 2最大池化进行下采样。Resblock由两层3 × 3卷积、批量标准化(BN)、ReLU激活函数和跳跃连接组成。ResSE模块在残差模块的基础上增加通道注意力机制,通过对每个通道重新计算权重,自适应地学习通道间依赖关系,提升模型对通道特征信息的敏感性。为了加强空间特征信息的提取,本文提出MDPAC模块,该模块中使用了膨胀率不同的三层空洞卷积提取多尺度特征,在扩大感受野的同时减少信息的损失,并使用密集连接的方式将每个卷积层的输出,输入到前面所有未访问的卷积层,提高特征利用率。解码器结构与编码器结构对称,每个解码层先使用步长为2的2 × 2转置卷积进行上采样,转置卷积拥有可训练的卷积核参数,使图像恢复到与编码器对应的特征图尺寸。再将上采样后的特征图与编码器对应特征图进行拼接,以特征复现的方式载入ResSE模块,对特征进行逐级还原,最后使用一个1 × 1卷积层调整特征通道数,进行输出。除此之外,本文对跳跃连接进行了改进,设计了一种残差路径式的跳跃连接方式,原有的跳跃连接直接将编、解码器对应的特征图进行拼接,会产生较大的语义差距问题,而本文的ResPath先将编码器特征穿过带有残差连接的卷积层链,调整至与解码器对应特征接近的深度,再进行特征拼接,充分利用上下文信息,解决特征拼接时产生的语义差距问题。
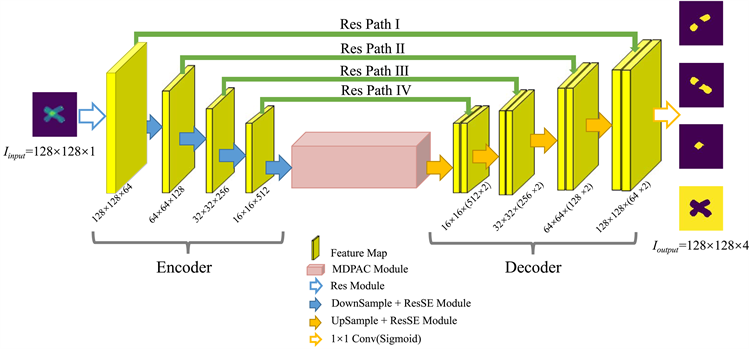
Figure 2. The structure of the RSE-UNet model
图2. RSE-UNet模型结构
2.2. 池化层数
在语义分割2任务中,多层池化是一种实现深度卷积的有效方法,但在增加感受野的同时,会导致分辨率降低,从而导致语义信息丢失,但这并不意味着应该放弃深度语义信息而追求特征图的清晰,因为深度语义信息也有助于网络实现复杂图像的分割。因此,必须在深度语义特征提取和高精度细节分割之间取得平衡。
研究表明,池化层数可以根据输入图像的大小和复杂度进行调整,例如GoogleNet [17] 、DenseNet [18] 、DeepLab [11] 都是使用五层池化来实现224*224大小自然图像的语义分割;对于96*96大小的脑神经图像,MADCNN [19] 基于DenseNet将池化层数减少到3层;U-Net [13] 对572*572大小的果蝇腹侧神经索(VNC)图像进行四层池化构建。Hu等人 [14] 和Saleh等人 [15] 将88*88大小的染色体图像的U-Net池化层数设置为2和3。由此可见,网络池化层数并没有严格的公式。与自然图像相比,医学图像特征相对单一,语义信息不足,因此,池化层数的选择不仅与图像的大小有关,还与图像的特征复杂度有关。本文的重叠染色体图像分辨率为128*128。通过比较不同池化层的网络分割性能,最终确定池化层数为4层。
2.3. ResSE模块
首先本文在网络的开始添加了一个Resblock,如图3所示,其中Addition表示加操作。Resblock由两个3 × 3卷积层和一个额外的跳跃连接组成。带有残差连接的卷积层有利于信息的传播,并能够防止网络退化和梯度消失等问题。卷积神经网络在开始的几个卷积层提取的特征是较低层次的细粒度信息,如纹理、边缘和位置信息等细节,因此在网络初始,本文平等地对待每个通道中的特征,并没有在Resblock中使用带有通道注意力的压缩–激励(Squeeze-and-Excitation, SE)机制。

Figure 3. Resblock module structure with two convolution layers
图3. 带有两层卷积的Resblock模块结构
接下来,本文将原U-Net的标准卷积块替换为ResSE模块,在残差块的基础上融合了通道注意力机制以获得通道间关系,ResSE模块的细节如图4所示。每个ResSE块由两个具有批量归一化(Batch Normalization, BN)和ReLU激活函数的3 × 3卷积层组成,并与SE机制集成。SE机制是一种允许网络进行特征再校准的通道注意力机制,学习各通道特征的重要程度并对每个特征通道进行重新加权,通过这种机制,网络可以有选择地权衡每个通道的重要性,从而输出更多有效的特征信息。SE机制的压缩squeeze (Fsq)操作首先对特征图进行全局平均池化,将一个通道内的空间特征编码为一个全局特征。输入特征映射为
,C、H和W分别表示特征图的通道数、高和宽,全局平均池化公式为
(1)
其中z为squeeze操作后的向量,维度为(1 × 1 × C)。全局平均池化将空间信息压缩至一个单位来获取C个特征通道的全局信息;而激励excitation (Fex)操作,使用两个全连接层学习通道间的非线性交互,最后通过Sigmoid函数为各个通道赋予合适的权重,公式如下
(2)
其中,
,
,δ为ReLU激活函数,σ为Sigmoid激活函数,降维系数r是一个超参数,
这里本文设置为16。向量z (1 × 1 × C)经过第一层全连接层后维度变为(
),再经过第二层全连接层后维度变为(1 × 1 × C),向量s为得到的通道权重值,维度为(1 × 1 × C)。图4中Scale (Fscale)操作为乘操作,通过权重向量s与特征图对应通道相乘,得到特征图
,公式如下
(3)
最后
与原始输入特征进行加操作,得到ResSE模块的输出特征。ResSE模块通过调整不同通道的权重,使模型聚焦于通道中的重要特征,从而提高模型的性能和泛化能力。
2.4. 多尺度密集金字塔空洞卷积模块
图像语义分割任务中,上下文信息以及不同尺度的特征信息对于分割结果十分重要。在之前改进U-Net的研究中,通常会通过连续的池化和卷积操作来降低特征分辨率,但是往往会损失较多空间细节信息;而通过扩大卷积核的大小来增大感受野,虽能捕捉到更多的纹理特征,但是模型的参数也会相应增加,不利于模型训练。
考虑到上述情况,本文提出了一种多尺度密集金字塔空洞卷积(Multi-scaled Dense Pyramid Atrous Convolution, MDPAC)模块,如图5所示,模块中空洞卷积的膨胀率逐层增加,具有较低膨胀率的层被放在低层级部分,具有较大膨胀率的层则放在高层,在密集连接的操作中,使用金字塔形式将每一层的输出
与较低层的所有输出
和初始特征图
拼接起来,并将这些拼接起来的特征图送入下一层,使得上层的卷积可以利用下层的特征,像素采样更加密集。

Figure 5. The structure of the MDPAC module
图5. MDPAC模块结构
(4)
通过不同膨胀率之间空洞卷积的密集连接,使得本文的网络可以在不损失分辨率的情况下获得更大的感受野以及密集的多尺度特征信息,提高特征利用率,从而提升网络分割的精度。
2.5. ResPath模块
针对U-Net网络中由朴素跳跃连接方式出现的语义差距问题,本文设计了一种新型跳跃连接方式——残差路径(ResPath)式跳跃连接,ResPath的结构如图6所示,在残差结构的基础上进一步融合了空间注意力机制与通道注意力机制,缓解语义差距的同时,自适应地捕捉输入特征之间的空间和通道相关性,提高网络性能。U-Net网络中原有的跳跃连接直接将编码器和解码器对应的特征图进行拼接,相当于在自顶向下和自底向上的路径之间直接传递特征,而本文认为编码器中的特征由于卷积层数较浅,是低层次的特征,而解码器中对应的特征由于卷积层次更深,是较高层次的特征,二者在语义之间存在较大差距,直接拼接会导致特征信息割裂。本文的ResPath式跳跃连接先将编码器特征穿过带有残差连接的卷积层链,调整至与解码器对应特征图接近的深度,再进行特征拼接,以充分利用编码器模块中丢失的空间和通道信息来弥补收缩和扩张路径中的语义差距。并且ResPath在过程中结合了空间注意力机制与通道注意力机制,在提取特征时更加准确地关注不同的空间区域和通道信息。具体而言,空间注意力机制可以使模型更好地关注特定的空间区域,从而提高对局部特征的感知能力;而通道注意力机制则可以使模型更好地区分不同通道的重要性,从而提高对全局特征的感知能力,在网络中更高效地利用编码器的特征信息,进而提高模型的性能和泛化能力。在这里,本文使用了四个ResPath模块来替代原来的跳跃连接。这四个模块分别表示为ResPath I、II、III、IV。考虑到在较浅的连接中,编码器和解码器之间有更大的语义差距,所以ResPath I模块的残差块数量最大。每个连接路径的配置参数如表1所示。
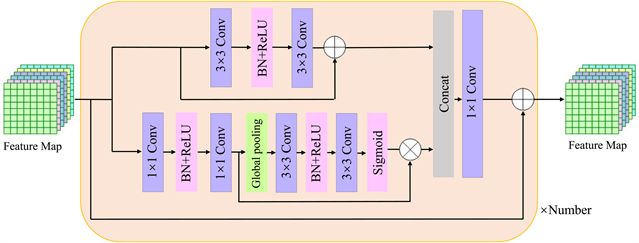
Figure 6. The structure of the ResPath
图6. ResPath结构

Table 1. ResPath module parameter configuration table in RSE-UNet network
表1. RSE-UNet网络中ResPath模块的配置参数表
2.6. 损失函数
在医学图像分割领域最常见的损失函数为二值交叉熵损失函数(cross-entropy, CE),公式如下
(5)
其中,p为预测值,y为真实标签。在本任务中,重叠染色体α和β的目标分割区域可以分为四个部分,每个目标区域的分割通过像素级分类来实现。然而这里存在着严重的类别失衡问题,如染色体区域(前景)占整个图像的一小部分,而周围的背景占比很高,前景、背景元素的像素比例差距过大,每个类别对损失的贡献不同,正样本过少时,负样本就会在损失函数占据主导地位。由于损失函数的倾斜,模型训练过程中会倾向于样本多的类别,造成模型对少样本类别的性能较差,因此交叉熵损失函数不再适用。
为解决这一问题,在本文中,本文引入了focalloss [20] ,它不仅能控制正负样本的权重,也能够控制易分类和难分类样本的权重,这些样本分类的难易程度由网络的输出和真实的偏差决定,实现网络自适应调整,能够解决正负样本极不平衡和难分类样本学习的问题,公式如下
(6)
(7)
(8)
在这里,
反映了预测值与真实类别y的接近程度,
越大说明越接近类别y,即分类越准确。α和γ是focalloss中的超参数,α用于控制正负样本的权重,当某一类样本数量特别少时,则可以通过增加该类的α值,来提高模型对该类别的关注度,处理样本不平衡的问题;γ用于控制易分类和难分类样本的权重,当γ = 0时,focalloss等价于上文中的交叉熵损失函数,而γ > 0时,focalloss逐渐增加对难分类样本的关注度。在本文中,重叠区域的α设置为0.25,γ设置为2,以解决样本不平衡的问题,并使损失函数倾向于难分类样本,提高模型准确度。
3. 实验和结果
3.1. 数据集
本文所使用的数据集是来自kaggle的公开重叠染色体数据集 [21] ,其中包含13,434张分辨率为94*93的重叠染色体图片,与Hu等人 [14] 、Saleh等人 [15] 、Compact Seg-UNet [20] 等重叠染色体分割任务研究中所使用的数据集一致。如图7所示,该数据集中的每张人类细胞分裂中期显微染色体图像都由一张包含DAPI (4’6-二脒基-2苯基吲哚)染色的人类中期染色体图像以及一张通过Cy3荧光端粒探针标记技术标记的端粒图像合成得到。正常人类拥有46条染色体,将所有染色体每4条为一组分割为12组后,每组
保留一条共计得到12条染色体,最终通过随机排列组合生成
对染色体。
对每组染色体对中的染色体施加随机的旋转和平移操作,使得2条染色体交叉重叠,该操作可以生成不同形态、大小的染色体重叠的图像。最终该数据集共计生成14,850张重叠染色体图像,其中1416张不存在重叠区域的被删除,保留13,434张94 × 93像素的重叠染色体图像,在本文中,这些图像被填充到128 × 128以满足输入要求。以两条重叠染色体的分割任务为例,将每组的2条染色体分别称作α和β,则目标分割区域包括四部分:染色体α的非重叠区域(
),染色体β的非重叠区域(
),染色体α与染色体β的重叠区域(
)和背景区域(
)。数据集中的每张图像都对应一组分割标签,如图8所示,图8(a)是染色体α和β重叠的图像,图8(b)~(e)是对应的分割标签图像,图8(b)和图8(c)分别对应染色体α和β的非重叠区域,图8(d)对应重叠区域,图8(e)对应背景区域。

Figure 7. Combination of DAPI (Chromosome) and Cy3 (Telomeres) grey images
图7. DAPI (染色体)和Cy3 (端粒)图像组合成的灰度图

Figure 8. Overlapping chromosomes image and category label. (a) Overlapping chromosomes image of chromosomes α and β, (b) Non-overlapping regions label of chromosome α, (c) Non-overlapping regions label of chromosome β, (d) Overlapping regions label of chromosome α and β, (e) Background label
图8. 重叠的染色体图像和类别标签。(a) 染色体α和β的重叠染色体图像,(b) α染色体非重叠区域标签,(c) β染色体非重叠区域标签,(d) α和β染色体重叠区域标签,(e) 背景标签
3.2. 实验细节
本文实验采用Pytorch框架,在Intel(R) Xeon(R) W-2175 CPU@2.50 GHz, 64 GB内存,NVIDIA GeForce RTX 2080Ti GPU处理器上运行。
本文采用5重交叉验证法 [22] 进行实验,以防止过拟合问题。实验中,测试集占全部数据集的20%,其余数据分为5个部分,其中4部分作为训练集,1个部分作为验证集,测试集与训练集和验证集保持分离。使用Adam优化器最小化损失函数,它能够根据梯度下降的情况自适应地修改学习率,初始学习率设置为默认值0.001,批处理大小设置为8,实验中使用早停(Early Stopping)策略,即每个epoch都使用验证集进行模型性能验证,并保存具有最佳验证性能(使验证集损失最小)的模型权重,如果在连续3个epoch后没有更新最佳验证性能,则进行学习率衰减,衰减率设置为0.5。如果在连续9个epoch后仍未更新最佳验证性能,则终止训练过程以避免过拟合,实验涉及的超参数如表2所示。

Table 2. The hyperparameters table of experiments
表2. 实验超参数表
3.3. 评价指标
本文将重叠染色体的分割任务看作像素级分类任务,即将图像中的每个像素分为4.1节中提及的4个类别,以实现图像分割任务,并使用交并比(intersection over union,以下简称IoU) [23] 和分割准确率(Accuracy,以下简称Acc)作为模型的性能评价指标。
1) 在重叠染色体分割任务中,目标分割区域通常只是整个分割区域的一小部分。也就是说,背景区域的分割精度通常较高,很容易造成模型性能优越的错觉。因此常用的分类评价指标如精确率、召回率等,不能完全评价网络性能。所以在本文中,本文采用交并比(IoU)作为模型的评价标准,如公式(9)所示。
(9)
其中A表示模型的分割结果,B表示对应的真实结果(ground truth),IoU越接近1,证明模型分割效果越好。染色体的重叠区域是一个难分割区域,因此以该区域的IoU作为评价指标可以更好地反应网络的分割性能。但是考虑到染色体的重叠区域很小,即使只有几个像素,一个很小的分割错误也会导致评估指标的显著降低,因此,本文不仅计算两条染色体重叠区域的IoU指标(记作IoU),同时引入两个染色体非重叠区域的IoU指标(分别记作IoU1、IoU2),共同评价网络性能。
2) 为了与现有模型的分割性能进行比较,本文还计算了分割准确率(记作Acc),其计算过程如公式(10)所示。
(10)
其中TP表示被正确预测为染色体区域的像素数,TN表示被正确预测为背景的像素数,ALL表示图像中的全部像素数。
3.4. 实验结果
本文提出模型对重叠染色体进行分割的可视化结果如图9所示,其中图9(a)是真实标签,图9(b)是RSE-UNet的分割结果。

Figure 9. RSE-UNet visual segmentation results. (a) Ground truth of overlapping chromosomes; (b) Segmentation results of RSE-UNet
图9. RSE-UNet可视化分割结果。(a) 重叠染色体真实标签;(b) RSE-UNet的分割结果
本文通过与6种最新的图像分割模型进行比较,验证了RSE-UNet模型在重叠染色体分割任务上的有效性。包括UNet [13] 、Hu等人提出的sim-UNet [14] 、Saleh等人提出的带有测试时间增强(TTA)的UNet [15] 、以及专门针对图像分割领域的DeepLabv3+ [11] 、SegNet [10] 、CE-Net [12] 。同时本文针对所提出模型进行了消融实验,以验证模型中每个模块的有效性,下面介绍本文开展的相关实验及对应的实验结果。
3.4.1. 不同池化层数对模型性能的影响
本文首先开展了池化层数量对RSE-UNet模型性能影响的实验,分别将模型中的池化层数量设置为3、4、5,并分别命名为RSE-UNet(3L)、RSE-UNet(4L)、RSE-UNet(5L)。它们在不同评价指标上的得分如表3所示,其中,RSE-UNet(3L)分割单个重叠染色体图像所需时间最短,但对深度语义信息的提取能力不足。因此,该网络对于简单的图像分割具有较高精度,但对于重叠区域过小或过大的复杂图像分割性能较差。相比之下,RSE-UNet(5L)能够提取出更深层次的语义信息,但也导致特征图分辨率过低,丢失了大量细节信息,不利于复杂图像的精确分割。而RSE-UNet(4L)结合了两者的优点,不仅具有适中的特征图分辨率,而且能够提取深层语义信息,所以其在测试数据集上目标重叠区域的IoU指标最高,且该网络在两条染色体的非重叠区域上的IoU指标和准确率也达到最高。因此,最终RSE-UNet网络选择四层池化的模型架构,以综合平衡深度语义提取和高精度细节分割的需求,提高重叠染色体的分割性能。

Table 3. Model performance comparison table with different pooling layers
表3. 不同池化层数的模型性能对比表
3.4.2. 模型性能对比
本节主要介绍RSE-UNet与其他染色体图像或医学图像分割模型的性能对比,RSE-UNet与其他6个网络在不同评价指标下的分割性能如表4所示。从图中可以看到,无论是以重叠染色体区域的IoU指标,还是完整染色体的IoU指标和准确率(Accuracy)作为评价标准,RSE-UNet网络的性能都优于其他网络,并远好于目前流行的医学图像分割模型CE-Net。

Table 4. Performance comparison table between RSE-UNet and other models
表4. RSE-UNet与其他模型的性能对照表
可以看到本研究提出的RSE-UNet网络在指标上均有所提升。与在本任务中表现最好的U-Net相比,本文方法在重叠染色体分割任务上将IoU指标和准确率分别从95.52%和98.10%提高到98.58%和99.41%,实现了3.06%和1.31%的提升;而与目前流行的医学图像分割模型CE-Net相比,本文将IoU指标和准确率分别从92.35%和98.01%提高到98.58%和99.41%,实现了6.23%和1.40%的提升;可以看出在重叠染色体分割任务中,RSE-UNet的性能远好于目前流行的医学图像分割模型CE-Net,主要原因是CE-Net中过度的池化操作导致DAC中的感受野远大于特征图大小,不利于多尺度特征的提取,RMP设计的池化大小也不利于上采样后图像原始大小的恢复,导致大量语义信息丢失,无法实现更高的分割性能。而实验结果看出,本文网络在准确率上略差于Saleh等人 [15] 提出的方法,主要原因是其方法中使用了TTA,而TTA是一种通常用于训练阶段之后提高测试集性能的方法,本研究的重点是比较不同网络模型的效果,因而在本文实验中并未使用。
为了更为直观地展现本文提出网络的性能,本文针对三种典型重叠情况,将RSE-UNet的分割结果与U-Net,CE-Net对重叠染色体的分割结果进行可视化对比,如图10所示。图10(a)为两条染色体的重叠区域很小的情况,图10(b)为两条染色体交叉,重叠区域大小适中的情况,图10(c)为两条染色体明显重叠,重叠区域很大的情况。通过对比发现,RSE-UNet在多尺度重叠区域的分割场景中表现更好,而CE-Net和U-Net在目标区域很小的情况下表现较差。

Figure 10. U-Net, CE-Net, RSE-UNet model segmentation results in three typical cases
图10. 三种典型情况下U-Net、CE-Net、RSE-UNet模型分割结果
3.4.3. 消融实验
为了评估RSE-UNet网络中的ResSE模块,MDPAC模块,以及ResPath模块的有效性,本文对RES-UNet网络进行了消融实验,共设计了7组对比实验,评估不同情况下网络的分割性能,如表5所示。具体而言,引入了ResPath后,缓解了编解码器之间的语义差距,分割性能较U-Net略有提高。在此基础上引入了多尺度密集金字塔空洞卷积模块后,能够自适应融合多尺度特征,分割性能又有进一步提升;而使用ResSE块对整体分割性能的提升是显著的。因此可以看出,消融实验中涉及的各个模块都有助于提高分割性能,其中ResSE模块带来的性能提升是最大的,MDPAC模块和ResPath模块也对RSE-UNet网络的整体分割性能具有一定贡献。

Table 5. Results of ablation experiments
表5. 消融实验结果
4. 结论
本文提出了一种RSE-UNet网络,用于高精度分割重叠染色体图像,能够为后续的染色体核型分析和疾病诊断提供更可靠的输入,使计算机辅助核型分析更加快速准确,节约医学成本,提高异常诊断的效率和准确度,对染色体异常所引起相关疾病的诊断和治疗具有重要意义。该网络采用Res-SE模块代替U-Net中原有的标准卷积,引入通道注意力机制,促进信息传播的同时,更关注通道间的特征依赖信息;设计的ResPath减少了编码器、解码器间的语义差距,更精确地恢复高分辨率图像;提出的多尺度密集金字塔空洞卷积模块,扩大特征感受野,实现多尺度特征密集融合,进一步提高分割精度;同时该网络设置了适当数量的池化层,以平衡深度语义信息提取和高精度细节分割的需求。为了验证本文提出的RSE-UNet模型的有效性,本文将其与6种先进的图像分割方法进行比较,从评价指标上来看,RSE-UNet网络的性能优于其他模型,提高了重叠染色体图像的分割精度。
在未来的工作中,可以尝试将本文所提出的RSE-UNet网络与目标检测算法相结合。首先使用目标检测算法在图像中检测重叠染色体,后基于本文模型进行重叠染色体图像的分割,因此,将RSE-UNet作为骨干网络应用于端到端的染色体识别和分割将成为本课题今后的主要研究方向。