1. 引言
原发性醛固酮增多症(primary aldosteronism, PA)是一种常见的继发性高血压,占高血压患者10%以上,在难治性高血压中占17%~23%。原醛症主要是肾上腺皮质球状带自主分泌过量醛固酮,过量的醛固酮作用于肾脏远曲小管和集合管上的醛固酮受体,导致水钠潴留,钾排出量增多,血容量增多,肾素–血管紧张素–醛固酮(RAAS)系统活性受制,临床表现主要为高血压、低血钾 [1] 。与原发性高血压(essential hypertension, EH)相比,更容易合并靶器官损害,有研究显示PA患者脑卒中、心肌梗死、心房颤动的发生率分别为EH患者的4.2,6.5及12.1倍。还有学者发现,PA患者与EH患者相比,靶器官损害分别为:心肌梗死(4.4%和1.7%)、心房颤动(3.9%和1.1%)、冠心病(3.9%和1.1%)、心力衰竭(4.1%和1.2%) [2] 。其造成的心脏、肾脏等靶器官的损害严重影响患者的生活质量 [3] 。研究发现,DNA的甲基化可以通过表观遗传学修饰,调控基因转录过程,从而在基因未发生变化的情况下,基因功能发生了变化,最终导致表观遗传变化 [4] 。有学者收集15例智力障碍或发育迟缓患儿,进行SNP微阵列检测后发现2例存在微缺失,且均为新生变异;13例SNP微阵列检测结果为阴性的患儿,经目标基因捕获高通量测序、基因变异–临床表型关联分析及遗传学分析,确诊5例患儿为单基因病,2例为疑似,6例阴性 [5] 。
近年来,随着微阵列分析和测序技术的发展,生物信息学技术已经成为筛选和识别致病基因,诊断和治疗疾病的有效技术手段 [6] 。因此,本研究旨在通过对GEO数据库中PA的基因表达谱及DNA甲基化谱进行生物信息学分析,揭示与PA疾病发生相关的核心基因及信号通路,为进一步阐明PA的发病机制提供有价值的信息,并为PA的诊断、治疗提供新的思路。
2. 研究对象
以“primary aldosteronism”作为关键词,在GEO数据库中筛选到两个PA的表达数据集:GSE60042和GSE60043。下载相应注释平台文件分别获得各数据集的基因表达矩阵。GSE60042作为PA基因表达数据集,GSE60043作为DNA甲基化数据集 [7] 。
3. 研究方法
3.1. 微阵列芯片初始数据的获取
从NCBI GEO公共数据库下载GSE60042的Series Matrix File数据文件,注释平台为GPL14550,共14组转录组数据,AAG组7例(n = 7),APA组7例(n = 7);从NCBI GEO公共数据库下载GSE60043的甲基化450 k矩阵,其中AAG组7例(n = 7),APA组7例(n = 7)。
3.2. 差异基因的筛选
使用R包“Limma”对转录组数据集中的两组患者进行差异分析,差异基因筛选条件为|logFC| > 1 & adj.P.Val < 0.05。DNA启动子甲基化通过转录沉默来调控其表达。本研究利用R包“CHAMP”分析GSE60043数据集中的甲基化数据,分析AAG组与APA组中探针的甲基化水平,以及筛选差异甲基化基因,差异甲基化的筛选条件为|logFC| > 0.2 & adj.P.Val < 0.05。通过韦恩图筛选高甲基化低表达和低甲基化高表达的基因。
3.3. GO和KEGG功能注释
使用R包“clusterProfiler”对基因进行功能注释,以全面探讨这些差异基因的功能相关性。基因本体论(GO)和京都基因与基因组百科全书(KEGG)被用来评估相关的功能类别。p值和q值均小于0.05的GO和KEGG富集通路被认为是显著性类别。
3.4. Lasso回归和SVM算法的特征选择过程
我们使用Lasso logistic回归和SVM算法对醛固酮增多症的诊断标志物进行特征选择。Lasso算法使用“glmnet”软件包。此外,SVM-RFE是一种基于支持向量机的机器学习方法,通过删除SVM生成的特征向量来寻找最佳变量,并通过“e1071”软件包建立支持向量机模型,进一步识别这些生物标志物对疾病的诊断价值。
3.5. GSEA
基因集富集分析(GSEA)用于筛选数据集中可能与PA相关的KEGG通路。GSEA采用GSEA-4.0.1 (Java)版本进行,背景基因集为c2.cp.kegg.v7.5.symbols.gmt,其中置换次数设置为1000,置换类型设置为phenotype。
3.6. 统计分析
统计分析采用R语言(version 3.6)进行。所有统计检验均为双侧,p < 0.05具有统计学意义。
4. 结果
4.1. 原发性醛固酮增多症与正常肾上腺组织的差异基因表达及分布特征
我们从NCBI GEO公共数据库下载GSE60042共14组转录组数据,根据GSE60042的表达谱进行差异分析,差异基因筛选条件为:|Log2FC| > 1 & adj.P.Val < 0.05,共筛选500个差异基因,其中上调基因192个,下调基因308个(图1、图2)。另一方面,我们从NCBI GEO公共数据库下载GSE60043的数据文件,共14组甲基化450 K的数据,用ChAMP包进行差异甲基化位点分析,共筛选出3015个差异甲基化探针,其中下调探针2975个(|Log2FC| < 0.2 & adj.P.Val < 0.05),上调探针40个(|Log2FC| > 0.2 & adj.P.Val < 0.05) (图3、图4)。
4.2. 差异表达基因GO和KEGG富集分析结果和核心基因筛选结果
对高甲基化低表达以及低甲基化高表达的基因取交集,韦恩图结果显示共有29个交集的mRNA(2 + 27),我们将这29个mRNA作为后续分析的候选基因集(图5、图6)。
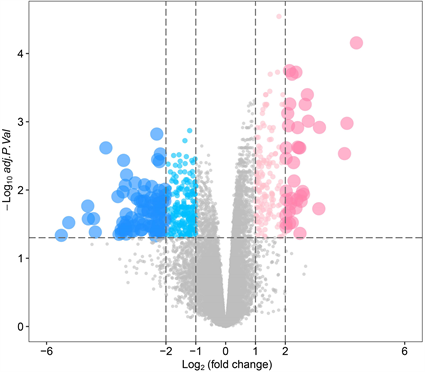
Figure 1. Volcano map of differentially expressed genes. Note: Blue represents significantly down regulated genes, pink represents significantly up regulated genes, and gray represents genes with no statistically significant differences in expression
图1. 差异表达基因的火山图。注:蓝色为显著下调基因,粉色为显著上调基因,灰色为表达差异无统计学意义的基因
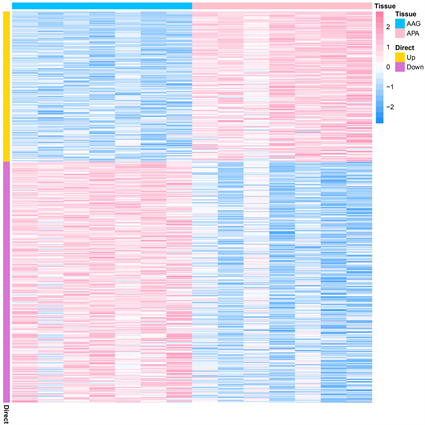
Figure 2. Heat map of differential gene expression profile. Note: Blue represents low expression, pink represents high expression, and the darker the color, the higher or lower the expression level of differential genes
图2. 差异基因表达谱热图。注:蓝色为低表达,粉色为高表达,颜色越深为差异基因的表达量越高或越低
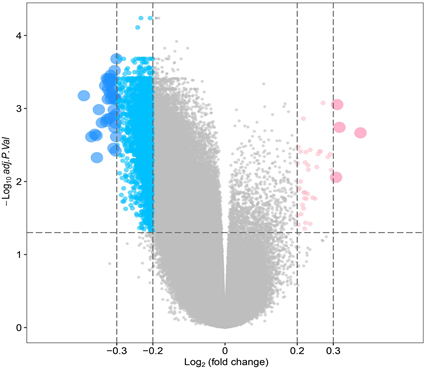
Figure 3. Volcano map of differentially expressed genes. Note: Blue represents significantly down regulated genes, pink represents significantly up regulated genes, and gray represents genes with no statistically significant differences in expression
图3. 差异表达基因的火山图。注:蓝色为显著下调基因,粉色为显著上调基因,灰色为表达差异无统计学意义的基因
 图4. 高表达前10位差异基因和低表达前10位差异表达基因热图。注:蓝色为低表达,红色为高表达,颜色越深为差异基因的表达量越高或越低
图4. 高表达前10位差异基因和低表达前10位差异表达基因热图。注:蓝色为低表达,红色为高表达,颜色越深为差异基因的表达量越高或越低
Figure 4. Heat map of high expression top 10 differentially expressed genes and low expression top 10 differentially expressed genes. Note: Blue represents low expression, red represents high expression, and the darker the color, the higher or lower the expression level of differential genes

Figure 5. Venn diagram of 29 intersections of mRNA (2 + 27)
图5. 29个交集的mRNA (2 + 27)韦恩图
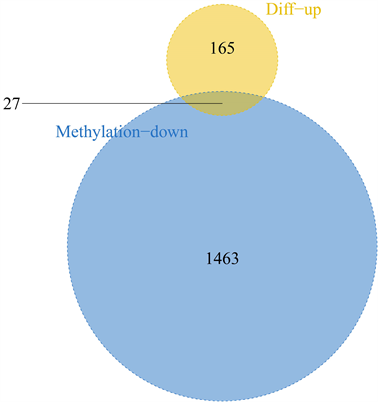
Figure 6. Venn diagram of 29 intersections of mRNA (2 + 27)
图6. 29个交集的mRNA (2 + 27)韦恩图
随后我们对这29个mRNA进行富集分析。GO富集结果表明,基因主要富集的通路是ATPase activity、polyol transport、regulation of interleukin-13 production、renal system process。KEGG富集结果表明,基因主要富集的通路是Pancreatic secretion、cAMP signaling pathway、Endocrine and other factor-regulated calcium reabsorption、Thyroid hormone signaling pathway、Aldosterone-regulated sodium reabsorption、Aldosterone synthesis and secretion (图7~9)。
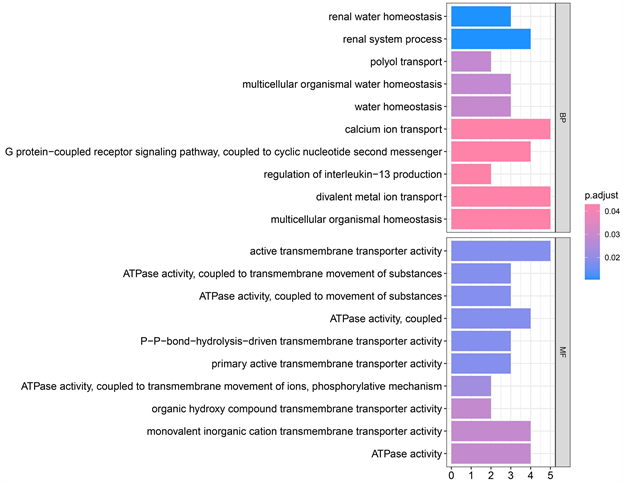
Figure 7. GO Analysis results. Note: color represents the size of the P-value
图7. GO分析结果。注:颜色表示P值的大小

Figure 8. KEGG analysis results. Note: color represents the size of the P-value
图8. KEGG分析结果。注:颜色表示P值的大小
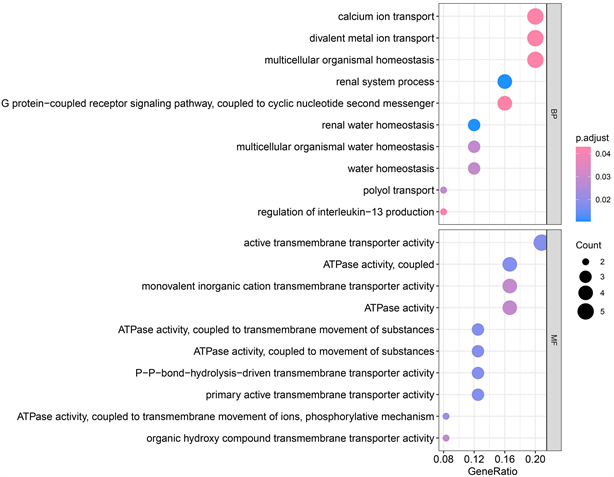
Figure 9. KEGG analysis results. Note: Bubble size represents the number of enriched genes, and color represents the size of the P-value
图9. KEGG分析结果。注:气泡大小表示富集的基因个数,颜色表示P值的大小
4.3. 核心基因表达水平验证
为了进一步找出影响醛固酮增多症的特征基因,我们联合使用lasso回归和SVM特征选择算法对29个候选基因进行筛选。结果显示,Lasso回归共识别7个基因作为醛固酮增多症的特征基因,图10,另一方面,我们通过SVM-RFE算法评估醛固酮增多症中的特征基因。结果显示,通过筛选准确率最高以及错误率最低的前10个的特征基因,与Lasso回归算法筛选出来的特征基因取交集,共筛选出4个交集基因。图11这4个基因将作为醛固酮增多症的核心基因,它们分别是:ABCB4、AQP2、ARG2和CLRN1
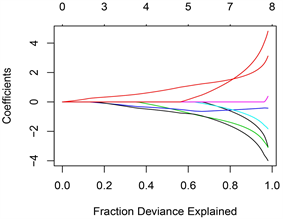
Figure 10. Lasso regression and SVM feature selection algorithm for screening 29 candidate genes
图10. Lasso回归和SVM特征选择算法对29个候选基因进行筛选
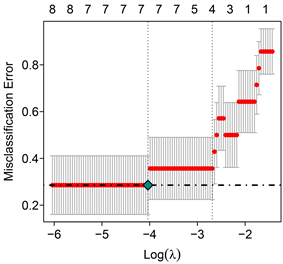
Figure 11. Screen the top 10 feature genes with the highest accuracy and lowest error rate, intersecting with the feature genes screened by the Lasso regression algorithm
图11. 筛选准确率最高以及错误率最低的前10个的特征基因,与Lasso回归算法筛选出来的特征基因取交集
5. 讨论
原发性醛固酮增多症是由于肾上腺皮质过度分泌醛固酮,使体内醛固酮水平升高,醛固酮的主要作用是保钠、保水、排钾,从而使体内水钠潴留、血容量增加,引起血压升高,是临床上最常见的继发性高血压 [8] 。PA患者通常患有长期中、重度高血压,且对多种降压药疗效欠佳。PA患病率在1、2、3级高血压中分别为1.99%、8.02%、13.2% [1] 。长期的血压控制不佳使PA患者总体心血管事件发生率高于EH患者。已有研究表明,PA中约有50%的患者合并糖代谢紊乱以及23%患者合并脂代谢异常 [9] 。目前认为PA引起糖代谢异常的原因主要是醛固酮分泌过多影响胰岛β细胞损害和靶器官对胰岛素敏感性降低两个方面 [10] 。PA导致脂代谢紊乱的机制目前尚不明确,有研究结果显示可能与高醛固酮影响脂肪细胞的分化和功能有关 [11] 。PA患者糖脂代谢的紊乱也会加重靶器官的损害。近年来根据经典遗传学方法和基于二代测序的基因组测序、全外显子测序或转录组测序(RNA Sequencing, RNA-Seq)已报道过影响钾通道的体细胞突变 [12] ,影响ATP酶功能的体细胞突变 [13] ,影响钙通道活性的CACNA1D、CACNA1H体细胞突变 [14] ,影响氯通道的CLCN2体细胞突变,还有CTNNB1基因突变、PRKACA基因突变等 [15] 。生物信息学是将生物数据存储在计算机,并利用计算机作为工具对生物数据进行检索和分析的科学,涉及生物学、医学统计学、应用数学以及计算机科学等学科知识。由于测序技术的不断提高和成本不断降低,越来越多的领域应用到生物信息学分析。从疾病的筛查、诊断到治疗、预后,还有肿瘤个性化用药、遗传病筛查等,通过从海量测序数据中挖掘有效的信息,为临床诊疗提供新思路 [16] 。基因表达综合数据库(Gene Expression Omnibus, GEO)为全球研究学者提供高通量基因表达数据,通过GEO等数据库的公共数据集进行生物信息学分析已广泛应用在基因组水平上筛选疾病的诊断、治疗和预后靶点 [17] ,这些方法帮助我们筛选原发性醛固酮增多症的差异表达基因(Differentially Expressed Genes, DEGs)。我们的研究发现,原发性醛固酮增多症的核心基因可能是ABCB4、AQP2、ARG2和CLRN1。ABCB4基因最初发现是与进行性家族性肝内胆汁淤积3型(PFIC3)的可遗传形式的胆汁淤积有关 [18] 。目前越来越多的研究表明,该基因在各种肝胆疾病中都有参与。ABCB4编码多药耐药蛋白3 (MDR3),这是一种介导磷脂酰胆碱(PC)流出胆汁的小管ATP结合盒(ABC)蛋白。到目前为止,有证据表明双等位基因或单等位基因ABCB4缺陷会导致或诱发多种人类肝脏疾病(PFIC3、低磷脂相关胆石症综合征、妊娠肝内胆汁淤积症、药物性肝损伤、短暂性新生儿淤积症) [19] 。这种磷脂被认为可以保护肝细胞和胆管细胞的管腔膜免受游离胆汁酸的去污作用 [20] 。AQP2是广泛分布于哺乳动物组织中的水通道蛋白,在水平衡中起主要作用。AQP2基因与胰岛素抵抗密切相关。血管紧张素II和醛固酮可以通过改变细胞内AQP2靶向和/或AQP2丰度以及加压素,在调节肾脏水的重吸收中发挥作用,引起血压升高 [21] 。目前有研究表明,AQP2蛋白在正常肾上腺髓质组织和髓质肿瘤(包括PCC、MPCC和NT)以及EAPGL中显著表达。AQP2的表达可能提示正常肾上腺组织的起源,其在肿瘤组织中的表达可能反映肿瘤发生过程中通过AQP2维持水代谢。AQP2可作为鉴别诊断肾上腺肿瘤的一个有价值的指标 [22] 。精氨酸酶是尿素循环(UC)中普遍存在的酶,其将L-精氨酸水解为尿素和L-鸟氨酸。两种哺乳动物精氨酸酶亚型,精氨酸酶1 (ARG 1)和精氨酸酶2 (ARG 2),两者都是通过调节L-精氨酸代谢、一氧化氮(NO)产生和炎症反应以及氧化应激,在β细胞功能、胰岛素抵抗(IR)和血管并发症的调节中起重要作用 [21] 。研究证实ARG2基因与糖脂异常密切相关 [23] 。本研究进一步分析了差核心基因涉及的具体信号通路,探讨核心基因影响醛固酮增多症进展的潜在分子机制。低表达ABCB4主要富集的通路是ALDOSTERONE-REGULATED-SODIUM-REABSORPTION、GLUTATHIONE-METABOLISM、VEGF-SIGNALING-PATHWAY等信号通路;低表达的AQP2主要富集的通路是ALDOSTERONE-REGULATED-SODIUM-REABSORPTION、LEUKOCYTE-TRANSENDOTHELIAL-MIGRATION、KEGG-AUTOIMMUNE-THYROID-DISEASE等信号通路;高表达ARG2主要富集的通路是PROTEIN-EXPORT、RNA-DEGRADATION、AMINO-SUGAR-AND-NUCLEOTIDE-SUGAR-METABOLISM等信号通路;低表达CLRN1主要富集的通路是TYROSINE-METABOLISM、ALDOSTERONE-REGULATED-SODIUM-REABSORPTION、AUTOIMMUNE-THYROID-DISEASE等信号通路。提示核心基因通过这些信号通路影响醛固酮增多症的发生和发展。
6. 结论
上述研究帮助我们更好地理解基因改变如何参与PA的发生和发展,并确定应该研究哪些基因和通路。最重要的是,我们发现了PA的4个核心基因可能作用的信号通路,有助于进一步探讨核心基因影响醛固酮增多症进展的潜在分子机制。
NOTES
*通讯作者。