1. 引言
2018年4月5日,《Cell》公布了泛癌症图谱(Pan-Cancer Altas),其分子特征是20,000多种原发性癌症和匹配的33种癌症类型的正常样本,其中,居于癌症发病率首位的肺癌是一种高度错综复杂的异质性疾病,目前肺癌的精确治疗已发展到新的阶段,已确定EGFR突变、ALK融合等七种不同的肺癌亚型,这七种不同的肺癌亚型需要不同的靶向药物进行精确治疗,然而,肺癌以及其它癌症的治疗也存在着许多问题,如何寻找未知的靶基因,确定未知的癌症亚型还需要更多地努力 [1] [2]。相似网络融合是一种有效确定癌症亚型的聚类方法,可以将病人不同的数据类型整合到一个网络中,通过相似网络融合,病人之间的弱相似性减少,降低了数据分析时的噪声,强相似性增加,增加了不同相似网络的数据间的互补性。2014年由Bo Wang [3] 等人首次提出相似网络融合方法((Similarity Network Fusion) SNF),利用TCGA计划中病人的三种特征DNA甲基化、mRNA表达以及microRNA表达三种数据构建病人的相似融合网络,最后对融合后的网络进行聚类,对比于其它利用单一种数据进行聚类的方法提高了聚类的性能,但也存在着不足,少量的数据样本对应着高维数据,降低数据噪声,扩大数据间的互补性还需要提高。2014年,Jiang Xingpeng [4] 改进了邻居的定义,提出了一致相似网络融合(CSNF),用一致k近邻代替原来的k近邻方法,以此来推断微生物之间的关系,提高了聚类性能。2017年,Zhang Yong [5] 等人提出一种新的基于相似网络融合的多视图聚类算法(RSNF),这种方法结合了随机森林的强度优势以及SNF聚类方法的鲁棒性优势,并将其应用于分析人体微生物数据,改善了SNF的聚类性能。2017年,Zhao Yaping [6] 提出了包含邻居信息的相似网络融合(NSNF),用包含邻居信息的多重紧密k近邻方法代替原来的k近邻方法来构建邻居间的相似网络,将其运用于癌症亚型聚类,与原有方法相比,聚类效果提高很多。2018年,张月 [7] 等人,提出了一种基于一致相似度网络融合的极化SAR图像非监督地物分类方法,修正了误分像素的类别标签,提高了地物分类的精度,和传统极化SAR图像地物分类方法相比,聚类性能有了很大的提高。2018年,Ning Chen [8] 提出了基于利用上下文信息改进SNF的信息检索(CI-SNF),对SNF融合相似性采用Jaccard距离,增强了样本的局部一致性,引入倒排索引技术,提高了聚类的效率,并且将其应用于歌曲识别、图像分类、癌症亚型识别以及药物识别中。
相似网络融合经过这几年有了很大的发展,但是,如何更精确地区分亚型,提高聚类的有效性一直是一个发展中的难题。本文提出了基于归一化欧氏距离和谱分邻相似网络融合方法(NSSNF),用两种方式改进了相似网络融合方法(SNF),一方面,用归一化欧氏距离对病人相似性中的距离重新定义,这样求得的更具有全局性,减少了病人相似数据的噪声;另一方面,用谱聚类分邻的方法重新定义病人邻居间相似网络中的邻居,分邻方法更加科学,同一组邻居强相似性增加,不同组邻居弱相似性减少,增加了数据间的互补性,融合网络更具有全局性,聚类效果优于SNF方法、NSNF方法和CSNF方法。
2. 方法
2.1. 数据来源
数据来源于TCGA网站,本文下载了五种癌症数据:肺癌(LSCC)、肾癌(KRCCC)、肠癌(COAD)、乳腺癌(BIC)和胶质瘤(GBM)。每种癌症数据都包含3种数据类型:DNA甲基化、mRNA表达、miRNA表达。每种癌症详细信息见表1。

Table 1. Detailed data for each cancer
表1. 每种癌症的详细数据
2.2. 相似网络融合
本文提出的方法归一化欧氏距离和谱分邻的相似网络融合(NSSNF)一共有五个步骤:构建病人相似网络、病人相似网络标准化、构建邻居间的相似网络、相似网络融合和对融合网络进行谱聚类。
2.2.1. 构建病人相似网络
假设有 个病人和 种数据类型(比如:DNA甲基化,mRNA表达miRNA表达)。病人的相似网络可以表示成
,其中顶点集V表示病人
,边集E表示病人之间相似性程度,病人之间的相似网络可以用
的相似矩阵来表示,其中
表示病人与病人间的相似性,受Danfeng Qin [9] 等人的启发,病人之间的相似性定义如下
, (1)
其中,
的值越大,代表着病人
和病人
相似性越高,
表示超参数并且可以经验性的给出,
[3] 是消除缩放的尺度参数,定义如下
, (2)
其中,
表示病人
病人
病人之间归一化欧氏距离,定义为
, (3)
表示病人
和
之间的欧氏距离,
表示距病人
最近的k个邻居归一化欧氏距离的平均值,Ni是指包含病人
在内距它归一化欧氏距离最近的k个病人组成的邻居。
本文用归一化欧氏距离为基础描述这种相似关系,构建病人之间的相似网络更具有全局性,和SNF方法相比,减少了数据噪声。
2.2.2. 病人相似网络标准化
因为要把不同数据类型的相似矩阵融合成一个矩阵,所以要对不同数据类型的相似网络进行标准化 [3]。病人相似网络被重新定义之后,病人相似性具有全局性,病人
和病人
之间的相似性
,标准化过程定义如下
(4)
从而,可以求出标准化后的病人相似网络
。
2.2.3. 构建邻居间的相似网络
下面通过谱分邻的方法构建邻居间的相似网络,谱分邻就是用谱聚类 [10] 算法定义病人邻居间相似网络中的邻居,步骤如下
1) 矩阵W作为相似性矩阵。
2) 求度矩阵D,其中
。
3) 求拉普拉斯矩阵
。
4) 求L最小的k个特征值对应的特征向量V。
5) 对特征向量V进行K-means聚类。
通过谱分邻方法,n个病人被聚成C组,一般情况下,病人邻居间的聚类数目为
。病人邻居间的相似网络定义如下
(5)
其中,
表示每一组邻居,每一组内的病人之间的相似性很高,不同组病人间的相似性为零,从而,可以求出病人邻居间的
。
本文通过谱分邻方法构建病人邻居间的相似网络,和SNF方法相比,同一组邻居间的相似性更高,不同组邻居间的相似性更低,提高了聚类效果。
2.2.4. 相似网络融合
相似网络融合采用了一种基于消息传递理论的非线性方法 [11],矩阵P包含所有病人的相似信息,而矩阵S只包含病人邻居间的相似信息,将矩阵P作为初始矩阵,矩阵S作为核矩阵进行迭代融合,进行一定次数的迭代,每种数据类型的网络逐渐收敛。
对于一种癌症,有n个病人和3种数据类型(DNA甲基化,mRNA表达和miRNA表达),通过公式(1)、(4)、(5),可以分别计算出病人DNA甲基化的相似网络
、标准化矩阵
和邻居间相似矩阵
,同理,计算出mRNA表达矩阵
、
和,miRNA表达矩阵
、
和
。令
,
,
,
,
,
。迭代步骤如下
, (6)
, (7)
, (8)
其中,t是迭代的次数。经过t次迭代之后,网络融合矩阵可用下列矩阵表示
。 (9)
从而,可以求出包含病人全局信息的融合网络
。
2.2.5. 对融合网络进行谱聚类
包含全局信息的融合矩阵P作为相似性矩阵进行谱聚类 [10],谱聚类的步骤如下:
1) 融合矩阵P作为相似性矩阵。
2) 求度矩阵D,矩阵D是相似矩阵P的度矩阵。
3) 求拉普拉斯矩阵L,其中
。
4) 求出L的最小的k个特征值和对应的特征向量V。
5) 将特征向量V进行K-means聚类。
从而,n个病人被聚成了k个不同的类别。
3. 结果
评价聚类的有效性指标主要有两种。第一种是Davies Bouldin (DB) [12] 指标,DB指标主要描述病人的类内散度与各聚类中心的间距,定义为
, (10)
其中,
和
分别是第i类和第j类的中心点,
和
分别是第i类和第j类的类内数量。从DB指标可以看出DB越小表示类与类之间的相似性越低,从而对应最佳的聚类结果。
第二种是Calinski Harabasz (CH) [13] 指标,CH中类内离差矩阵描述紧密度,类间离差矩阵描述分离度,定义为
, (11)
其中,
是整个数据集的中心。从CH指标可以看出CH越大代表着类自身越紧密,类与类之间越分散,即聚类结果更好。
通过SNF、CSNF、NSNF和NSSNF方法,分别对5种癌症进行了相似网络融合,并计算了4种方法的聚类性能。详细结果见表2,表3。

Table 2. Detailed data for CH Value
表2. 每种癌症CH指标的详细数据

Table 3. Detailed data for DB Value
表3. 每种癌症DB指标的详细数据
对比四种SNF、NSSNF、NSNF和CSNF方法,数据表明NSSNF方法的DB值小于其它三种方法的DB值,NSSNF方法的CH值大于其它三种方法的CH值,因此本文改进的NSSNF方法聚类性能优于其它三种方法。具体对比结果见图1,图2。
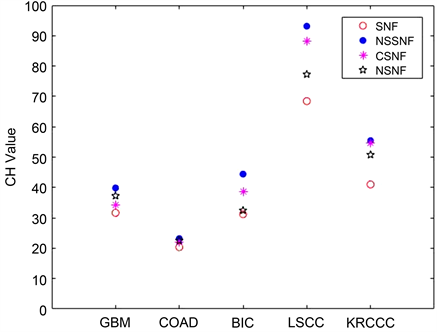
Figure 1. Comparison of CH values of five cancers data
图1. 五种癌症数据CH值的比较

Figure 2. Comparison of DB values of five cancers data
图2. 五种癌症数据DB值的比较
4. 结论
本文采用基于归一化欧氏距离的NSSNF方法,通过谱聚类改进邻居定义,构建患者相似度网络,将不同数据类型的相似性网络融合到一个相似性网络中,然后进行谱聚类。最后,通过DB值和CH值来评价谱聚类的有效性,数据分析结果均表明,NSSNF聚类方法明显优于SNF方法、CSNF方法和NSNF方法。
基金项目
国家自然科学基金项目(11271341)资助。
参考文献