摘要: 目的:采用生物信息学的方法探讨影响胃肠道间质瘤风险分级的差异基因表达及其生物学功能富集。方法:检索并获取GEO数据库中关于胃肠道间质瘤恶性程度差异基因表达的数据,利用GER2O工具对差异基因进行筛选和分析,采用DAVID数据库进行GO、KEGG富集分析,使用STRING,Cytoscape软件行蛋白–蛋白互作(PPI)网络构建。结果:筛选出两个数据集:GSE31802和GSE47911,查找出其中共有的差异表达基因有42个,它们的生物学过程可能主要富集在cAMP分解代谢过程、信号转导、基因表达的正向调控和酪氨酸激酶信号通路这几个方面。细胞学组分主要定位于质膜以及内质网腔。分子功能富集于信号素受体活性、序列特异性双链DNA结合。KEGG信号通路的富集分析显示差异基因主要富集于轴突的指导、癌症的转录失调等。对蛋白质相互作用分析中有关联的基因进行排序,显示IL-6,PGR的节点分数最高。结论:通过对影响胃肠道间质瘤风险分级的差异基因的分析,本研究发现的关键差异基因有助于了解影响胃肠道间质瘤危险分级的潜在因子。
Abstract:
Objective: To explore the differential gene expression and biological function enrichment affecting the risk classification of gastrointestinal stromal tumors by bioinformatics. Methods: The data of differential gene expression of malignant degree of gastrointestinal stromal tumors (GSE31802, GSE47911) in GEO database were retrieved and obtained. The differential genes were screened and analyzed by GER2O tool, GO and KEGG enrichment analysis were performed by DAVID database, and protein-protein interaction (PPI) network was constructed by STRING, Cytoscape software. Results: A total of 42 differentially expressed genes from the two data sets were screened, and their biologi-cal processes may be mainly concentrated in cAMP catabolism, signal transduction, positive regula-tion of gene expression and tyrosine kinase signal pathway; cellular components are mainly located in plasma membrane and endoplasmic reticulum lumen; molecular function is enriched in signal receptor activity and sequence-specific double-stranded DNA binding. The enrichment analysis of KEGG signal pathway showed that the differential genes were mainly enriched in the guidance of axons and transcriptional disorders in cancer. The sequencing of related genes in protein-protein interaction analysis showed that IL-6, PGR had the highest node score. Conclusion: Through the analysis of differential genes related to high-risk gastrointestinal stromal tumors, the key differen-tial genes found in this study are helpful to understand the potential factors affecting the grading of gastrointestinal stromal tumors.
1. 引言
胃肠道间质瘤(Gastrointestinal stromal tumor, GIST)是胃肠道最常见的恶性上皮下病变,它们起源于位于肌层内的Cajal间质细胞 [1] [2] 。胃肠道间质瘤主要由酪氨酸激酶受体KIT和/或血小板衍生生长因子受体-α (PDGFR-α)的致癌突变引起,大约10%到30%的胃肠道间质瘤有恶性的临床病程 [1] ,大约占所有胃肠道恶性肿瘤的1%~3%,最常见的是发生在胃(50%~60%)和小肠(30%~35%),也可发生于食管、结直肠,有文献报道肠系膜、网膜等也可发生 [3] [4] ,但不能完全排除是转移灶 [5] 。GIST具有从良性小病变到致命肉瘤的恶性潜能 [6] ,因此对该疾病进行科学的风险分级具有十分重要的意义。修改后的美国国立卫生研究院(National Institutes of Health, NIH)标准是评估胃肠道间质瘤潜在恶性风险的最常用工具,根据肿瘤的位置、大小、有丝分裂和肿瘤是否破裂,修改后的NIH标准将GIST分为极低风险、低风险、中风险和高风险 [7] 。往往中高危GIST患者手术切除后肿瘤转移和复发的风险显著增加,一项基于人群队列的综合分析构建了修改后的NIH标准的受试者工作曲线,曲线下面积为0.78,95%CI为0.75~0.80 [8] 。但即使同是高危侵袭性患者,其预后情况仍不尽相同,所以单纯采用NIH分级标准评估GIST的恶性潜能并不完全准确 [9] 。
随着生物信息技术的发展,微阵列基因表达分析的方法以及生物信息学方法已被应用于获得基因表达数据,并可在GEO (Gene Expression Omnibus)数据库中下载,本研究中,我们在GEO数据库中下载了两个微阵列数据集,探究影响胃肠道间质瘤风险分级的差异表达基因,并对其进一步分析。
2. 资料与方法
2.1. 数据获取
在GEO数据库(http://www.ncbi.nlm.nih.gov/geo) 检索公开发表的基因芯片数据集,检索条件:1) 检索词为“gastrointestinal stromal tumor”;2) 物种为“Homo sapiens”。最终选取了GSE31802,GSE47911两个数据集,分为高风险组和非高风险组。
2.2. 差异基因的识别
利用GEO数据库提供的在线分析工具GEO2R下载差异表达的基因数据,以差异倍数|logFC| > 2,p < 0.05作为差异基因的筛选条件,并利用在线工具Draw Venn Diagram (https://bioinformatics.psb.ugent.be/webtools/Venn)绘制维恩图,识别两个数据集共有的差异性表达基因。
2.3. 功能富集分析
利用注释可视化和集成发现数据库(Database for Annotation Visualization and Integrated Discovery, DAVID) (https://david.ncifcrf.gov/)对两个数据集共有的差异性表达基因进行基因本体论(Gene Ontology, GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)的功能富集分析。GO分析包括细胞成分(cellular component, CC)、生物过程(biological process, BP)和分子功能(molecular function, MF)。
2.4. 蛋白质相互作用分析
将筛选的两个数据集共有的差异性表达基因信息输入STRING数据库(http://string-db.org/),评估它们之间的功能关联,再用Cytoscape软件(version 3.9.1)分析这些基因的关联程度。
3. 结果
3.1. 胃肠道间质瘤高风险组和非高风险组中差异表达基因的筛选
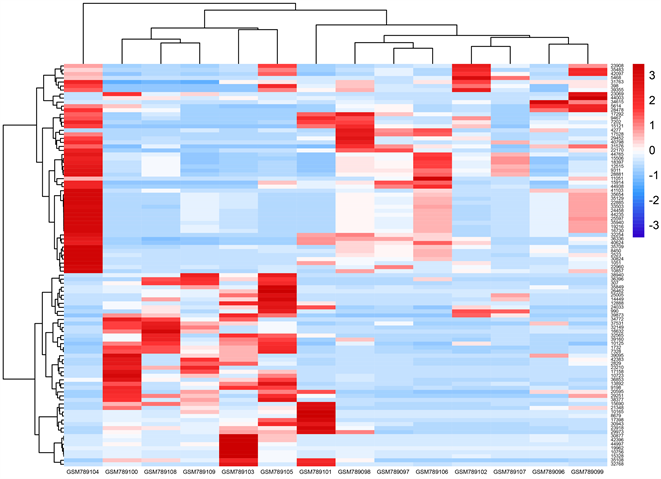
我们检索到了GEO数据库中关于胃肠道间质瘤恶性程度的差异基因表达数据集:GSE31802和GSE47911,利用GEO2R分别筛选出这两个数据集中差异性排名前100的基因及其相关数据,绘制热图如图1所示。进一步分析到两个数据集之间重叠的差异性表达基因有42个,如图2所示。
3.2. GO富集分析与KEGG信号通路富集分析
通过GO分析,两个数据集共有的差异表达基因的生物学过程可能主要富集在以下几个方面:cAMP分解代谢过程,信号转导,基因表达的正向调控,酪氨酸激酶信号通路。细胞学组分主要定位于质膜以及内质网腔。分子功能富集于信号素受体活性,序列特异性双链DNA结合,如图3所示。KEGG信号通路的富集分析显示:差异基因主要富集于轴突的指导,癌症中的转录失调等,如图4所示。
3.3. 蛋白质相互作用分析
将重叠的差异表达基因信息输入STRING评估他们之间的功能关联,再用Cytoscape软件分析这些基因的关联情况,并将关联程度进行等级排序,结果显示IL-6,PGR的节点分数最高,如图5所示。
 (a) GSE31802
(a) GSE31802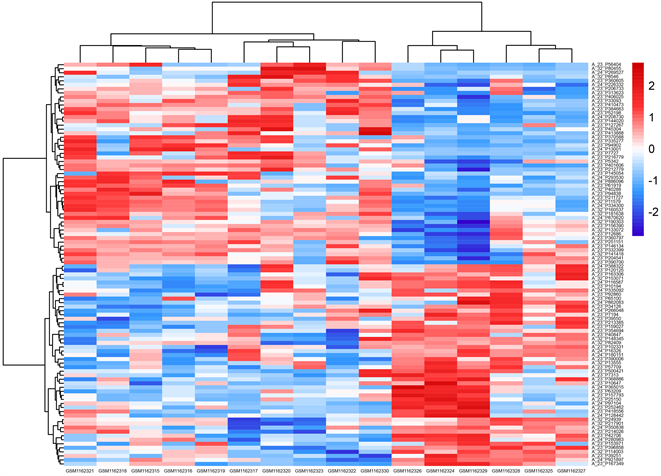 (b) GSE47911
(b) GSE47911
Figure 1. Differential gene expression Heat map of GSE31802, GSE47911 dataset
图1. GSE31802,GSE47911数据集差异基因表达热图
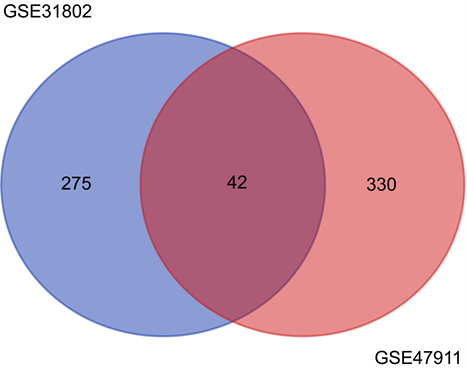
Figure 2. Venn diagram revealing the differentially expressed genes
图2. 两组芯片数据差异表达基因韦恩图
Figure 3. GO analysis of the differentially expressed genes
图3. 差异性表达基因的GO分析
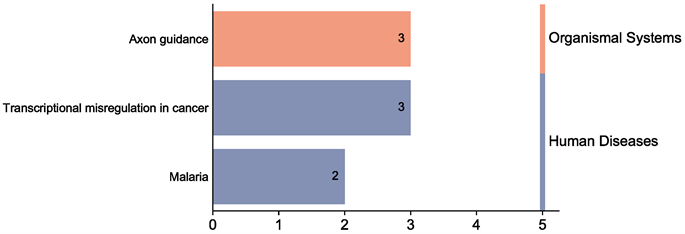
Figure 4. Enrichment analysis of KEGG signaling pathway of the differentially expressed genes
图4. 差异性表达基因的KEGG信号通路富集分析
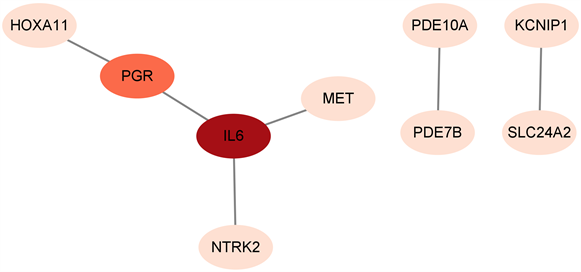
Figure 5. PPI network diagram of shared differentially expressed genes
图5. 差异性表达基因的蛋白质相互作用
4. 讨论
在本研究中,我们以GEO数据库为基础,检索到了2个关于胃肠道间质瘤恶性程度的差异基因表达数据(GSE31802和GSE47911),分析出这两个数据集之间重叠的差异性表达基因有42个。这42个共有的差异基因的生物学过程可能主要富集在cAMP分解代谢过程,信号转导,基因表达的正向调控和酪氨酸激酶信号通路这几个方面。细胞学组分主要定位于质膜以及内质网腔。分子功能富集于信号素受体活性、序列特异性双链DNA结合。KEGG信号通路的富集分析显示差异基因主要富集于轴突的指导、癌症中的转录失调等。我们采用Cytoscape软件进一步分析差异基因的功能关联,并进行了排序,其中IL-6,PGR的节点分数最高,它们可能在胃肠道间质瘤的风险分级中起到了一定的调控作用。
近年来,细胞因子相互作用的生化和免疫网络受到了极大的关注,这些网络有助于促进这些肿瘤的进展和转移扩散 [10] 。巨噬细胞来源的细胞因子IL-6、TNF-a、IL-1b和干扰素-g在炎症的诱导和放大中起着关键作用,Silke Cameron等人研究发现,在GIST中,急性期细胞因子RNA表达很低,尤其是TNF-α和IL-6 [11] 。孕激素受体(progesterone receptor, PGR)通过配体依赖和非配体依赖的激活来调节细胞的生长和分化,在乳腺癌和妇科肿瘤中,它们的表达是内分泌治疗益处和良好的非治疗预后的已知预测因子 [12] 。但在软组织肉瘤中,它们的表达谱不明确,其意义也不确定。本研究提供了一些可能性,但这些基因是否与胃肠道间质瘤的风险程度相关,在其中的作用机制如何,则有赖于更进一步的研究。
5. 结论
胃肠道间质瘤是消化系统最常见的间叶肿瘤,可涉及整个胃肠道,多见于胃和小肠。在治疗手段上,主要是开腹、腹腔镜、内镜切除等手术方式,再结合分子靶向药物进行辅助治疗。尽管当前治疗方法取得了较好效果,但部分患者依然会出现复发或转移。因此明确影响患者危险分级的因素有助于更好地评估胃肠道间质瘤患者,为诊断和治疗提供参考。
NOTES
*通讯作者。