1. 引言
病毒诱导基因沉默(virus induced gene silencing, VIGS)技术是一种RNA介导的抗病毒防御反应机制,目前在植物反向遗传学领域已经表现出巨大的潜力。与其它导致基因功能缺失的研究方法(如反义抑制、基因突变等)相比,VIGS技术不仅优于传统的植物转基因技术,方法简便,高效耐用,而且具有高通量特性 [1] 。而且病毒诱导的基因沉默具有研究周期短、不需要遗传转化、可在不同的遗传背景下生效以及能在不同的物种间进行基因功能的快速比较等优点,在功能基因组学领域的研究中,这些优越性已经使VIGS技术成为最具吸引力的首选技术手段。在VIGS中,通常以八氢番茄红素去饱和酶(PDS)基因作为报告基因,探索病毒载体在不同种植物上应用的可能性。最早Kumagai等 [2] 将八氢番茄红素脱氢酶(PDS)克隆到烟草花叶病毒(TMV)中,浸染烟草后植株表现白化表型,说明内源的PDS基因被沉默掉了,因为已知PDS参与类胡萝卜素类物质的合成,而缺乏类胡萝卜素的植物组织将产生白化症状。PDS抑制表型容易观察,因此PDS基因成为VIGS体系评价的参照基因。最近已经有报道在小麦中利用VIGS (BSMV-VIGS)技术沉默六倍体小麦基因的研究 [3] ,在研究植物进化方向也有应用VIGS的报道,Oriane Hidalgo [4] 利用VIGS技术研究罂粟家族的多元进化史。这些载体的开发和利用为单子叶植物的功能基因组研究提供了有效的工具。烟草脆裂病毒(TRV)是目前应用最广泛的VIGS载体,它便于外源序列的插入和随后对植物的浸染,具有病毒感染症状轻、沉默效率高等优点,在拟南芥、烟草、番茄等多种植物上得到成功应用 [5] [6] 。
随着分子生物学研究的进展,几乎所有重要的基因或者基因片段都已经被克隆,对其功能也有了一些了解。但是,不同的基因的互作研究,特别是在小经济作物中还只是刚刚开始起步。这些分子生物学研究尚未很好的与植物品质研究和改良结合起来,对品质育种的指导意义还是十分有限,魔芋中更是如此,此技术的应用有望解决魔芋中品质改良因周期长而效率低下的育种瓶颈。本文根据已报道的烟草中PDS基因序列,克隆得到魔芋中其部分基因PDS片段命名为NbPDS。构建病毒载体(TRV)体系并利用携带目的基因的TRV重组载体病毒感染魔芋幼苗叶片,分析并鉴定魔芋PDS基因的功能。
2. 材料与方法
2.1. 实验材料
本实验的研究材料为魔芋(Amorphophallus konjac)组培幼苗,TRV载体p TRV1、p TRV2及农杆菌GC3103实验室保存。
2.2. 实验方法
2.2.1. 目的基因的克隆
依据RNA试剂盒(天根)的方法提取魔芋总的RNA,再依据反转录试剂盒(天根)合成cDNA第一链。根据已报道的NCBI数据库中本氏烟(Nicotiana benthamiana)的烟草八氢番茄红素脱氢酶PDS基因序列(登录号ABE99707),选取部分编码区片段为目标片段,设计上游引物F1(5-CGATCCCGGATAGGGTGACAGATGA-3',下画线为BamH I酶切位点)和下游引物R1(5'-CGGAATTCACACACTGAGCAGCGCAACT-3',下画线为EcoR I酶切位点),引物由上海生工公司合成。以cDNA为模板,进行OCR扩增,扩增条件:PCR反应体系为E × Taq 0.2 μL、10 × Taq buffer 2 μL、Mg2+ (25 mmol/L) 2 μL、dNTP (2.5 mmol/L) 2 μL、DFR-1F/DFR-2F (10 μmol/L) 0.5 μL、DFR-1R/DFR-2R (10 μmol/L) 0.5 μL、cDNA 模版2 μL、ddH2O 15.8 μL,总体积为25 μL。PCR反应条件:预变性94℃ 3 min,94℃变性30 s,58℃退火30 s,72℃延伸30 s,30个循环,最后72℃延伸5 min。PCR扩增产物经1.0%琼脂糖凝胶电泳检测后回收和纯化。
2.2.2. VIGS载体的构建
通过PCR反应扩增得到目的基因PDS片段。将此片段根据pMD19-T载体试剂盒(TaKaRa)操作说明书将目的基因片段PDS与T载体连接一起,通过蓝白斑筛选得到阳性克隆后送华大基因公司测序确认片段序列。将含有正确插入片段的T载体用BamH I和EcoR I对pMD19-T载体进行酶切,与经BamHI和EcoR I双酶切的p TRV2载体通过T4-DNA连接酶连接, 转化大肠杆菌,经PCR以及用双酶切鉴定重组质粒,将其命名为p TRV2-DFR。然后通过冻融法转化农杆菌菌株GV3101,阳性克隆筛选,保存菌种待用。
2.2.3. VIGS浸染实验
VIGS浸染实验参考侯巧明等 [7] 的VIGS部分的实验方法进行:略有改动,将质粒载体和空的质粒载体以及带有目的基因的农杆菌菌落取样l mL接种到20 ml LB选择培养液中,37℃水浴振荡培养5 h后,置于28℃下继续过夜震荡培养。5000 rpm离心10 min收集农杆菌,上清液倒掉,再加入悬浮液直到其吸光度OD 600值为1.5左右,不同之处在于静置时间根据情况改为4 h左右,将等体积的质粒农杆菌GV3101悬浮液和p带有目的基因的质粒悬浮液混合均匀后,再置于28℃下震荡2 h,然后开始注射实验材料魔芋叶片。浸染后培养的幼苗正常管理,大约20 d可以观察到基因沉默导致的表型变化(图1)。
 (a)
(a) (b)
(b)
Figure 1. Phenotypic change of plant. (a) Plants after virus immersion; (b) Control
图1. 植株表型变化。(a) 病毒浸染后的植株;(b) 对照
2.2.4. 基因表达检测
选取被农杆菌侵染20 d的魔芋叶片提取总RNA,提取RNA的方法以及合成cDNA的方法参考1.2.1。使用实时荧光定量PCR检测DFR基因沉默的效果。内参基因Actin (登录号:DN551593;F:5'-ggaaaagtgcagagagacacg-3', R:5'-tacagtgtctggatcggtggt-3', 150bp)。实时荧光定量PCR检测引物DFR(登录号 GU324979,DFR-F:5'-atgtctgttgtacatgagaga-3' DFR-R:5'-acgctagagatacgcactcaa-3')。实时荧光定量PCR反应采用天根(TIANGEN)试剂(RealMasterMix (SYBR Green)),每个反应重复3次,反应体系20 μl,含cDNA模板1. 6 μl,2. 5 × RealMasterMix 8 μl,20 × SYBR solution1 μl,Primer F 0. 4 μl,Primer R 0. 4 μl,ddH2O 8. 6 μl。荧光定量PCR仪(Smart CyclerII型,USA)上完成测定。扩增程序50℃温育2 min,(94℃,1 min→退火温度,30 s) × 1;(94℃,10 s→60℃,30 s) × 40。并按PfafflM W [8] 的方法计算基因的相对表达量。
3. 结果与分析
3.1. 八氢番茄红素脱氢酶PDS基因的克隆
从魔芋叶片中利用PCR扩增得到PDS部分基因序列,1%的琼脂糖检测大约在453 bp出有扩增出目的条带(图2),将该扩增片段利用回收试剂盒(天根)回收并连接到pMDl9-T载体种并测序。测序结果表明和目的基因片段大小一致450 bp,与已知的烟草PDS序列(登录号ABE99707)同源性到达77%。
3.2. 重组病毒载体的构建及转化农杆菌
将成功构建的重组质粒pTRV2-PDS转化大肠杆菌top 10,提取质粒pDNA经PCR鉴定为阳性,将质粒置于37℃水浴中经双酶切。酶切结果表明条带大小与目的基因片段一致,约453 bp左右(图3),说明pTRV2-PDS构建成功。将成功构建的病毒载体pTRV2-PDS转化农杆菌GV3101,挑取阳性克隆提取质粒并送华大基因公司测序,结果与已知的基因序列一致,可以用于浸染植株叶片。
3.3. 基因沉默结果及功能鉴定
采用叶片注射法浸染魔芋叶片,接种病毒20 d后观察发现,浸染重组病毒p TRV2-PDS的植株新长出的叶片出现白化现象,而且出现整棵植株叶片发育变慢,而对照植株叶片没有此现象(见图1)。株型变化推测成因可能是诱导的结果,取浸染20 d的植株叶片进行实时荧光定量PCR技术检测,发现PDS基因表达确实出现下调现象(图4),VIGS病毒诱导载体是否可以成功的沉默魔芋中的PDS基因还需进一步的实验验证。

Figure 2. PCR products. M. DL2000; 1~4. PCR products
图2. PCR扩增产物。M. DL2000; 1~4. PCR产物

Figure 3. Restrictive enzymes digestion. M. DL2000; 1. Enzymes digestion products
图3. 酶切图谱。M. DL2000; 1. 酶切产物
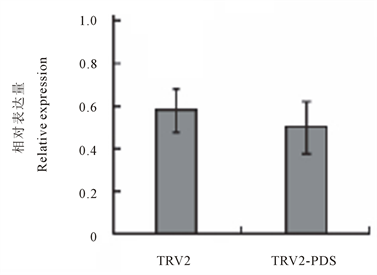
Figure 4. q RT-PCR analysis of PDS expression in the leaves. TRV2, plants infiltrated with p TRV2 as a control; p TRV2-PDS, plants infiltrated with p TRV2-PDS
图4. 叶片中PDS 基因表达的q RT-PCR分析。图中的TRV2表示浸染TRV2的空白对照植株,p TRV2-PDS 表示浸染的植株
4. 讨论
本研究利用PCR方法从魔芋中克隆了PDS基因的部分序列, 该基因片段与NCBI数据库中本氏烟(Nicotiana benthamiana)的烟草八氢番茄红素脱氢酶PDS基因序列(登录号ABE99707)同源性为77%,原因可能是由于品种间存在多态性基因导致的 [9] ,这在吕山花的研究中也有所表现。将PCR得到的PDS基因片段连接入pTRV2载体, 并转化入农杆菌GV3103中,本实验成功的构建了魔芋的VIGS体系,但是我们在用构建好的VIGS菌液(pTRV2-PDS和pTRV1载体的农杆菌)浸染不同植物, 发现在天南星科植物魔芋中表现不佳。这可能是本实验中构建的VIGS体系具有种的差异性。这与孙威等 [10] 观点一致,尽管VIGS载体的应用一直是依赖于植物种的,但是近年来,众多新载体尤其是通用载体的研发 [10] ,必将不断地拓宽VIGS的应用范围。
本实验还发现采用农杆菌叶片注射法浸染魔芋叶片,接种病毒20 d植株新长出的叶片出现植株叶片白化,生长缓慢现象,而对照植株叶片没有此现象。侯巧明等 [7] 在病毒诱导的基因沉默技术实验教学中指出:待注射农杆菌液的浓度,OD600值为1.0左右是比较合适的,太高则病毒毒性太大,植株病毒感染症状重,太低则达不到诱导效果。而本文中所用OD600值为1.5,可能是浓度太高所致。
本研究使用VIGS法,使魔芋PDS基因的表达下调,通过qRT-PCR法检测到DFR基因表达出现下调且基因表达下调了10%,该基因下调后,植株表型发生变化。但是下调未达到显著性变化,VIGS病毒诱导载体是否可以成功的沉默魔芋中的PDS基因还需进一步的实验验证。
5. 结论
本实验构建的VIGS病毒诱导体系可应用于魔芋基因功能的分析,根据前人研究结果VIGS在逆境抗性相关基因中的应用 [11] [12] ,结合本实验结果可以推测其在魔芋逆境胁迫研究中也具有一定的积极意义。
基金项目
贵州省科学技术厅项目:黔科合J字[2013]2246号;黔高教发[2017]158号:贵州省2017年一流大学重点建设项目师资团队建设子项目;贵州师范学院自然科学研究基金(2015BS005)。
NOTES
*通讯作者。