1. 引言
克氏原螯虾(Procambarus clarkii)俗称小龙虾,是我国重要的淡水经济养殖虾品种,具有良好的环境适应性和繁殖力强等突出优点 [1]。然而由于养殖环境的恶化和水产病害等因素的影响,克氏原螯虾养殖业的发展受到一定程度的制约并导致可观的经济损失。因此,研究克氏原螯虾的免疫反应机制对于提高虾个体的免疫能力及抗病品种的选育等方面具有重要意义。
已有研究表明肿瘤抑制因子在人类癌症的发病机理中起到了关键作用 [2],而TUSC3 (Tumor suppressor candidate 3)是子宫癌、前列腺癌以及胰腺癌的候选抑癌基因 [3] [4] [5] [6],TUSC3基因的缺失会导致癌细胞数量增加 [7] [8]。虽然肿瘤抑制因子在高等脊椎动物的功能已经得到了广泛研究,但该基因在甲壳类动物中的功能有待于进一步研究。
本实验旨在以克氏原螯虾为材料,克隆鉴定免疫相关基因TUSC3并采用荧光定量PCR技术研究该基因的组织表达特征和免疫应答模式,为今后进一步探讨克氏原螯虾的先天免疫机制及增强虾的抗病能力提供参考依据。
2. 材料与方法
2.1. 实验材料
克氏原螯虾(体长6.0 ± 1.0 cm,体重9.0 ± 1.0 g)购于合肥市琥珀市场,于室温下循环水系统中采用商业化饲料等暂养一周后再挑取具有活力且健康的个体进行实验 [9]。所选个体随机分为三组,每组21只。第一组注射20 μg LPS (细菌脂多糖);第二组注射20 μg poly I:C (双链RNA类似物);第三组为对照组,注射同体积的PBS。注射方式采用微量注射器进行腹部注射,注射剂量参考前期研究 [10] 并加以调整。注射3 h、6 h、9 h、12 h、24 h、48 h、72 h后,分别取血淋巴细胞、肠和鳃组织并提取总RNA用于实验分析。
2.2. cDNA模板的制备
取100 mg组织样品放入预冷的研钵中,加入液氮并迅速研磨成粉状,利用Trizol (Takara)法提取组织总RNA。随后使用1%琼脂糖凝胶电泳检测RNA的完整性并通过核酸微量定量仪(Nanodrop 2000 Thermo Scientific,美国)检测其浓度和纯度。最后使用PrimeScriptTMRT Master Mix (Takara)试剂盒合成cDNA [11]。
2.3. TUSC3序列的解析
根据课题组之前获得的克氏原螯虾转录组信息 [9],利用DNASTAR软件设计特异性的TUSC3扩增引物(TUSC3-F1, TCTTGTGGACTTTGATGAGG; TUSC3-R1, GAGCATAGCAACAATGTAGGT),PCR采用20 μl反应体系,扩增程序如下:95℃预变性5 min;30个循环(95℃变性30 s,55℃退火30 s,72℃延伸40 s); 72℃延伸5 min。PCR产物经纯化后送北京华大基因科技有限公司测序。测序结果采用Blast (https://blast.ncbi.nlm.nih.gov)和Clustal X [12] 程序进行比对分析。
2.4. 实时荧光定量PCR
运用Primer premier 5.0设计定量引物(TUSC3-F2, CAGATTCGTGTGTTCCGTCC; TUSC3-R2, GGCTTGGGTTTTCTGCAGAA),同时选取18S rRNA基因(18S-F, CTGTGATGCCCTTAGATGTT; 18S-R, GCGAGGGGTAGAACATCCAA)作为内参。使用SYBR Premix Ex TaqTM (TaKaRa)试剂盒进行荧光定量PCR操作 [9],每个实验设置3个重复,采用2−ΔΔCT法计算基因的相对表达量 [13]。数据处理采用学生t测验或单因素ANOVA检验方法,数值以平均值±标准差表示,当p ≤ 0.05时为差异显著。
3. 实验结果
3.1. TUSC3序列分析
克氏原螯虾TUSC3基因含有一个981 bp的ORF框,编码326个氨基酸。预测的蛋白大小为36.76 kDa,等电点为6.58,包含一个与OST3/OST6同源的结构功能域(31-321aa)。该TUSC3与其他无脊椎动物的TUSC3蛋白序列比对结果表明,克氏原螯虾TUSC3与南美白对虾的TUSC3同源性最高,达93.25%,其次为美洲钩虾(相似度76.56%),其中与其他昆虫的TUSC3蛋白序列相似度介于67%~69%之间(见图1)。这些结果表明,克氏原螯虾TUSC3在进化上具有一定的保守性。
3.2. TUSC3基因组织分布情况
取肠、鳃、心脏、肌肉、血细胞及肝胰腺组织,利用实时荧光定量PCR检测TUSC3基因的表达特征。TUSC3在各被检组织中均有表达,其中在血细胞、肠和肌肉中的表达水平较高,而在心脏和腮中的表达量较低(见图2)。
3.3. LPS刺激对TUSC3基因表达水平的影响
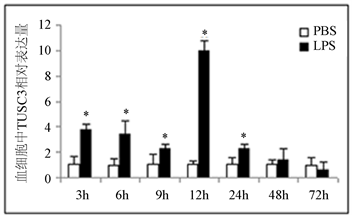
为了研究TUSC3在克氏原螯虾免疫刺激后的表达模式变化,本实验采用LPS处理克氏原螯虾,并以PBS处理对照组。结果表明(见图3),LPS刺激后3 h至24 h血细胞中TUSC3的mRNA表达水平显著上调,其中12 h时最高(增加近10倍)。肠中TUSC3的转录水平在3 h至6 h时相对于对照无明显变化,但9 h至72 h内显著上调。其中9 h达到最高(增加约24倍),随后表达量逐渐下降。在鳃中,9 h至72 h内TUSC3的表达水平明显上调,其中24 h达到最高(增加约19倍),但在3 h至6 h时无明显变化,与肠中的表达特点相似。以上数据都说明了TUSC3可能参与克氏原螯虾的免疫反应。

Figure 1. Sequence alignment of P. clarkii TUSC3 with other TUSC3 proteins. Identical amino acids were indicated by black and gray represented similar amino acids
图1. 克氏原螯虾TUSC3蛋白序列的同源比对图。相同的氨基酸用黑色表示,相似的氨基酸用灰色表示

Figure 2. Expression level of P. clarkii TUSC3 in various tissues. Bars labeled with different letters are statistically significantly different (p < 0.05)
图2. 克氏原螯虾TUSC3的组织表达情况图。不同字母表示不同组织中的表达量存在显著差异(p < 0.05)
3.4. Poly I:C刺激对TUSC3表达水平的影响
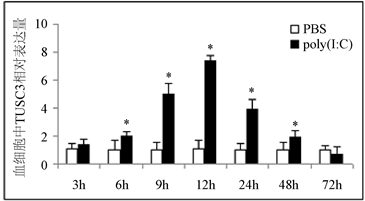
Poly I:C刺激后6 h至48内血细胞中的TUSC3表达水平先升高再下降的趋势,但均高于对照组,其中12 h时达最高。在刺激后3 h至12 h内鳃中TUSC3的表达量明显增加,其中9 h达到最高(增加约20倍),但在24 h后明显下降。虽然肠中TUSC3表达在3 h时表现为上调,但其后发生了显著下降,直至72 h时与对照表达水平相近(见图4)。
 (a)
(a) 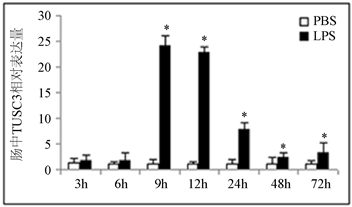 (b)
(b) (c)
(c)
Figure 3. Expression pattern of TUSC3 in hemocytes (a), gut (b) and gill (c) stimulated by LPS Asterisks. (*) represent significant differences between control and experimental groups
图3. LPS刺激后TUSC3在血淋巴细胞(a),肠(b)和腮(c)中的表达情况。星号(*)表示处理组与对照组相比存在差异显著
 (a)
(a)  (b)
(b) (c)
(c)
Figure 4. Expression of TUSC3 in hemocytes (a), gut (b) and gill (c) stimulated by poly(I:C). Asterisks (*) represent significant differences between the control and experimental groups
图4. Poly(I:C)刺激后TUSC3在血淋巴细胞(a),肠(b)和腮(c)中的表达情况。星号(*)表示处理组与对照组相比存在差异显著
4. 讨论
TUSC基因参与了哺乳动物的细胞凋亡、细胞周期调控及各种蛋白质激酶活化等多种信号通路,然而关于其在无脊椎动物中的具体生物学功能研究相对较少。本实验以克氏原螯虾为材料,探讨了其在先天免疫中的功能。
克氏原螯虾TUSC3基因编码氨基酸序列与其他虾类和昆虫的TUSC3序列同源性较高,尤其与甲壳类动物中的对虾和钩虾,这与传统的分类学结果相一致。从TUSC3的表达模式上看,该基因在血淋巴细胞和肠免疫组织中的表达量相对较高,预示着该基因与克氏原螯虾的免疫应答过程有关。脂多糖(LPS)和聚肌胞苷酸(poly I:C)是重要的病原体模拟分子,被广发用于研究动物的免疫应答机理 [14]。除poly I:C处理后的肠组织外,处理后的其他组织中的TUSC3表达量均表现为上调趋势,其中以肠和腮的表达量变化较大。相对而言,LPS诱导的效果好于poly I:C,这可能与TUSC3参与的信号传导途径的差异有关。虽然TUSC3在克氏原螯虾先天免疫应答过程中具有功能,但其分子激活机制有待阐明。
基金项目
2017年省级大学生创新创业训练计划项目,安徽省高校自然科学研究项目[KJ2017A145]。
NOTES
*通讯作者。