摘要:
本实验通过PCR方法检测34株广西凡纳滨对虾源副溶血弧菌中喹诺酮类耐药基因qnrC、qnrS和qnrVC的分布情况。耐药基因检测结果表明,34株副溶血弧菌中qnrC、qnrS和qnrVC基因的检出率分别为2.9%、0.0%与2.9%。qnrC基因在防城港市、钦州市、北海市的检出率分别为16.7%、0%、0%。qnrS基因在防城港市、钦州市、北海市的检出率均为0%。qnrVC基因在防城港市、钦州市、北海市的检出率分别为0%、0%、6.3%,三种耐药基因的检出率在三个市的副溶血弧菌之间差异均不显著(P > 0.05)。基于qnrC基因的氨基酸序列同源性比较结果表明,防城港株F17383与副溶血弧菌TOE26912.1、TOP53441.1、OQU02262.1亲缘关系最近,氨基酸同源性分别为100.0%、98.8%和98.8%。基于qnrVC基因氨基酸序列同源性比较结果表明,北海株B13121与副溶血弧菌AXI69764.1亲缘关系最近,同源性为99.4%。
Abstract:
In this study, the distribution of quinolone resistance genes qnrC, qnrS, and qnrVC in 34 strains of Vibrio parahaemolyticus from shrimp in Guangxi was detected by PCR. The detection results of antibiotic resistance genes showed that the detection rates of qnrC, qnrS and qnrVC genes in 34 strains of V. parahaemolyticus are 2.9%, 0.0% and 2.9% respectively. The detection rates of qnrC gene in Fangchenggang City, Qinzhou City and Beihai City were 16.7%, 0% and 0%, respectively. The detection rates of qnrS gene in Fangchenggang City, Qinzhou City and Beihai City were all 0%. The detection rates of qnrVC gene in Fangchenggang City, Qinzhou City and Beihai City were 0%, 0% and 6.3%, respectively. The detection rates of the three antibiotic resistance genes were not significantly different among the strains of Vibrio parahaemolyticus of three cities (P > 0.05). Based on the homology comparison of the amino acid sequences of qnrC gene, the Fangchenggang strain F17383 had the closest genetic relationship with V. parahaemolyticus TOE26912.1, TOP53441.1 and OQU02262.1, sharing homology of 100.0%, 98.8% and 98.8%, respectively. And based on the homology comparison of the amino acid sequences of qnrVC gene, the Beihai strain B13121 had the closest genetic relationship with V. parahaemolyticus AXI69764.1, sharing homology of 99.4%.
ABSTRACT
In this study, the distribution of quinolone resistance genes qnrC, qnrS, and qnrVC in 34 strains of Vibrio parahaemolyticus from shrimp in Guangxi was detected by PCR. The detection results of antibiotic resistance genes showed that the detection rates of qnrC, qnrS and qnrVC genes in 34 strains of V. parahaemolyticus are 2.9%, 0.0% and 2.9% respectively. The detection rates of qnrC gene in Fangchenggang City, Qinzhou City and Beihai City were 16.7%, 0% and 0%, respectively. The detection rates of qnrS gene in Fangchenggang City, Qinzhou City and Beihai City were all 0%. The detection rates of qnrVC gene in Fangchenggang City, Qinzhou City and Beihai City were 0%, 0% and 6.3%, respectively. The detection rates of the three antibiotic resistance genes were not significantly different among the strains of Vibrio parahaemolyticus of three cities (P > 0.05). Based on the homology comparison of the amino acid sequences of qnrC gene, the Fangchenggang strain F17383 had the closest genetic relationship with V. parahaemolyticus TOE26912.1, TOP53441.1 and OQU02262.1, sharing homology of 100.0%, 98.8% and 98.8%, respectively. And based on the homology comparison of the amino acid sequences of qnrVC gene, the Beihai strain B13121 had the closest genetic relationship with V. parahaemolyticus AXI69764.1, sharing homology of 99.4%.
Keywords:Litopenaeus vannamei, Vibrio parahaemolyticus, qnrC Gene, qnrS Gene, qnrVC Gene

Copyright © 2020 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
凡纳滨对虾(Litopenaeus vannamei)具有营养价值高、肉质鲜美、生长快以及对盐度和温度的适应性较强等特点,是全球养殖产量最高的对虾品种之一 [1]。然而近年来,副溶血弧菌(Vibrio parahaemolyticus)引起的病害问题已成为影响凡纳滨对虾养殖成功的重要因素。因此,深入研究副溶血弧菌的生物学特性,寻找控制副溶血弧菌病的办法尤为重要 [2]。目前,副溶血弧菌病的常用控制措施是使用抗生素或消毒剂。喹诺酮类药物是一种具有广谱杀菌作用的合成类抗生素 [3],其种类包括恩诺沙星、氧氟沙星、诺氟沙星等 [4]。该类抗生素的抑菌原理主要为通过抑制细菌的DNA促旋酶和DNA拓扑异构酶IV来阻止细菌DNA的合成 [5]。已有研究表明,细菌对喹诺酮类耐药性产生机制主要包括DNA促旋酶和DNA拓扑异构酶IV基因的突变、通过改变细胞膜来主动外排药物 [6] 以及质粒介导喹诺酮类耐药基因(PMQR)的参与 [7] 等。qnrC、qnrS、qnrVC均为PMQR基因,其编码蛋白可与DNA促旋酶及拓扑异构酶IV结合,从而保护喹诺酮类的作用靶位,使细菌形成对喹诺酮类的抗性,这些PMQR基因通常与整合子或插入序列相连、并与其他耐药基因一起在菌株间进行水平传播 [3]。目前,有关副溶血弧菌对喹诺酮类耐药分子机制的研究已有不少报道 [8] [9]。但是关于广西凡纳滨对虾源副溶血弧菌中qnrC、qnrS、qnrVC基因分布情况的相关研究仍为少见。本试验对34株广西凡纳滨对虾源副溶血弧菌进行三种喹诺酮类耐药基因qnrC、qnrS、qnrVC基因的检测和相关序列分析,其结果可为凡纳滨对虾副溶血弧菌病的科学防控提供指导,为副溶血弧菌的喹诺酮类耐药分子机制的深入研究提供基础数据。
2. 实验材料与方法
2.1. 实验材料
试验菌株为本实验室于2013年至2018年从广西防城港市、钦州市和北海市患病凡纳滨对虾中分离的34株副溶血弧菌(表1),在试验前均已进行API生化鉴定和16S rRNA分子鉴定以确认。细菌基因组DNA提取试剂盒购自北京康润诚业生物科技有限公司。2 × Taq MasterMix由北京艾德莱生物科技有限公司生产。

Table 1. Source information of 34 strains of Vibrio parahaemolyticus
表1. 34株副溶血弧菌来源信息
2.2. 细菌DNA的提取及耐药基因PCR鉴定
依照细菌基因组DNA提取试剂盒的说明书来进行细菌总DNA的提取。qnrC、qnrS和qnrVC的PCR反应引物分别参照Wu [10] 等、Kim [11] 等和Liu [12] 等的方法进行设计并由生工生物工程(上海)有限公司合成(表2)。通过PCR法检测3种耐药基因,其25 µL反应体系为:DNA模板2 µL,上、下游引物各1 µL,2 × Taq Master Mix 12.5 µL,灭菌去离子水8.5 µL。设置PCR反应程序为95℃预变性3 min;95℃ 30 s,退火30 s (退火温度见表2),72℃ 90 s,30个循环;72℃延伸10 min。以1.0%琼脂糖凝胶电泳鉴定PCR产物,在凝胶成像系统观察电泳结果。将符合预计片段长度的PCR产物送广州华大基因科技有限公司进行测序。

Table 2. Primer sequences of PCR reaction
表2. PCR反应引物序列
2.3. 序列分析
在美国国家生物技术信息中心(NCBI)网站搜寻不同细菌来源的耐药基因序列。使用DNAStar软件的Megalign模块计算耐药基因的氨基酸序列的同源性。采用ClustalX进行序列完全比对分析后采用Mega5软件的邻接法构建系统发育进化树,Bootstrap重复1000次检验其可信度。
2.4. 统计分析
使用SPSS18.0软件的χ2检验分析在防城港市、钦州市和北海市之间qnrC、qnrS和qnrVC基因检出率的差异性,当P < 0.05时表示差异显著。
3. 实验结果
3.1. 耐药基因的PCR检测结果
对34株副溶血弧菌进行喹诺酮类耐药基因qnrC、qnrS、qnrVC检测,结果表明部分受试菌株可扩增出大小约为308 bp的qnrC基因片段或521 bp的qnrVC基因片段(图1),与预计片段长度基本一致。将阳性PCR产物送测序,其测序结果经比对确认这些产物片段均为qnrC基因或qnrVC基因的序列片段。经反复PCR验证,所有菌株均未扩增出约为642 bp的qnrS基因片段。
 注:M为DL1000 DNA Marker;N为阴性对照;qnrC基因电泳图(A图)的1至9号分别为菌株B17137、B17139、B17143、B17144、F17383、F17386、F17387、F17388、Q17438的PCR扩增产物;qnrS基因电泳图(B图)的1至9号分别菌株B17139、B17143、B17144、F17383、F17386、F17387、F17388、Q17438、Q13030的PCR扩增产物;qnrVC基因电泳图(C图)的1至9号分别为菌株Q13017、Q13023、Q13030、Q13031、B13062、Q13079、Q13085、B13121、B13138的PCR扩增产物。
注:M为DL1000 DNA Marker;N为阴性对照;qnrC基因电泳图(A图)的1至9号分别为菌株B17137、B17139、B17143、B17144、F17383、F17386、F17387、F17388、Q17438的PCR扩增产物;qnrS基因电泳图(B图)的1至9号分别菌株B17139、B17143、B17144、F17383、F17386、F17387、F17388、Q17438、Q13030的PCR扩增产物;qnrVC基因电泳图(C图)的1至9号分别为菌株Q13017、Q13023、Q13030、Q13031、B13062、Q13079、Q13085、B13121、B13138的PCR扩增产物。
Figure 1. Homology comparison of the amino acid sequences of qnrC gene among different species
图1. qnrC、qnrS和qnrVC基因PCR扩增结果
3.2. 耐药基因的序列分析
qnrC基因的氨基酸序列同源性比较(图2)结果表明,防城港株F17383与副溶血弧菌广西株TOE26912.1、湖北株TOP53441.1、美国株OQU02262.1亲缘关系最近,氨基酸同源性分别为100.0%、98.8%和98.8%;与噬海藻弧菌V. algivorus (WP_144389023.1)、V. gangliei (WP_105901077.1)和奇异变形杆菌Proteus mirabilis (ACK75961.1)的氨基酸同源性为98.8%、97.6%、97.6%;与Moritella viscosa (CED58093.1)氨基酸同源性相对较低,为61.9%。

Figure 2. Homology comparison of the amino acid sequences of qnrC gene among different species
图2. 不同物种qnrC基因的氨基酸序列的同源性比较
进化树构建结果如图3所示,可见F17383与副溶血弧菌广西株TOE26912.1、湖北株TOP53441.1、美国株OQU02262.1聚为一支,在进化关系上相对较近;与Moritella viscosa (CED58093.1)在进化关系上相对较远。

Figure 3. Phylogenetic tree based on the amino acid sequences of qnrC gene
图3. 基于qnrC基因氨基酸序列构建的进化树
qnrVC基因氨基酸序列同源性比较(图4)结果表明,北海株B13121与副溶血弧菌香港株AXI69764.1亲缘关系最近,其同源性为99.4%;与副溶血弧菌深圳株AXA19787.1、藤江弧菌(V. fujianensis) WP_099610042.1、弗氏柠檬酸杆菌(Citrobacter freundii) AQT26619.1、鳗弧菌(V. anguillarum) TYC88856.1的氨基酸同源性在97.5%~98.7%之间;与副溶血弧菌深圳株ANS55787.1、肺炎克雷伯菌(Klebsiella pneumoniae) BCD58983.1、嗜水气单胞菌(Aeromonas hydrophila) AXV36521.1、创伤弧菌(V. vulnificus) WP_072606284.1、拟态弧菌(V. mimicus) TXZ06185.1、霍乱弧菌(V. cholerae) KAA1213539.1的同源性在84.9%~85.5%之间。
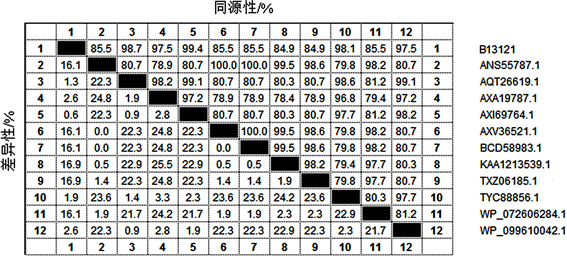
Figure 4. Homology comparison of the amino acid sequences of qnrVC gene among different species
图4. 不同物种qnrVC基因的氨基酸序列的同源性比较
进化树构建结果如图5所示,可见北海株B13121与副溶血弧菌香港株AXI69764.1、副溶血弧菌深圳株AXA19787.1、藤江弧菌WP_099610042.1、弗氏柠檬酸杆菌AQT26619.1、鳗弧菌TYC88856.1聚为一支,在进化关系上相对较近;与副溶血弧菌深圳株ANS55787.1、肺炎克雷伯菌BCD58983.1、嗜水气单胞菌AXV36521.1、创伤弧菌WP_072606284.1、拟态弧菌TXZ06185.1、霍乱弧菌KAA1213539.1等在进化关系上相对较远。
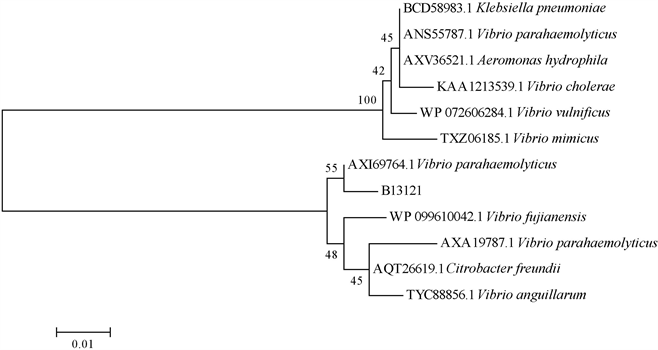
Figure 5. Phylogenetic tree based on the amino acid sequences of qnrVC gene
图5. 基于qnrVC基因氨基酸序列构建的进化树
3.3. 耐药基因在广西三个市菌株的检出率
34株副溶血弧菌中,qnrC、qnrS、qnrVC基因总检出率分别为2.9% (1/34)、0% (0/34)、2.9% (1/34)。三种耐药基因在三个市的分布情况如表3所示,qnrC基因在防城港市、钦州市、北海市的检出率分别为16.7% (1/6)、0% (0/12)、0% (0/16)。qnrS基因在防城港市、钦州市、北海市的检出率均为0%。qnrVC基因在防城港市、钦州市、北海市的检出率分别为0% (0/6)、0% (0/12)、6.3% (1/16)。比较分析结果表明,三种耐药基因的检出率在三个市的副溶血弧菌之间差异均不显著(P > 0.05)。

Table 3. Comparison of the detection rates of three resistance genes of Vibrio parahaemolyticus in three cities
表3. 三个市之间副溶血弧菌三种耐药基因检出率比较
注:括号内的数值为携带耐药基因的菌株数与受试菌株总数之比。“-”表示P值无法计算。
4. 讨论
副溶血弧菌为凡纳滨对虾养殖中的常见致病菌,在一定条件下会造成对虾红体、烂鳃、早期死亡综合症等病症 [13]。同时,副溶血弧菌也是人畜共患病原菌,可导致腹泻、呕吐等胃肠疾病。抗生素治疗目前被认为是治疗副溶血弧菌病的有效手段。已有研究表明,我国一些地区水产养殖源弧菌对部分喹诺酮类抗生素已产生耐药性,如广西凡纳滨对虾源副溶血弧菌对盐酸沙拉沙星、环丙沙星的耐药率分别为94.03%、4.48% [14];福建、海南、江苏和浙江等地的副溶血弧菌对恩诺沙星的耐药率在4.5%~18.5%之间 [15];上海、山东、江苏等地水产养殖源弧菌对环丙沙星、恩诺沙星和诺氟沙星的耐药率达16.7%~32.5% [16] 等,这些有关水产养殖源弧菌的喹诺酮类耐药性的报道给人们敲响了警钟。对副溶血弧菌进行喹诺酮类耐药基因调查,对于收集副溶血弧菌菌株的分子流行病学特征以及监测其喹诺酮类耐药性具有重要意义。
自1998年首次报道了一种质粒介导喹诺酮类耐药基因并将其命名为qnrA基因后,研究人员相继在许多不同的细菌上发现各种qnr基因 [17]。qnrC、qnrS、qnrVC等质粒介导喹诺酮类耐药基因的编码蛋白可以保护喹诺酮类的作用靶位,使细菌免受喹诺酮类抑制 [3]。本试验qnrC基因的氨基酸序列同源性分析结果表明,防城港株F17383与副溶血弧菌广西株TOE26912.1、湖北株TOP53441.1、美国株OQU02262.1的同源性在98.8%~100.0%之间,表明来自不同地区的几株副溶血弧菌菌株之间其qnrC基因相对比较保守。qnrVC基因的同源性分析和进化树构建结果表明,北海株B13121与副溶血弧菌香港株AXI69764.1、深圳株AXA19787.1聚为一支,同源性分别为99.4%和97.5%,但B13121与另一副溶血弧菌深圳株ANS55787.1位于进化树的不同分支上,其同源性仅为85.5%。由于qnrVC基因可以通过质粒以及插入序列、转座子、整合子等可移动遗传组件在不同细菌种群之间进行水平传播 [18],推测B13121、香港株AXI69764.1、深圳株AXA19787.1这3株菌株的qnrVC基因可能与深圳株ANS55787.1的qnrVC基因具有不同的来源,但具体来源为何仍需要进一步的探讨。
根据本试验结果,广西凡纳滨对虾源副溶血弧菌中喹诺酮类耐药基因qnrC、qnrS、qnrVC的总检出率分别为2.9%、0%、2.9%,三种基因的检出率在防城港市、钦州市、北海市菌株之间的差异均不显著(P > 0.05)。与其它水产养殖源弧菌相比,本试验受试菌株的qnrC检出率与娄阳(0%) [19]、周海波(5.6%) [20] 的检出率接近,qnrS检出率与娄阳(0%) [19]、李健(0%) [16]、周海波(0.9%) [20] 的研究结果接近,qnrVC检出率小于李健(25%) [16] 和刘旭(50%) [18] 的检出率、与周海波(2.8%) [20] 的检出率接近。可见,不同来源的弧菌之间qnrVC检出率差异性相对较大,造成该结果的原因可能与菌株所处的水环境含qnrVC基因浓度不同有关,水产养殖用水也可能被含有qnrVC基因的生活废水或畜禽养殖废水污染 [21]。据报道,与抗性基因碱基突变相比,质粒介导的qnr基因水平传播能力更强,且含有质粒介导喹诺酮类耐药基因的临床分离细菌往往还会携带诸如广谱β-内酰胺酶基因等其它抗生素类耐药基因 [3]。由于qnrC、qnrVC等质粒介导耐药基因的存在增加了广西凡纳滨对虾源副溶血弧菌对喹诺酮类抗生素产生耐药的可能性,为尽可能控制副溶血弧菌对喹诺酮类和β-内酰胺类等其他抗生素耐药状况的加剧,建议应继续加强对qnrC、qnrS、qnrVC等喹诺酮类耐药基因的监控,同时避免水产养殖用水受生活废水或畜禽养殖废水的污染。
5. 结论
34株广西凡纳滨对虾源副溶血弧菌中,qnrC、qnrS、qnrVC基因的检测率为2.9%、0%、2.9%,在三个市之间三种耐药基因的检出率没有显著差异。防城港株F17383的qnrC基因的氨基酸序列与副溶血弧菌TOE26912.1、TOP53441.1、OQU02262.1亲缘关系最近,北海株B13121的qnrVC基因的氨基酸序列与副溶血弧菌AXI69764.1亲缘关系最近。
基金项目
国家现代农业产业技术体系广西创新团队建设专项(nycytxgxcxtd-14-02),广西大学博士启动项目(XBZ160128),广西农业农村厅水产养殖病害测报项目(桂财预函[2019] 105号)。