1. 引言
新型冠状病毒的溯源问题仍然是一大挑战,已有研究团队在此方面进行了探索与研究。在发布2019-nCoV基因组后不久,石正丽研究团队 [1] 发布了一种蝙蝠冠状病毒RaTG13的全基因组,RaTG13与2019-nCoV在全基因组水平上有96%的同源性,这表明2019-nCoV很有可能来自蝙蝠。然而,由于这类蝙蝠与人类的直接接触非常罕见,类似于SARS和MERS,2019-nCoV似乎更有可能是从另一个中间宿主向人类的溢出,而不是直接来自蝙蝠。
此外,管轶等人 [2] 对华南地区马来亚穿山甲中2019-nCoV相关冠状病毒进行了鉴定,沈永义等人 [3] 对马来亚穿山甲中新冠类似病毒进行了分离与鉴定,陈金平 [4] 分析了穿山甲是否为新冠病毒的中间宿主,以及Matthew C. Wong等人 [5] 通过对冠状病毒进行重组,表明了新冠病毒或来源于穿山甲。张志刚等人 [6] 首次表述了2019-nCoV与穿山甲冠状病毒(Pangolin-CoV)、RaTG13、SARS以及MERS的关系,研究表明穿山甲和蝙蝠一样,是β类冠状病毒的天然宿主,认为除了蝙蝠和穿山甲之外,2019-nCoV还存在其它未知中间宿主。
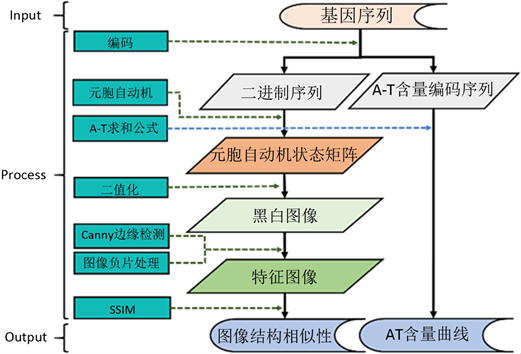
Figure 1. Data flow diagram of virus sequence visualization model
图1. 病毒序列可视化模型数据流程图
本文使用元胞自动机对病毒的基因序列进行可视化处理(图1),可以将2019-nCoV与非SARS相关病毒序列准确区分,并分析了序列图像特征形成的原因,推测出2019-nCoV的中间宿主。
2. 数据与方法
2.1. 数据集
从NCBI网站下载了属于冠状病毒的2019-nCoV的全基因组序列,同时为了研究2019-nCoV的潜在中间宿主,还下载了其他病毒的全基因组序列进行对比分析。具体的病毒序列及其相关信息见表1。

Table 1. List of virus sequence information of various species
表1. 各物种病毒序列信息表
2.2. 病毒序列可视化模型算法流程描述
元胞自动机 [7] (Cellular Automata, CA)与和一些传统的方法相比,其可以较为容易地模拟仿真物种演化、晶格生长、流体形成和化学反应过程等难以解析表达的复杂现象 [8]。
在元胞自动机中每个元胞的状态都受到其相邻元胞状态的影响。一维初等元胞自动机(ECA)是状态集中只有两个元素且邻居半径为1的一维元胞自动机。对于ECA,邻居个数为
,因此确定下一状态的映射函数如下所示:
(1)
F是元胞自动机的演化规则 [9],
表示元胞i在t + 1时刻的状态。图2显示了一维CA的演变。水平轴是空间,垂直轴是时间步长。行表示元胞空间在某一时间的整体状态,列表示同一个元胞在不同时间的状态。

Figure 2. Evolution of one-dimensional CA
图2. 一维CA的演变
公式(1)中演化规则含有中心元胞及其两个邻居,每个元胞都分别有两种状态,所以输入状态中一共有
种组合方式:
(2)
每一个输入条件都对应着两种输出状态0或1,一共存在
种状态组合。即一维初等元胞自动机总共存在256种演化规则。Wolfram在研究这些元胞自动机的时候对他们进行了标号,即将每种输入条件的输出0或1排列看成一个8位2进制数。
例如对于Wolfram的184号规则,其输出状态可表示为:
(3)
即184号规则的映射关系为:
(4)
2.2.1. 基因编码
为了降低元胞自动机的计算复杂度,本文将DNA的四种碱基编码为01数字信号,每一种碱基都由两位二进制的形式表示。腺嘌呤(A)、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T)分别编码为“00”、“10”、“01”和“11”。通过以上编码,DNA序列转化成数字序列,例如序列为“TTGCAGC”的基因片段由上述规则可以编码为“11111001001001”。这种编码方式具有可逆性,给定一个数字序列,也可以通过上述编码规则将其解码为DNA序列。
2.2.2. 元胞自动机状态矩阵生成
假设某DNA序列S的长度为N,编码后可以得到长度2N的数字序列,将此一维序列赋值为一维初等元胞自动机的初始元胞空间状态。在设置演化规则F、时间(演化次数) T后,本文采用循环边界条件对初始序列进行演化。
演化过程中,将各个元胞的状态存放在一个长度为2N,高度为T的二维矩阵Q,元胞状态的二维矩阵Q的映射关系如下所示:
(5)
(6)
(7)
表示第i行、第t列元素的值,
表示在t时刻所处位置为i的元胞的状态。
2.2.3. 图像生成
元胞自动机图通常使用不同的颜色表示各个元胞的状态,在元胞状态为0、1的元胞自动机图中,常常使用黑白两种颜色来表示,即£ = 0,¢ = 1。
在黑白图像中,灰度值为0表示白色,灰度值为255表示黑色。在将上述的元胞自动机状态矩阵Q可视化过程中,本文将该矩阵的元素进行二值化处理,从而使得生成的图像呈现出明显的纹理效果,具体公式如下所述:
(8)
通过上述操作可以生成一个尺寸为
的黑白图像。
为了缓解的计算机的运行压力,本文中将所有生成的黑白图像进行缩放,其缩放后的图片尺寸为10000 × 3000 (合计30,000,000个像素点)。
2.2.4. 图像特征处理
图像边缘是指某区域像素点的灰度值有阶跃变化或屋顶变化,其反映了图像中各个区域的灰度不连续性,边缘检测方法有助于突出图像的特征。Canny边缘检测是John Canny在1986年提出的图像边缘检测算法,本文使用该算法提取病毒序列可视化后的图像特征,并使用图像中最大灰度值的像素点作为阈值
,对所有的像素点进行负片处理:
(9)
L是待处理图片中最大灰度值的像素点,r是待处理图片中的像素点灰度值,
是已经过负片处理图像中像素点的灰度值。
如图3所示,图片3A为元胞自动机状态矩阵可视化的图像,图片3B为经过缩放后的图像,图片3C是经过Canny边缘检测后的特征图像,图片3D为特征图像经过负片处理后的特征图像。

Figure 3. Feature images of the virus sequence after various stages of processing
图3. 病毒序列经过各个阶段处理后的特征图像
2.3. 评估指标
2.3.1. SSIM指标
结构相似性(Structural Similarity, SSIM)是由Wang等人提出并用于衡量两个图像之间相似性的方法,通过比较两图像的亮度、对比度和结构来衡量图像之间的相似性。为了比较各种病毒元胞自动机图的相似性,本文使用SSIM值描述病毒序列特征图像之间的相似性。
2.3.2. 基因序列A-T含量统计
DNA序列中A与T之间含有2个氢键,C与G之间含有3个氢键,这些氢键在DNA结构的稳定性中发挥着关键的作用。在DNA单链中,序列内部A-T和C-G之间的连接也影响着单链的稳定性和单链的结构。统计A-T和C-G在序列各个位置中的分布,可以得出不同病毒之间的基因序列的异同。
本文主要对序列中各个位置的A-T含量分布进行研究,DNA序列的编码方式如下:
(10)
通过以上编码,DNA序列便可以用“0,1,-1”的序列表示。通过求和函数可以将DNA序列转换成二维空间的一条曲线:
(11)
其中
表示处于第i个位置的碱基的编码,L为序列P的长度。横坐标为序列碱基的位置,纵坐标表示各个位置的公式(11)的求和值,将各个位置所对应的值绘制成曲线,通过分析曲线的变化可以反映序列的特征。
3. 实验结果
本文应用以上的算法步骤,使用184号规则,演化次数为5000,将病毒的基因序列转化为二维的图像,然后分析和比对各个病毒序列之间的图像特征。
3.1. 病毒基因序列的特征图像特点
2019-nCoV、RaTG13和pangolin-CoV的特征图像,如图4、图5和图6所示,其余病毒序列的特征图像已上传至GitHub (https://github.com/MMCXXVI/Viral-sequence-visualization-model)。
通过对所有病毒的基因序列进行可视化处理,可以明显地发现所有冠状病毒的特征图像都有向左向右倾斜的条纹特征,但图片中向左倾斜“/”形条纹占大多数,且SARS相关病毒2019-nCoV、SARS-CoV

Figure 4. Sequence image of 2019-nCoV (MN908947)
图4. 2019-nCoV (MN908947)病毒序列特征图像
中都含有6个明显的“V”字形交叉区域,且大多分布在“ORF1a”、“ORF1b”、“S”这三个基因区域,其中2019-nCoV特征图像的“V”字形交叉区域映射在序列中的位置大约为1-2348bp、2394-7802bp、12251-14692、17610-18854、19787-20983bp、23878-25336bp。
在对蝙蝠RaTG13 (MN996532) [1] 和穿山甲pangolin-CoV (MT040336)病毒序列的进行可视化处理后(图5、图6),可以发现两者的特征图像都含有六个明显的“V”字形交叉区域,在与2019-nCoV的病毒序列特征图像进行比对后可以发现,三者的特征图像无论是在斜条纹的类型分布还是在“V”字形交叉区域的大小和位置分布上都极为相似。
同时,与冠状病毒特征图像有所不同的是,虽然埃博拉病毒的特征图像也有向左向右倾斜的条纹特征,但大多数为向右倾斜的“\”形条纹。

Figure 5. Sequence image of bat coronavirus RaTG13 (MN996532)
图5. 蝙蝠冠状病毒RaTG13 (MN996532)序列特征图像

Figure 6. Sequence image of pangolin-CoV (MT040336)
图6. 穿山甲冠状病毒pangolin-CoV (MT040336)序列特征图像
3.2. 特征图像相似性分析
使用SSIM算法对数据集中的各个病毒特征图像之间的相似性进行计算,同时将结果绘制成聚类热图,如图7所示。
在感染人类的8种病毒序列中,只有2019-nCoV和SARS之间的图像相似性达到了73.87%,其余病毒的之间的特征图像相似性均小于60%。
在分析感染人类的病毒和其他物种病毒之间的图像相似性后可以得知,与2019-nCoV特征图像相似性最高的病毒分别为蝙蝠冠状病毒RaTG13 (77.12%),穿山甲冠状病毒(73.36%),蝙蝠SARS类冠状病毒HKU3-2 (72.86%);与SARS特征图像相似性最高的病毒分别为蝙蝠SARS类冠状病毒HKU3-2 (74.97%),蝙蝠冠状病毒RaTG13 (74.22%),穿山甲冠状病毒(72.67%),其余病毒的之间的特征图像相似性均小于65%。同时,蝙蝠冠状病毒RaTG13、穿山甲冠状病毒与2019-nCoV之间的图像相似性均高于这两种冠状病毒与SARS-CoV之间的图像相似性。

Figure 7. Clustering heat map of similarity between feature images of various virus sequences
图7. 各个病毒序列特征图像之间相似性聚类热图
3.3. A-T含量曲线
通过1.3.2所描述的方法对数据集中的病毒序列进行编码,然后计算得出的A-T含量曲线如图8所示。
在图8A中,曲线主要分为三种类型,一种是以MERS病毒为代表的呈平滑上升趋势的曲线,另一种是以2019-nCoV为代表的呈凹凸状上升的曲线,还有一种是以Ebola病毒为代表的一直呈下降趋势的曲线。
为了分析2019-nCoV的潜在宿主,本文对类型为凹凸状上升的曲线进行分析,其A-T含量曲线图像如图8B所示,该类型的曲线包含有SARS-CoV、2019-nCoV、穿山甲冠状病毒、蝙蝠冠状病毒RaTG13和蝙蝠SARS类冠状病毒HKU3-2。
在图8B中,可以明显看出有6个凹形区域,且五种病毒的A-T曲线变化趋势大致相同。本文以2019-nCoV特征图像中六个“V”字形交叉区域与该曲线进行比对,发现A-T含量曲线中的凹形区域与2019-nCoV特征图像中六个“V”字形交叉区域高度重合。通过公式10可以得知,曲线中出现凹形说明该区域的前半段以A为主,而后半段以T为主,所以在特征图像中出现“V”字形交叉区域。
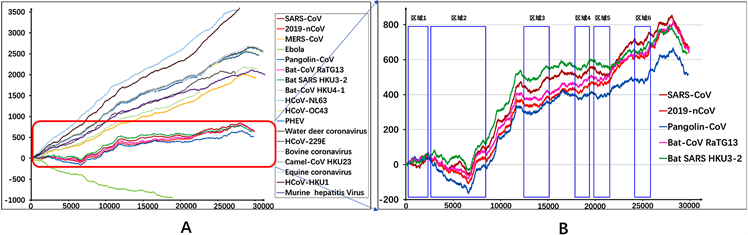
Figure 8. A: A-T content curve of each viral gene sequence; B: A-T content curve of ascending and descending; the blue box corresponds to the six “V” cross areas in the 2019-nCoV feature image; The X coordinate represents the position of base in the sequence (in bp), and the Y-coordinate represents the value calculated by Formula (11)
图8. A:各个病毒基因序列的A-T含量曲线;B:凹凸状上升的A-T含量曲线,蓝色方框对应着2019-nCoV特征图像中六个“V”字形交叉区域;横坐标代表碱基在序列中的位置(单位为bp),纵坐标代表由公式(11)计算出的值
由此可以得出,在某个序列片段中,如果A的含量大于T的含量,会导致该片段所对应特征图像出现向左倾斜的“/”形条纹,反之则出现向右倾斜的“\”形条纹。且基因序列的特征图像中若要出现“V”字形交叉区域,该区域所处的序列片段中,前半段多以A为主,后半段多以T为主,且T与A的比例接近于1:1。
同时,从图8可以看出,SARS病毒和蝙蝠SARS类冠状病毒曲线之间的变化趋势更为接近;而蝙蝠冠状病毒RaTG13、穿山甲冠状病毒的曲线与2019-nCoV更为接近。
4. 讨论与总结
本文从各个病毒序列的特征图像得出,蝙蝠冠状病毒RaTG13、穿山甲冠状病毒和2019-nCoV的图像极为相似,都存在6个V”字形交叉区域。再经过SSIM算法计算各个特征图像的相似性后得知,除SARS病毒之外,只有蝙蝠冠状病毒RaTG13、穿山甲冠状病毒的特征图像与2019-nCoV序列的图像相似性均高于70%。同时,通过A-T含量图可知,蝙蝠冠状病毒RaTG13、穿山甲冠状病毒的曲线与2019-nCoV更为接近。
综上,我们推测2019-nCoV很有可能来自蝙蝠,且穿山甲很可能是该病毒的中间宿主。虽然本章节所提出的方法并不能直接证明2019-nCoV的来源与中间宿主,但若是通过对“V”字形交叉区域的序列片段进行分析,进一步了解产生该现象的机理,这将会有助于新冠肺炎的差异化的治疗和防控。
虽然现在没有证据能完全确定2019-nCoV的是经由穿山甲传播,但不可否认的是,穿山甲有很大可能是2019-nCoV的中间宿主。要回答2019-nCoV起源与中间宿主这一科学问题,不仅需要在生物信息学、免疫学、分子流行病学和病毒传播模式学等各个方面进行分析,同时还需要生物学家使用病毒组学、反转录聚合酶链反应以及酶联免疫吸附测定等方法进行验证,才能得出确定的答案。
基金项目
国家自然科学基金项目(31860312);科技部政府间科技合作项目(国科外字[2018] 31号);江西省自然科学重点基金项目(20171ACB20023);景德镇市科技计划项目(20192GYZD008-04)。