1. 引言
乳腺癌逐渐成为女性的高发病症,位居于中国女性癌症发病的首位 [1]。三阴性乳腺癌(TNBC)是雌激素受体、孕激素受体和原癌基因Her-2的检测结果全都呈现为阴性的一种乳腺癌,在乳腺癌的所有分类中占据10.0%~20.8% [2] [3]。其病原体在病患体内有着高度扩散性和浸染性,容易在内脏间发生转移,且缺少有用的靶向治疗和内分泌靶点,晚期化疗效果欠佳。由于具有侵袭早、转移快、病死率高的特点,TNBC是乳腺癌中预后最差的亚型 [4] [5]。为了找到有效的治疗策略,降低TNBC的复发率、提高预后,许多研究人员做了大量的工作 [6] [7],寻找有效的治疗靶点是TNBC有效治疗的关键 [8] [9]。
微小RNA (miRNA)为一段长度约在19~25 nt的非编码单链RNA,靶基因3’-非编码区(UTR)能和其相结合,因而获得抑制靶基因表达的功能并且能够参与很多种生物过程,比如:细胞的增殖、凋亡和分化等生物过程。microRNA对于肿瘤发挥着双重作用,着重表现为抑癌和致癌。据探究,miRNA作为肿瘤抑制因子在TNBC中起着诱导肿瘤细胞凋亡和分化的功能;同时,miRNA调节TNBC的进展主要就是通过上皮–间充质转化(EMT)参与的,这也许与乳腺癌的诊断以及治疗有关系 [10]。导致TNBC患者死亡的主要理由是癌细胞的转移,miRNA有可能通过EMT参与调节TNBC的侵袭性进展。
生物信息学是生命科学和计算机科学相结合综合性学科。研究证明,整合的生物信息学方法可以帮助研究人员更好地探索疾病潜在的分子生物学机制,为临床提供新的诊断标志物和治疗靶点 [11]。本研究以GEO数据库中三阴性乳腺癌(TNBC)上皮间充质转换(EMT)模型的miRNA芯片数据集GSE121657为研究对象,分析三阴性乳腺癌患者差异表达的miRNA,预测与其相匹配的靶基因,并对靶基因进行生物信息学分析,最终获得调控三阴性乳腺癌的核心miRNA,以此为三阴性乳腺癌的相关研究供给新的思路。
2. 材料与方法
2.1. 三阴性乳腺癌(TNBC) miRNA芯片数据
本研究运用到的芯片数据集GSE121657来自于美国国立生物技术信息中心基因公共表达数据库GEO,由Boston University Microarray and Sequencing Resource上传 [12],为三阴性乳腺癌(TNBC)的上皮–间质转化(EMT)模型的miRNA表达数据,共6个样本,其中GSM3442850、GSM3442851、GSM3442852为MIIIshGFP对照组,GSM3442853、GSM3442854、GSM3442855为MIIIshSMAD2处理组。上皮–间质转化使原发癌细胞获得间质特征,并且在转移部位上皮再生形成继发性肿瘤。在该过程中癌细胞的蛋白转录调节会引起细胞特征性改变,所以可以通过对MIIIshGFP对照组与MIIIshSMAD2处理组两组样本进行差异分析,并能够提供预测依据和治疗靶点。对该数据集进行质量分析,结果显示,数据集中包含的六个样本的中心大致处于同一水平线(图1),样本数据总体上差异不大,对其进行差异分析结果可靠。

Figure 1. Boxplot graph of GSE121657
图1. 样本GSE121657数据箱线图
2.2. 三阴性乳腺癌miRNA芯片数据的生物信息学处理
2.2.1. 差异表达miRNA的获取
运用GEO数据库自带的GEO2R在线分析工具,根据|logFC| > 1,P-Value < 0.05,筛查出三阴性乳腺癌患者差异表达的miRNA。
2.2.2. 差异表达miRNA的靶基因预测
本研究使用ENCORI (starBase v3.0)预测靶基因 [13] [14]。starBase数据库是miRNA靶基因预测的综合数据库,整合了多个数据库如TargetScan、miRanda等的信息,大大降低了预测的假阳性。将之前获得的差异miRNA依次输入到ENCORI中,设定Number of supporting Experiments ≥ 5,Program Number ≥ 5 prediction software进行预测。
2.2.3. 差异表达微小RNA对应靶基因的相关功能分析、信号通路分析
使用DAVID在线工具对预测到的靶基因进行GO生物学功能分析和KEGG信号通路分析。把上述的靶基因输入DAVID,从Biological Process、Molecular Function以及Cellular Component三个方面阐明靶基因的生物学功能,分析其参与调控的信号通路。
2.2.4. 靶基因编码蛋白相互作用(PPI)网络的构建与分析
将靶基因上传至STRING在线分析工具,构建靶基因编码的蛋白质相互作用网络,并利用Cytoscape软件对网络进行处理和筛查,筛选出较为关键的靶基因,进一步获得三阴性乳腺癌关键的miRNA分子。
3. 结果
3.1. 差异表达的miRNA
经分析共获得61个差异miRNA,与MIIIshGFP对照组相比,表达上调的miRNA有27个,下调的有34个,见表1和图2。
3.2. 差异miRNA对应的靶基因
将61个差异表达的miRNA输入到在线靶基因预测数据库starBase v3.0后,共获得165个与之相对应的靶基因。

Table 1. Differentially expressed miRNAs
表1. 差异表达的微小RNA

Figure 2. Differentially expressed miRNA detection map of GSE121657 (Red represents upregulation, and blue represents downregulation)
图2. GSE121657差异miRNA的表达值图(红色代表上调,蓝色代表下调)
3.3. 基因功能(GO)富集分析
运用DAVID对靶基因进行Gene_Ontology功能分析,结果显示,165个靶基因主要参与了细胞的定位、细胞过程的正调控等生物学过程,参与了细胞器部分、细胞内外组分等细胞成分的构成,涉及蛋白质及杂环化合物结合等分子功能(如图3)。
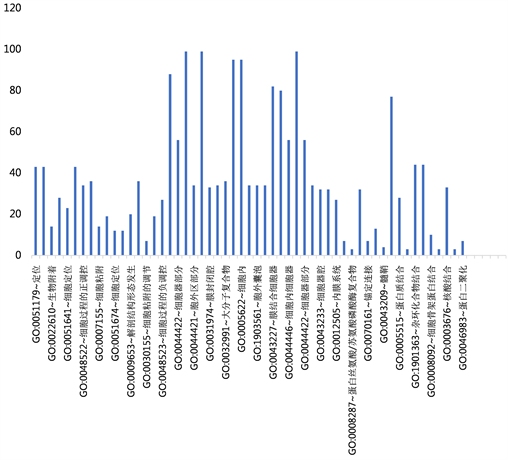
Figure 3. GO function enrichment analysis of target genes (P < 0.05) histogram
图3. 靶基因的GO功能富集分析(P < 0.05)柱状图
3.4. KEGG Pathways分析
通过DAVID在线数据库对165个靶点基因进行信号通路富集分析,以P < 0.05为筛选标准,共获得10条KEGG信号通路,涉及PI3K-Akt、血小板活化、黏附斑激酶、激动蛋白细胞骨架的调节、癌组织中的miRNA等信号通路(表2、图4)。

Table 2. Signaling pathways involved in target genes (P < 0.05)
表2. 靶基因涉及的信号通路(P < 0.05)
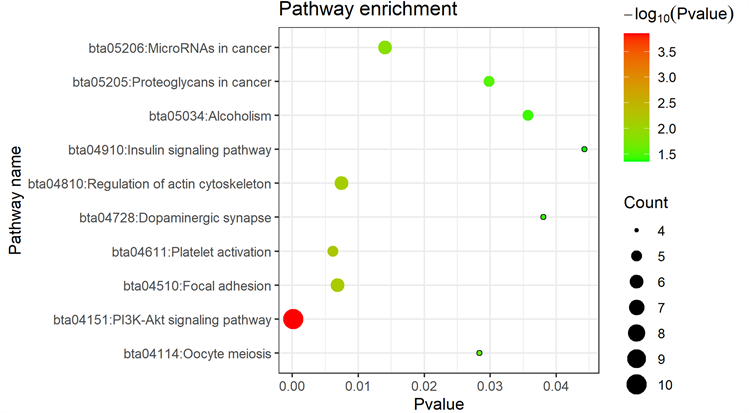
Figure 4. KEGG bubble chart of target genes (P < 0.05)
图4. 靶基因涉及的KEGG气泡图(P < 0.05)柱状图
3.5. 核心基因及关键miRNA
将差异表达的miRNA所对应到的靶基因导入STRING,得到一个涵盖124个靶基因蛋白节点和126条互作关系的蛋白质相互作用网络(图5),利用Cytoscape对蛋白互作网络进行进一步的分析处理。以Degree > 10为筛选条件,从蛋白互作网络中筛选出3个核心靶基因,分别是YWHAG、ACTR2、ACTR3 (图6),并找到了与这3个靶基因相对应的miRNA,即hsa-miR-141-3p,图7显示了该miRNA在TNBC中的表达情况。

Figure 5. Protein interaction network coded by target genes
图5. 靶基因编码的蛋白互作网络图
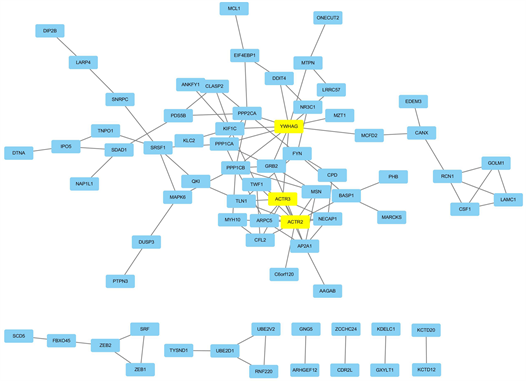
Figure 6. The protein interaction network including key target genes is shown, and the nodes shown in blue in the figure are the key target genes
图6. 显示出关键靶基因的蛋白互作网络,图中蓝色显示且形状较大的节点为关键靶基因

Figure 7. Location of hsa-miR-141-3p in mean difference plot (plotMD)
图7. hsa-miR-141-3p在平均差异图中的位置
4. 讨论
TNBC是指ER、PR以及原癌基因Her-2检查结果全都为阴性的乳腺癌,尽管其有许多的生物学特征与BLBC相类似,但是TNBC和基底细胞样型乳腺癌之间还存在着处在某些免疫表型和基因表达谱上的差异。目前缺乏与之相对应靶向治疗受体,而且对TNBC发生的分子机制也没有完整的了解,对于TNBC的医治手段有限。为此,对TNBC的靶向治疗的探究在如火如荼的开展。
微小RNA是一种非编码小RNA分子,截止到目前人们已经发现了三千多种微小RNA,在这之中绝大多数在动植物体内发挥着关键性的调控作用,是最主要的基因表达调控因子之一。预计在人体内将近2/3的基因都会受到某一个或一组微小RNA的调控 [15]。近几年来,有研究表示单个miRNA可以同时靶向多个mRNA,单个mRNA可以被不同的miRNA协同靶向,miRNA拥有着高度的保守性,参与多种生物学过程,如细胞增殖、细胞脂肪代谢、糖代谢和胰岛素信号转导等 [16]。近些年来,更多的人开始对微小RNA在很多相关疾病中的作用增加关注。在本研究中,采取了生物信息学的手段对三阴性乳腺癌的微小RNA芯片数据进行了研究,筛选后得到61个差异表达微小RNA中,预测得到165个靶基因,这些靶基因主要涉及细胞的定位、细胞周期、细胞分裂以及凋亡、PI3K-Akt信号通路等生物学过程。
PI3K-Akt信号转导通路宽泛分散在细胞中,是一条重要的信号传导途径在细胞内。这一通路过度激活后会促进细胞的过度增殖、抑制细胞凋亡、促使细胞分化异常以及参与细胞自噬等导致肿瘤形成和促进肿瘤转移 [17] [18]。有学者发现,PI3K-Akt信号转导通路在TNBC的发展中起着十分关键作用 [8],这可能与TNBC侵袭性强、预后差的生物学特征和临床特点有紧密关联。所以,找到存在于这一通路中的关键靶点能够为TNBC的患者提供新的医疗方法并且拥有相对稳定的医治结果 [18]。
通过分析蛋白质互作网络,获得了与TNBC相关的核心基因及其对应的miRNA分子,即hsa-miR-141-3p,其在TNBC中表达降低(表1、图7)。研究表明,hsa-miR-141-3p可以通过沉默下游靶点来调节多种癌症如前列腺癌、结肠癌、卵巢癌细胞的迁移和侵袭 [19] [20] [21] [22]。Wong [12] 等人发现,hsa-miR-141-3p与其他一系列假定的肿瘤抑制miRNAs如hsa-miR-4417、hsa-miR-200c-3p、hsa-miR-210-3p等在恶性TNBC细胞中显著下调,经过对hsa-miR-4417、miR-200c-3p、miR-210-3p的RT-qPCR验证,认为hsa-miR-4417是三阴性乳腺癌的抑癌和预后标志物。而本文生物信息学分析结果显示,hsa-miR-141-3p有可能才是调控TNBC发展的更加重要的miRNA分子,需要进一步进行实验验证。