1. 引言
Cdc42全称为细胞分裂周期蛋白(cell division cycle 42, Cdc42),属于Rho GTPases家族蛋白之一,在肿瘤转移和恶化中扮演极其重要的角色。由于Cdc42在细胞分裂调节和肌动蛋白细胞骨架重排中起重要作用,因此细胞生长、迁移、侵袭、极性和粘附都与Cdc42相关 [1] [2]。Cdc42在细胞信号通路中充当分子开关,在细胞皮层之间从非活性的GDP结合形式转化为有活性的GTP结合形式,进而激活下游效应分子,以此对细胞外信号做出生理学应答 [3] [4]。在癌细胞细胞中过表达Cdc42突变体Cdc42G12V,会造成细胞生长阻滞,这说明Cdc42致细胞恶性转化的潜能与Cdc42-GTP和Cdc42-GDP之间的动态平衡有关 [5]。另外,Cdc42也参与了抗癌药物抗性的产生,例如乳腺癌 [6]。与GTPases家族的Rho和Rac蛋白一样,Cdc42也是潜在的研发抗癌药物的靶标 [7] [8] [9],MiRNA和靶向Cdc42的小分子药物以及关联的信号通路,通过抑制Cdc42异常的过表达从而减缓癌细胞转移,为抗肿瘤药物的开发提供新的策略 [10]。近年来针对乳腺癌的治疗,已研发数种以Cdc42为靶标的治疗药物 [11] - [18]。拟肽是一类利用人工合成的物质模拟天然多肽的结构,或者利用构象性模板诱导相邻的多肽序列而形成的具有特异结构的化合物 [19]。我们前期药物合成实验,获得了一些拟肽化合物。本研究拟构建含荧光报告基因的Cdc42真核表达载体,并初步探讨Cdc42协同拟肽化合物对细胞迁移的影响。
2. 实验材料与方法
2.1. 试剂和细胞
真核表达载体pDsRed-C1、pEGFP-C1、大肠杆菌感受态细胞DH5α、A549细胞和293T细胞均为本实验室保存。培养细胞所用材料均购自Gibico公司;限制性核酸内切酶购自Thermo公司;T4 DNA ligase购自Takara公司;Ex Taq master Mix购自康为世纪生物公司;CCK8试剂盒购自日本同仁化学研究所,质粒提取及胶回收试剂盒均为Omega公司产品;PCR引物合成及DNA测序由擎科生物公司完成。PMC4为我们前期合成,其结构如图1,在R1位置处为-F取代基。
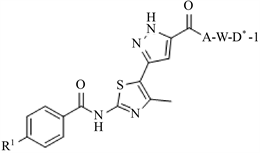
Figure 1. The molecular Structure PMC4
图1. PMC4分子结构示意图
2.2. 携带红、绿荧光报告基因的真核表达载体的构建
以pT-cdc42-2/3质粒为模板,分别使用其对应的引物(表1) PCR扩增目的基因Cdc42-2/3,PCR产物经EcoR I和BamH I两种酶消化后,连接到经相同酶切的真核质粒载体pEGFP-C1和pDsred-C1中,所得质粒命名为pGFP-cdc42-2、pGFP-cdc42-3、pRed-cdc42-2、pRed-cdc42-3。经菌液PCR、质粒PCR以及重组质粒双酶切鉴定分析其正确性。最后将鉴定成功的重组质粒送去测序公司精确鉴定。
2.3. 重组质粒转染293T细胞
提取去内毒素的超纯重组质粒。将生长状态良好的293T细胞以每孔2 × 105个细胞种于6孔细胞培养板,
37 ℃
培养过夜后使用聚乙烯亚胺(polyethylenimine, PEI)作为转染试剂,转染试剂用量DNA:PEI = 3:1,将重组质粒转染至293T细胞。转染6 h后换液,继续培养24 h。在454 nm波长蓝光激发下观察绿荧光,在556 nm波长的绿光激发下观察红荧光。
2.4. CCK-8法检测拟肽化合物细胞毒性
用CCK-8法测定化合物对细胞的毒性,将A549细胞以每孔5 × 104个细胞种于取96孔板,
37 ℃
过夜培养。待细胞完全贴壁后,在对照组加入1 μL DMSO溶液,在实验组加入1 μL不同浓度化合物溶液,控制终浓度为1 μmol/L、10 μmol/L、100 μmol/L,每组三个复孔。继续培养48 h,在每孔加入10 μL的CCK-8试剂,再次放入细胞培养箱中孵育1 h~2 h。取出细胞培养板,用多功能酶标仪在450 nm下测定吸光值。
2.5. 细胞划痕法研究Cdc42协同化合物对细胞迁移的影响
培养A549细胞,种于6孔板,转染重组质粒,转染12 h后,用枪头尖端在细胞孔板中划3条平行直线,PBS冲洗去悬浮的细胞。添加化合物,控制试验浓度为10 μmol/L。分别在0 h,24 h,48 h于显微镜下观察细胞迁移情况,并拍照记录迁移距离,以便后期计算迁移率。
3. 实验结果与分析
3.1. 真核表达载体构建结果与分析
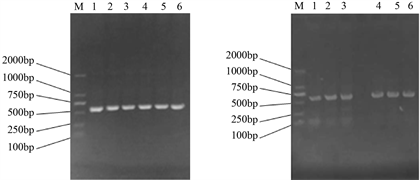
Cdc42基因扩增结果见图2,获得的基因片段长度与预期(576 bp)一致。扩增得到的目的片段、载体pEGFP-C1、pDsred-C1均用EcoR I和BamH I进行双酶切,检测结果见图3。双酶切后的目的基因在500 bp~750 bp之间,双酶切载体条带在5000 bp附近,初步判断双酶切是正确的。
 (a) (b)
(a) (b)
Figure 2. Amplification of the target gene Cdc42-2/3. (a): Amplification of Cdc42-2/3 gene for pEGFP-C1 vector from pT-cdc42-2/3; (b): Amplification of Cdc42-2/3 gene for pDsred-C1 vector from pT-cdc42-2/3; M: DNA maker; 1~3: Cdc42-2 gene; 4~6: Cdc42-3 gene
图2. 目的基因Cdc42-2/3的扩增。(a):从pT-Cdc42-2/3中扩增用于连接pEGFP-C1载体的Cdc42-2/3目的基因;(b):从pT-Cdc42-2/3中扩增用于连接pDsred-C1载体的Cdc42-2/3目的基因;M:DNA Maker 2000;1~3:Cdc42-2基因;4~6:Cdc42-3基因
 (a) (b)
(a) (b)
Figure 3. Double digestion of target gene and expression vector. (a): Double-digested gene and pEGFP-C1; (b): Double-digested gene and pDsred-C1; M: DNA Maker 5000; 1: Double digestion of Cdc42-2; 2: Double digestion of Cdc42-3; 3: Double digestion of pEGFP-C1, pDsred-C1; 4: pEGFP-C1, pDsred-C1
图3. 目的基因和载体的双酶切。(a):目的基因和载体pEGFP-C1的双酶切;(b):目的基因和载体pDsred-C1的双酶切;M:DNA Maker 5000;1:双酶切Cdc42-2;2:双酶切Cdc42-3;3:双酶切pEGFP-C1 (a图)、pDsred-C1 (b图);4:pEGFP-C1 (a图)、pDsred-C1 (b图)
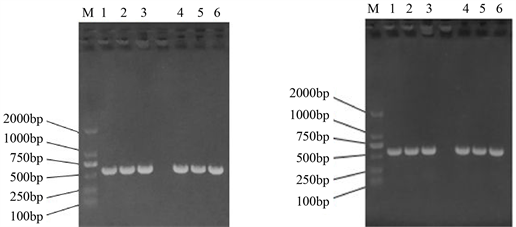
回收酶切产物,连接转化大肠杆菌,分别挑取单菌落进行菌落PCR鉴定,结果如图4,所构建的重组子均能扩增出目的大小的片段(576 bp)。提取重组质粒,结果见图5,重组质粒pGFP-cdc42-2、pGFP-cdc42-3、pRed-cdc42-2和pRed-cdc42-3均提取成功(重组质粒大小为5276 bp),然后以重组质粒为模板,进行质粒PCR鉴定,结果见图6,均能扩增出目的大小的片段(576 bp)。
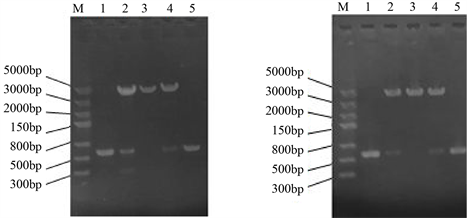
重组质粒进一步进行双切分析,结果如图7,可以看出,成功的切出了大小在567 bp左右的目的基因条带,可以得出Cdc42-2/3成功克隆到pEGFP-C1和pDsred-C1载体上。构建好的表达载体送去测序,
 (a) (b)
(a) (b)
Figure 4. Identification of recombinants by cloning PCR. (a): Identification of pGFP-cdc42-2 and pGFP-cdc42-3 recombinant colonies by PCR; (b): Identification of pRed-cdc42-2 and pRed-cdc42-3 recombinant colonies by PCR; M: DNA Maker 2000; (a1)~(a3): pGFP-cdc42-2 recombinant colony PCR; (a4)~(a6): pGFP-cdc42-3 recombinant colony PCR; (b1)~(b3): pRed-cdc42-2 recombinant colony PCR; B4~B6: pRed-cdc42-3 recombinant colony PCR
图4. 重组子菌落PCR鉴定。(a):pGFP-cdc42-2、pGFP-cdc42-3重组子菌落PCR鉴定;(b):pRed-cdc42-2、pRed-cdc42-3重组子菌落PCR鉴定;M:DNA Maker 2000;(a1)~(a3):pGFP-cdc42-2重组子菌落PCR;(a4)~(a6):pGFP-cdc42-3重组子菌落PCR;(b1)~(b3):pRed-cdc42-2重组子菌落PCR;(b4)~(b6):pRed-cdc42-3重组子菌落PCR
 (a) (b)
(a) (b)
Figure 5. Extraction of recombinant plasmids DNA. (a): Isolation the recombinant plamids pGFP-cdc42-2 and pGFP-cdc42-3; (b): Isolation the recombinant plamids pRed-cdc42-2 and pRed-cdc42-3; M: DNA Maker 5000; (a1) & (a2): The recombinant plasmid pGFP-cdc42-2; (a3) & (a4): The recombinant plasmid pGFP-cdc42-3; (b1) & (b2): The recombinant plasmid pRed-cdc42-2; (b3) & (b4): The recombinant plasmid pRed-cdc42-3
图5. 提取重组质粒DNA。(a):提取pGFP-cdc42-2、pGFP-cdc42-3 重组质粒DNA;(b):提取pRed-cdc42-2、pRed-cdc42-3 重组质粒DNA;M:DNA Maker 5000;(a1) & (a2):重组质粒pGFP-cdc42-2;(a3) & (a4):重组质粒pGFP-cdc42-3;(b1) & (b2):重组质粒pRed-cdc42-2;(b3) & (b4):重组质粒pRed-cdc42-3
 (a) (b)
(a) (b)
Figure 6. PCR identification with the recombinant plasmids used as template. (a): PCR identification with the recombinant plasmids pGFP-cdc42-2 and pGFP-cdc42-3 used as template; (b): PCR identification with the recombinant plasmids pRed-cdc42-2 and pRed-cdc42-3 used as template; M: DNA Maker 2000; (a1) & (b1): PCR with pT-cdc42-2 used as template; (a2) & (a3): PCR with pGFP-cdc42-2 used as template; (a4) & (b4): PCR with pT-cdc42-3 used as template; (a5) & (a6): PCR with pGFP-cdc42-3 used as template; (b2) & (b3): PCR with pRed-cdc42-2 used as template; (b5) & (b6): PCR with pRed-cdc42-3 used as template
图6. 以重组质粒为模板进行PCR鉴定。(a):以pGFP-cdc42-2、pGFP-cdc42-3重组质粒DNA为模板进行PCR鉴定;(b):以pRed-cdc42-2、pRed-cdc42-3重组质粒DNA为模板进行PCR鉴定;M:DNA Maker 2000;(a1) & (b1):以pT-cdc42-2为模板做PCR;(a2) & (a3):以pGFP-cdc42-2为模板做PCR;(a4) & (b4):以pT-cdc42-3为模板做PCR;(a5) & (a6):以pGFP-cdc42-3为模板做PCR;(b2) & (b3):以pRed-cdc42-2为模板做PCR;(b5) & (b6):以pRed-cdc42-3为模板做PCR
 (a) (b)
(a) (b)
Figure 7. Identification of recombinant plasmids by double enzyme digestion. (a): Double enzyme digestion of the recombinants pGFP-cdc42-2 and pGFP-cdc42-3; (b): Double enzyme digestion of the recombinants pRed-cdc42-2 and pRed-cdc42-3; M: DNA Maker 5000; (a1) & (b1): Double enzyme digestion of Cdc42-2 fragments; (a2): Double enzyme digestion of the recombinant plasmid pGFP-cdc42-2; (a3): Double enzyme digestion of pEGFP-C1vector; (a4): Double enzyme digestion of the recombinant plasmid pGFP-cdc42-3; (a5) & (b5): Double enzyme digestion of Cdc42-3 fragments; (b2): Double enzyme digestion of the recombinant plasmid pRed-cdc42-2; (b3): Double enzyme digestion of pDsed-C1 vector; (b4): Double enzyme digestion of the recombinant plasmid pRed-cdc42-3
图7. 重组质粒双酶切鉴定。(a):重组质粒pGFP-cdc42-2、pGFP-cdc42-3双酶切鉴定;(b):重组质粒pRed-cdc42-2、pRed-cdc42-3双酶切鉴定M:DNA Maker 5000;A1&B1:Cdc42-2片段双酶切;(a2):pGFP-cdc42-2重组质粒双酶切;(a3):pEGFP-C1空载体双酶切;(a4):pGFP-cdc42-3重组质粒双酶切;(a5) & (b5):Cdc42-3片段双酶切;(b2):pRed-cdc42-2重组质粒双酶切;(b3):pDsRed-C1空载体双酶切;(b4):pRed-cdc42-3重组质粒双酶切
测序结果与NCBI里面Cdc42比对分析,序列一致,没有碱基缺失与突变。通过以上鉴定分析可以得出,目的基因已经成功插入荧光表达载体中。
3.2. 重组质粒转染293T细胞实验结果与分析
将重组质粒分别转染293T细胞,转染结果见图8,单独的pEGFP-C1转染见图8(b),转染效率高达90%。pGFP-cdc42-2、pGFP-cdc42-3单独转染293T细胞见图8(d)、图8(f),均能有效表达。没有单独的pEGFP-C1亮(见图8(b)),这可能是Cdc42-2/3蛋白与GFP蛋白形成融合蛋白折叠的过程中,Cdc42-2/3遮盖了部分GFP所致。

Figure 8. Transfection of 293T Cells with Recombinant Plasmids. (a) & (b): Transfection of 293T cells with pEGFP-C1; (c) & (d): Transfection of 293T cells with pGFP-cdc42-2; (e) & (f): Transfection of 293T cells with pGFP-cdc42-3; (a), (c), (e): At common optical path; (b), (e), (f): At fluorescence optical path
图8. 重组质粒转染293T细胞。(a) & (b):pEGFP-C1转染293T细胞;(c) & (d):pGFP-cdc42-2转染293T细胞;(e) & (f):pGFP-cdc42-3转染293T细胞;(a)、(c)、(e) 图为白光;(b)、(e)、(f)图为激发光
携带红荧光基因报告基因的质粒转染293T细胞结果见图9,图9(h)是pDsRed单独转染293T细胞的情况。pRed-cdc42-2、pRed-cdc42-3分别转染293T细胞见图9(j)和图9(l),观察到比较亮的红色荧光,与pGFP-cdc42-2 (见图9(d))相比,pRed-cdc42-2/3更有利于Cdc42的表达。

Figure 9. Transfection of 293T Cells with Recombinant Plasmids. (g) & (h): Transfection of 293T cells with pDsRed-C1; (I) & (J): Transfection of 293T cells with pRed-cdc42-2; (K) & (L): Transfection of 293T cells with pRed-cdc42-3; (a), (c), (e): At common optical path; (b), (e), (f): At fluorescence optical path
图9. 重组质粒转染293T细胞。(g) & (h):pDsRed-C1转染293T细胞;(i) & (j):pRed-cdc42-2转染293T细胞;(k) & (l): pRed-cdc42-3转染293T细胞;(a)、(c)、(e)图为白光;(b)、(e)、(f)图为激发光
由图8和图9可以看出,Cdc42的同源异构体基因克隆至pDsRed-C1载体上的表达量要高于该基因克隆至pEGFP-C1载体,这表明后续实验可以采用红荧光报告基因系列重组质粒。
3.3. CCK-8法测定化合物细胞毒性实验结果与分析
在不同拟肽化合物浓度下,用CCK-8法测定的细胞活度结果见表2。所以后续细胞实验时,选择了对细胞毒性较弱的10 μmol/L拟肽化合物浓度,作为试验工作浓度。

Table 2. Cell viability assay by CCK-8
表2. CCK-8法测定细胞活度表
3.4. Cdc42协同化合物对细胞迁移的影响实验结果与分析
将重组质粒转染至迁移能力较强的A549细胞,进行细胞划痕实验。根据细胞划痕实验结果,制作细胞迁移对比图,如图10所示。通过对比分析表3和图10,可以得出以下结论:
① 细胞中过表达基因Cdc42后,细胞迁移率提高;
② 加拟肽化合物刺激细胞后,细胞迁移率同样是升高的;
③ 细胞中过表达基因Cdc42,同时用化合物刺激,细胞迁移率更大。

Table 3. The results of Cell wound scratch assay
表3. 细胞划痕实验结果
4. 讨论
Cdc42作为一类小GTPases,参与了众多的细胞生理过程。当有胞外信号刺激细胞时,胞内的GEFs会被激活,活化的GEFs催化GDP从Cdc42上解离,GTP与Cdc42结合,Cdc42由于GTP的结合而被活化,活化的Cdc42与效应分子结合,导致多种信号级联的激活,调节细胞的增殖、存活、侵袭和迁移等过程 [2]。本研究中用的拟肽化合物PCM4,由本实验合成,针对的靶标是细胞膜上的整联蛋白。实验结果表明,过表达Cdc42后,细胞迁移率提高,当添加PCM4后,细胞迁移率进一步增强,推测PCM4可能在胞外与膜整联蛋白结合,刺激胞内Cdc42的活化,进而通过Cdc42与效应分子结合,引起通过Cdc42介导的信号通路的开启,进而使细胞迁移率增强,具体机制有待于进一步研究。
致 谢
感谢全国大学生创新创业大赛项目(201710488013)的资助;感谢武汉科技大学大学生创新创业大赛项 目(201710488013)的资助;感谢湖北大学生物催化与酶工程国家重点实验室开放基金(SKLBEE2018002) 的资助。
参考文献
NOTES
*通讯作者。