1. 引言
利用分子生物学技术对环境样本中的微生物进行研究,成为目前研究的趋势和潮流。而这一切研究的基础都依赖于其DNA的提取质量和对其特异性片段的PCR扩增效果,因此对某一环境样本中的总DNA的提取方法的选择和PCR条件的选择的研究就显的很有意义。本论文将针对常州北市河河流底泥DNA的提取及PCR (聚合酶链式反应polymerase chain reaction)条件的优化展开研究 [1] [2]。
2. 材料与方法
2.1. 实验材料
样本采集:常州北市河元丰桥、金谷庄园河段的泥质沉积物(样本采集于2012年3月进行,采集位于河流表层0~20 cm处的底泥,随后将采集完成的底泥样品转移至无菌袋内,在4℃条件下立即运回实验室,并置于−80℃下保存)。
2.2. 实验设备
实验设备如下表1。
2.3. DNA的提取 [3] - [9]
2.3.1. 试剂盒提取法 [10]
采用生工生物工程(上海)公司土壤基因组DNA快速抽提试剂盒来提取底泥样品中的总DNA,而后将得到的总DNA溶解于50 μL TE Buffer,再置于−20℃保存。
2.3.2. 氯仿–异丙醇法提取DNA方法 [11] [12] [13] [14]
用氯仿–异丙醇法提取的底泥样品总DNA,得到的DNA可以存储在−20℃或−80℃下长期储存。
2.4. DNA浓度和纯度的检验
利用微量分光光度计对用试剂盒和氯仿–异丙醇法提取出的元丰桥和金谷庄园河段底泥样本总DNA纯度进行测定,然后根据A260/A280、A260/A230和DNA产量的值,对DNA中的蛋白质、酚等进行定量分析。
2.5. DNA的纯度分析
纯度分析首先需要采用琼脂糖凝胶电泳来处理提取得到的底泥总DNA,再采用凝胶成像系统来拍摄胶体,之后便可以直观的得到实验结果。
2.6. PCR的优化 [15] [16] [17] [18]
2.6.1. 引物的选择
本研究所采用的引物为生工生物工程(上海)有限公司生产的相关引物,引物序列见表2。其中,通用引物1可以扩增近乎细菌中含有的所有16SrRNA的序列;通用引物2和通用引物3是16SrDNA的V3区的通用引物 [19]。
如表2所示,在本实验中通过用3种不同的引物对提取出的底泥样品总DNA进行PCR扩增,并比较扩增后的DNA的量和纯度,检验其扩增特异性,找出适于该研究河段的扩增引物。Watanabe等 [20] 研究结果表明,通过采用不同的引物对16SrDNA靶序列进行扩增,可使得其在DGGE分析中的结果不同。在实验中主要研究V3区的扩增特异性,而通用引物1是可以几乎可以扩增细菌中所有16SrRNA序列的引物。
2.6.2. 反应体系的优化
PCR反应体系在一定程度上会受到提取得到的总DNA中的所含有的微量杂质的影响。如若反应体系中所含有的杂质过高或过低,都会导致PCR反应效果不佳。综上,确定最适的DNA模板浓度将会是优化PCR反应体系的重点之一。
2.6.3. PCR反应体系
PCR反应体系为25 μL,具体试剂量如表3。

Table 3. The first PCR amplification template matching table
表3. PCR扩增模板一配比表
2.6.4. PCR扩增条件
扩增程序 [21] [22] [23]:首先进行时长为4 min且温度为94℃的预变性,随后再进行30个循环,各个循环主要包括时长为1 min温度为94℃的变性,再于55℃下进行1.5 min的退火,最后再于72℃下进行1 min的延伸,待循环结束后再于72℃下进行10 min的延伸。
2.6.5. 反应程序的优化
根据相关文献可知退火温度在一定程度上会影响PCR的特异性。当完成变性实验第一步后,反应系统温度会快速下降至40℃~60℃之间,进而有可能会导致引物与模发生结合。鉴于模板DNA较引物的复杂程度要高一些,因此引物与模板之间的碰撞结合率会远高于模板互补链间的碰撞。退火温度及时间的变化在一定程度上会受到引物、靶基序列的长度,碱基组成及其浓度的影响。退火温度范围可由Tm 5℃以上跨越至Tm 10℃以下,如果条件允许,退火温度的降低可以是1℃或0.5℃。
退火温度的选择方面主要体现在降落PCR中相当于对降落起始温度的选择。参考大量文献中所叙述的PCR反应程序,来设计相关对比试验再进行相应的验证。采用3种不同的退火温度,起始温度分别为55℃、60℃和65℃,每个循环降低1℃,20个循环后降低10℃,而后将温度稳定,再相应的持续增加几个循环。同时每组循环各设置共3个平行样,并加入分别为10倍、100倍的稀释液以及空白对照的三个不同浓度的模板。
2.6.6. PCR反应体系
PCR反应体系为25 μL,具体试剂量如表4。

Table 4. PCR amplification template matching table
表4. PCR扩增模板配比表
2.7. PCR扩增条件
扩增程序一:首先进行时长为4 min且温度为94℃的预变性,随后再进行30个循环,各个循环主要包括时长为1 min温度为94℃的变性,再于55℃下进行1.5 min的退火,最后再于72℃下进行1 min的延伸,待循环结束后再于72℃下进行10 min的延伸。
扩增程序二:首先进行时长为4 min且温度为94℃的预变性,随后再进行30个循环,各个循环包括1 min的94℃变性,再于60℃下进行1.5 min的退火,最后再于72℃下进行1 min的延伸,待循环结束后再于72℃下进行10 min的延伸。
扩增程序三:首先进行时长为4 min且温度为94℃的预变性,随后再进行30个循环,各个循环主要包括时长为1 min温度为94℃的变性,在65℃下进行1.5 min的退火,在72℃下继续延伸1 min,待循环结束后再于72℃下进行10 min的延伸。
3. 结果与分析
3.1. 样本DNA提取结果
用试剂盒提取的底泥样品总DNA在干燥后是不可见的,当用TE Buffer溶解后金谷庄园的溶液呈浅褐色,元丰桥的溶液呈无色透明;而用氯仿—异丙醇法提取的底泥样品总DNA在干燥后,可以清楚的看到白色颗粒,金谷庄园颗粒边缘有褐色物质。之后,用TE Buffer溶解后元丰桥的溶液呈无色透明,金谷庄园的溶液呈浅褐色。由此可见,氯仿—异丙醇法提取的底泥样品总DNA比试剂盒提取的DNA的量多。金谷庄园河段的沉积物所还的杂质较多,提取出的总DNA不够纯,所含的腐殖质含量较多。
经比较,可证明氯仿—异丙醇法提取的DNA含量明显。但是最后金谷庄园处的总DNA溶液有明显的变色,这就说明了两种方法提取的DNA都不够纯。DNA中的褐色物质就是明显的未去除干净的腐殖质。由于河流底泥中成分复杂如含有大量有机物、腐殖质等难去除物质,所以在以后提取河流底泥样本总DNA的实验中应当先进行去腐殖质的相关工作。
3.2. DNA浓度及纯度的相应检验结果
DNA的浓度及纯度检测的结果可见表5,用氯仿—异丙醇法提取的底泥样品总DNA比用试剂盒提取的总DNA纯度高且含量多。试剂盒提取的总DNA的OD260/OD280的值均小于1.60,这表明提取的总DNA受到了蛋白质、腐殖质等污染。上述的两种提取方法OD260/OD230的比值均要低于2.0,其结果表明样品在一定程度上受到了有机溶剂的污染。用试剂盒法提取的总DNA受到严重污染,对后续实验有不利影响。

Table 5. Different methods for the DNA concentration and purity
表5. 不同方法所得DNA浓度及纯度
注:表5中DNA样品A和B分别为氯仿—异丙醇法提取的金谷庄园底泥样品总DNA、元丰桥底泥样品总DNA、C和D试剂盒提取的金谷庄园底泥样品总DNA和元丰桥底泥样品总DNA。虽然氯仿–异丙醇法提取的底泥样品总DNA纯度和含量较高,但是其纯度还是受到蛋白质、酚、腐殖质的影响。试剂盒提取的底泥样品总DNA纯度不是很理想。所以在提取环境样本总DNA时需要注意减少蛋白质、腐殖质、酚等物质的污染。
3.3. DNA的纯度分析结果
两种方法提取的总DNA样品凝胶电泳图如图1所示。A是利用氯仿—异丙醇法提取的底泥样品总DNA,B是利用DNA提取试剂盒提取的DNA。其中M是标记(NormalRunTM1kb-11),1是元丰桥处的底泥样品总DNA,2是金谷庄园底泥样品总DNA。

Figure 1. Different methods to extract DNA
图1. 不同方法提取的DNA
由图1可知,图1(a)和图1(b)是提取底泥样品总DNA的两种不同的方法,采用不同的提取方法所提出的DNA的纯度及量在一定程度上也会有所不同。去除掉大量细胞裂解及杂质是贯穿DNA提取过程的关键点,其中用试剂盒(图1(b))提取的样品1,DNA提取物在整体上呈现出褐色,图中可以明显的看出较其它泳道而言,其泳道的背景色显得更深,该结果表明腐殖酸等一系列杂质对提取物有较大的干扰作用。当粗提物的颜色变得越深,表明杂质的干扰作用越大。氯仿–异丙醇法(图1(a)中1,2号泳道也有较明显扩增条带,但其泳道DNA条带比较亮,都有清晰条带,DNA提取量较大,而图1(b)图的扩增条带不够清晰)。所以用氯仿–异丙醇法和试剂盒分别提取的DNA含量不同,亮度有所差别。
如图2所示此图是氯仿–异丙醇法法和试剂盒法提取底泥DNA的重复性实验,其中M是标记(NormalRunTM1kb-11),1、3是氯仿–异丙醇法提取的底泥DNA,2、4是试剂盒提取的底泥DNA。1、2是金谷庄园的底泥DNA样品,3、4是元丰桥的底泥DNA样品。
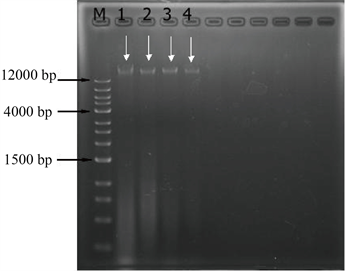
Figure 2. Extraction sediment DNA repetitive experiments
图2. 提取底泥DNA的重复性实验
由图2也可以清楚的看到1、2号泳道颜色较深,这证明金谷庄园的底泥DNA样品中含有较多的腐殖酸、蛋白质等物质。通过条带1和条带3的对比以及条带2和条带4的对比,1、2条带明显比3、4条带清晰。由此可知氯仿—异丙醇法提取的DNA含量要比试剂盒提取的含量高。由实验结果可知金谷庄园河段底泥所含的微生物和腐殖质较多,但提取的总DNA不够纯。而元丰桥河段底泥所含的微生物和腐殖质较少,所以提取总DNA较纯。由此可知在提取底泥样品总DNA之前最好先做去腐处理。
3.4. PCR的优化结果
3.4.1. 反应体系的优化
图3为经过PCR扩增模板一程序扩增后所得到的DNA的琼脂糖凝胶电泳图。由图中可知,M是标记(AccurateRunTM100bp-IV DNA Ladder),1、2、3分别是空白样、稀释到10倍、100倍用通用引物3扩增的DNA,4、5、6分别是空白样、稀释到10倍、100倍用通用引物2扩增的DNA,7、8、9分别是空白样、稀释到10倍、100倍用通用引物1扩增的DNA。
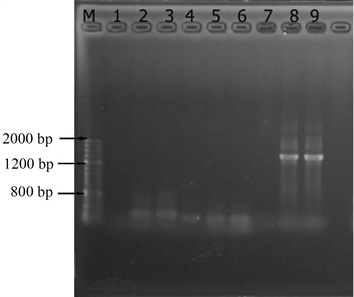
Figure 3. A template for PCR amplification, DNA agarose gel electrophoresis
图3. PCR扩增模板一DNA琼脂糖凝胶电泳图
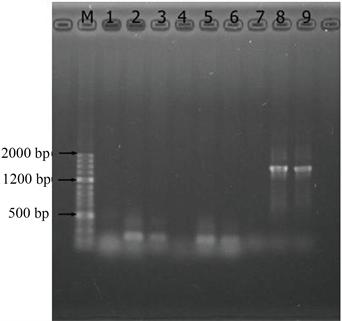
Figure 4. Template for PCR amplification of DNA agarose gel electrophoresis
图4. PCR扩增模板二DNA琼脂糖凝胶电泳图
图4为经过PCR扩增模板二扩增后所得的DNA琼脂糖凝胶电泳图。由图可知,M是标记(AccurateRunTM100bp-IV DNA Ladder),1、2、3分别是空白样、稀释10倍、100倍用通用引物3扩增的DNA,4、5、6分别是空白样、稀释10倍、100倍用通用引物2扩增的DNA,7、8、9分别是空白样、稀10倍、100倍用通用引物1扩增的DNA。
由图3、图4可知,经过PCR扩增之后DNA模板的浓度及纯度都得到了明显的提高。这说明提取的DNA中有与引物相对应的基因片段得到了扩增。由于DNA样品稀释后,DNA样品中的腐殖质、蛋白质等污染物质的浓度显著减少,所以当DNA样品稀释到10倍、100倍的时候污染物质对DNA扩增时的影响减小,PCR扩增的效果更好。图3、图4中1~7泳道中都没有明显的DNA条带,只有8、9两条泳道中有明亮条带,条带位置约为1400 kb。虽然PCR扩增程序不同,但图3、图4的明亮条带位置相同,这证明通用引物1是可以扩增元丰桥河段底泥DNA的特异性引物。
通用引物1中的GC含量位于40%~60%之间,且其核苷长都位于18~24个之间,并且它在最后5个核苷中含3个A或T,上游引物和下游引物溶解温度Tm值接近 [24]。这些条件都使通用引物1有效的与靶序列杂交,使该试验得到了有效的PCR扩增产物。
通用引物2的GC含量大于60%,且在3'末端都含有丰富的GC,这些都能够在一定程度上降低引物的及其同目序列杂交的相关稳定性。
相反通用引物3的序列较其他引物要长许多,一般情况下杂交可能发生于较长的序列与错误配对序列之间,从而会导致其特异性大幅度降低,同时比短序列的杂交进程要慢很多,最终导致产量下降。
3.4.2. 反应程序的优化
由图5可知,该图是经过PCR扩增后的DNA琼脂糖凝胶电泳图。其中,M是标记(Normal RunTM100bp-IV DNA Ladder),2、5和8是稀释到10倍的溶液、3、6和9是稀释到100倍的溶液。用通用引物扩增的DNA,1、4、7是空白样。7~9是经过扩增程序一、1~3是经过扩增程序二、4~6是经过扩增程序三扩增的总DNA样品。
由图可知PCR产量会随着起始温度的不断升高而逐渐减少,主要的表现为条带会变得越来越暗,同时证明了PCR反应在退火温度太高的情况下进行会较为困难。此外,伴随着温度的升高,PCR产物的特异性并没有发生显著的改善。综上所述,本实验中最为合适的起始退火温度为55℃。
从结果与分析中可以明显的看出,通用引物1是对元丰桥的DNA样品进行PCR的扩增的最佳引物,扩增产物大概在1600 bp。10倍稀释液和100倍稀释液都是最适DNA模板的浓度,考虑到经济成本在今后的实验中DNA模板只要稀释10倍即可。而PCR扩增的最佳起始退火温度是55℃。
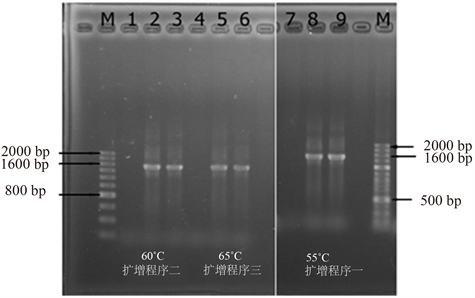
Figure 5. The PCR amplification of DNA gel electrophoresis
图5. PCR扩增DNA凝胶电泳图
由图可知PCR产量会随着起始温度的不断升高而逐渐减少,主要的表现为条带会变得越来越暗,同时证明了PCR反应在退火温度太高的情况下进行会较为困难。此外,伴随着温度的升高,PCR产物的特异性并没有发生显著的改善。综上所述,本实验中最为合适的起始退火温度为55℃。
从结果与分析中可以明显的看出,通用引物1是对元丰桥的DNA样品进行PCR的扩增的最佳引物,扩增产物大概在1600 bp。10倍稀释液和100倍稀释液都是最适DNA模板的浓度,考虑到经济成本在今后的实验中DNA模板只要稀释10倍即可。而PCR扩增的最佳起始退火温度是55℃。
4. 结论
本文以常州北市河元丰桥、金谷庄园河段的泥质沉积物为研究对象,提取总DNA采取的方法主要包括以下两种:氯仿—异丙醇法、试剂盒法,比较这两种提取方法的优劣所在。结果表明,由于河流底泥中成分复杂如含有大量有机物、腐殖质等难去除物质,两种方法提取的DNA纯度都不够高,氯仿—异丙醇法比试剂盒法对北市河河流沉积物样品DNA提取的效果好。
通过用3种不同的引物对提取的DNA样品进行PCR扩增,琼脂糖凝胶电泳的结果对比证明通用引物1可以扩增元丰桥河段底泥DNA的特异性引物。
分子生物学技术对环境样本中的微生物的研究的基础都依赖于其DNA的提取质量和对其特异性片段的PCR扩增效果,引物浓度的不同,会影响PCR扩增产物的纯度。结果表明,10倍稀释溶液和100倍稀释溶液的扩增效果都比较适合。经过改变退火温度,PCR扩增的最佳起始退火温度为55℃。
NOTES
*通讯作者。