1. 引言
胰腺癌(Pancreatic adenocarcinoma, PAAD)是一种恶性程度高的胃肠道肿瘤,其诊断和治疗难度大。PAAD发病率逐年上升,2021年预计新增60,430例,死亡48,220人 [1],其发生与饮酒、吸烟、高脂肪高蛋白饮食、过量饮用咖啡、环境污染和遗传因素有关。糖尿病或慢性胰腺炎患者常发生胰腺癌,并且大约90%属于胰腺上皮导管腺癌 [2] [3]。有数据显示,胰腺癌患者的5年生存率不到10%,使胰腺癌成为恶性胃肠道肿瘤中预后最差的一种 [1],严重威胁着人类的生命健康。胰腺癌的发病隐匿并且进展迅速,一旦发现,大多数患者已经失去了最佳治疗机会。因此,寻找准确、快速预测胰腺癌的肿瘤生物标志物成为当务之急。
细胞焦亡(Pyroptosis)是一种炎症性细胞程序性死亡,伴随着大量炎性细胞因子的释放 [4] [5]。大量研究表明,细胞焦亡作为一种炎性反应,所产生的炎性环境能够诱导正常细胞发生癌变,为肿瘤的发生和进展提供良好的生存环境 [6] [7]。因此,本文旨在筛选出胰腺癌中的细胞凋亡相关基因,为胰腺癌的诊断及靶向药物的研究提供新的方向,同时构建风险预测模型来进一步评估胰腺癌患者的预后。总之,本文将有助于我们更好地理解胰腺癌的进展机制,为胰腺癌的诊断和治疗提供新的靶点。
2. 材料与方法
2.1. 人胰腺癌基因表达数据的收集
从TCGA (The Cancer Genome Atlas)数据库(https://portal.gdc.cancer.gov/)获取人胰腺癌和正常组织的RNA-seq数据及相应的临床病理信息,包括178例胰腺癌患者肿瘤组织及4例癌旁的正常组织 [8]。考虑到TCGA数据集中PAAD的正常样本数量较少,结合GTEx (Genotype-Tissue Expression Project)从UCSC-Xena (https://xenabrowser.net/datapages/)下载了167例人正常胰腺组织的基因表达谱。后续所有处理和归一化均采用R3.6.1完成。
2.2. 细胞焦亡相关基因的获取
在MsigDB (http://www.gsea-msigdb.org/gsea/msigdb/index.jsp)上搜索并下载细胞焦亡相关的数据集,然后利用GSEA4.0.2进行基因集富集分析 [9],提取细胞焦亡相关的基因及表达量,应用“GSVA”软件包计算每个样本中特定基因集的富集分数 [10],使用单样本GSEA (ssGSEA)得分对细胞焦亡通路进行生存分析。
2.3. 构建蛋白互作网络与差异表达基因的筛选
利用STRING (https://www.string-db.org/)构建细胞焦亡基因的蛋白质相互作用网络 [11],构建标准位相关系数大于0.4,线的粗细代表相关性的强弱。利用“limma”软件包进行差异分析 [12],筛选P < 0.05的基因,并用“pheatmap”软件包进行可视化 [13]。
2.4. 生存分析
将差异表达的基因与生存数据合并,运用单因素Cox回归分析筛选出与预后显著相关的细胞焦亡相关基因。同时,利用“survival”软件包进行总生存率(overall survival, OS)的生存分析 [14],筛选标准为P < 0.05。
2.5. 细胞焦亡预后风险预测模型的构建及验证
结合差异分析、单因素Cox回归分析和生存分析,利用“VennDiagram”筛选出交集基因用于后续分析 [15]。通过Lasso回归分析确定风险特征,风险评分按以下公式计算:
。Coefi是系数,Exi是每个选定的细胞焦亡基因的表达。该公式用于计算每位患者的风险评分,然后根据风险评分中位数,将TCGA-PAAD数据集分为高风险组和低风险组。采用Kaplan-Meier生存分析比较两组患者的预后,受试者操作特征(ROC)曲线用于评估风险预测模型的准确性。
GEPIA2.0 (http://gepia2.cancer-pku.cn/)是一个综合分析来自TCGA和GTEx数据库的RNA-Seq表达数据的在线网站 [16],我们利用GEPIA对风险模型基因的差异表达及生存分析进行验证。
2.6. 确定模型风险评分与临床病理特征之间的关系
为了更好地了解风险评分在PAAD中的预测作用,我们采用“pheatmap”软件包系统地研究了风险评分与临床病理特征之间的关系,包括生存状态、年龄、性别、病理分级、分期和TNM分期。不仅如此,我们还研究了风险评分与所选细胞焦亡风险基因表达水平之间的关系。此外,我们使用“forestplot”软件包进行了单因素Cox回归分析和多因素Cox回归分析确定风险评分是否是胰腺癌患者的独立预后指标,P < 0.05表示有统计学意义。
2.7. 统计学分析
所有统计学分析均采用R3.6.1完成,P < 0.05被认为具有统计学意义。
3. 结果
3.1. 细胞焦亡基因富集分析与差异基因
从MsigDB仅下载了1个细胞焦亡相关基因的数据集:REACTOME_PYROPTOSIS,包含了26个相关基因。GSEA富集分析发现细胞焦亡在肿瘤样本中更加活跃,标准化的富集分数(NES)为1.62,标准化的P = 0.041,错误发现率(FDR)为0.041 (图1(A))。同时,通路的生存分析表明细胞焦亡(P = 0.041)与胰腺癌的生存率呈负相关(图1(B))。图1(C)显示了细胞焦亡相关基因的蛋白–蛋白相互作用(PPI)。通过差异分析发现,与细胞焦亡相关得26个基因中有23个均在胰腺肿瘤组织中上调(图1(D)),***,P < 0.001;**,P < 0.01;*,P < 0.05。

Figure 1. Enrichment analysis and differential expression analysis of pyroptosis-related genes in pancreatic adenocarcinoma
图1. 胰腺癌中细胞焦亡基因的富集分析与差异分析
3.2. 细胞焦亡相关基因的预后作用
为了建立与细胞焦亡相关的风险预测模型,我们采用了单变量Cox回归分析分析了26个细胞焦亡基因与总生存率的关系,发现CASP4、IL18、CHMP4C、CHMP2B、BAK1及CHMP3与OS显著相关(P < 0.05;表1)。Kaplan-Meier生存分析发现IL18 (P = 0.003)、CHMP2B (P = 0.007)、CHMP3 (P = 0.027)、CASP3 (P = 0.045)及CHMP4 (P = 0.009)与胰腺癌患者的生存呈显著负相关(图2(A)~(E))。韦恩图筛选出3个交集基因(图2(F)),分别是IL18、CHMP2B和CHMP3,用于后续分析。

Table 1. Univariate Cox regression analysis of prognosis related genes
表1. 预后相关的单因素Cox回归分析

Figure 2. Survival analysis of pyroptosis-related genes in pancreatic adenocarcinoma
图2. 胰腺癌细胞焦亡基因的生存分析
3.3. 构建胰腺癌的风险预测模型
应用LASSO-Cox回归算法构建胰腺癌患者焦亡相关基因的风险评分模型,选择IL18、CHMP2B和CHMP3构建风险信号,计算胰腺癌患者的风险评分(图3(A),图3(B))。以风险评分的中值6.824711作为基础,将胰腺癌患者分为高风险组和低风险组。生存分析结果表明,高风险组胰腺癌患者的总体生存率更差(图3(C))。由于胰腺癌患者的总体生存时间不长,因此ROC曲线仅验证了0.5年、1年和3年,图3(D)~(F)显示该风险特征的0.5年、1年和3年ROC曲线的曲线下面积(AUC)分别为0.763、0.639和0.684,表明该风险标志物对胰腺癌患者的短期生存率预测具有良好的特异性和敏感性。
3.4. 风险预测模型基因标志物的验证
为了提高胰腺癌细胞焦亡基因风险评分模型的准确性,我们利用GEPIA2.0对这三个基因标志物进行了差异表达和生存分析的验证。如图4(A)~(C)所示,与正常组织相比,IL18、CHMP2B和CHMP3在胰腺癌肿瘤组织中的表达显著升高。并且,从生存曲线可知,IL18、CHMP2B和CHMP3均是预后不良因素(图4(A)~(C))。多基因生存分析显示,与低表达IL18、CHMP2B和CHMP3的胰腺癌患者相比,高表达的患者总体生存率(P = 0.0014)和无病生存率(DFS, P = 0.013)均显著较差(图4(D),图4(E))。
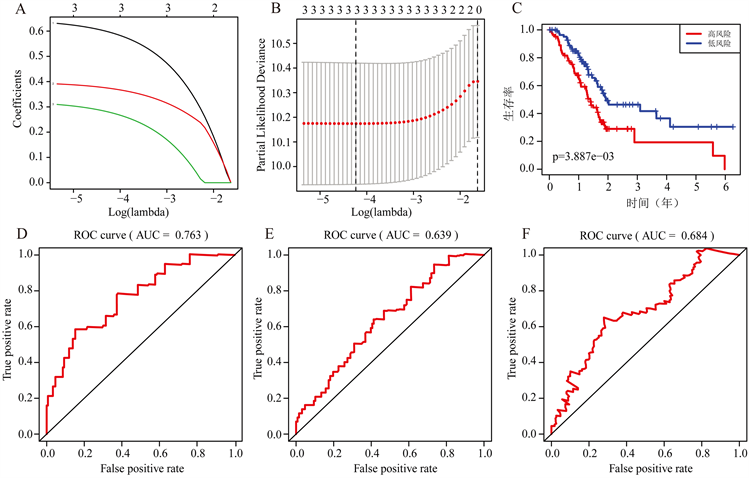
Figure 3. Construction of risk prediction model of pancreatic cancer
图3. 胰腺癌焦亡基因风险预测模型的构建

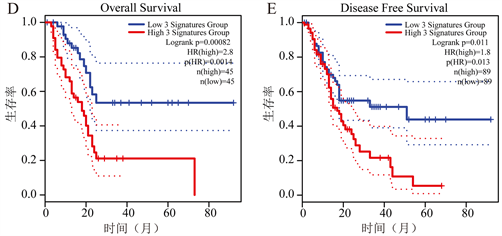
Figure 4. Validation of gene markers of risk prediction model
图4. 风险预测模型基因标志物的验证
3.5. 风险评分和其他临床病例特征的关系
M分期有大量的缺失而被排除,仅保留了具有完整生存数据和临床病理信息的167个患者。研究风险评分与临床病理特征之间的关系,发现风险评分与胰腺癌患者的生存状态密切相关(图5(A))。此外,与低风险组患者相比,高风险组的胰腺癌患者的IL18、CHMP2B和CHMP3的表达水平更高(图5(A)),***,P < 0.001;**,P < 0.01;*,P < 0.05。接下来,研究风险评分是否能作为一个独立的预后指标,单因素Cox回归分析发现风险评分、年龄和N分期与胰腺癌患者的预后密切相关(图5(B))。结合多因素Cox回归分析表明风险评分可以作为胰腺癌患者的独立预后指标(HR = 1.105, 95%CI = 1.059~1.154) (图5(C))。

Figure 5. Independent prognostic analysis of risk prediction model
图5. 风险预测模型的独立预后分析
4. 讨论
胰腺癌作为高度恶性的胃肠道肿瘤,其生存率极低,仅为10% [1]。因此,寻找一种可靠的胰腺癌预后指标一直是热门话题。细胞焦亡作为广泛存在于肿瘤细胞中的死亡方式,在肿瘤发生发展中的作用随着研究的深入而日渐凸显。对于肿瘤而言,细胞焦亡是一把双刃剑。一方面,作为一种固有免疫机制,焦亡能抑制肿瘤的发生发展;另一方面,作为一种炎性细胞死亡方式,焦亡又为肿瘤生长提供了适宜的微环境。细胞焦亡通路的关键成分:炎性小体、焦孔素蛋白和促炎细胞因子等均与肿瘤的发生、侵袭和转移相关 [17]。有研究表明,炎症小体NLRP3参与宫颈癌的固有免疫反应过程,激活的NLRP3炎症小体能够诱导宫颈癌细胞发生焦亡 [18] [19]。人肺癌细胞A549的研究表明细胞焦亡的发生可以促进肺癌细胞的生长、增殖、迁移以及侵袭 [20]。由此可见,细胞焦亡在肿瘤的发生发展过程中有着重要作用,因此细胞焦亡相关基因不仅是细胞焦亡的调节因子,更有望成为肿瘤早期诊断的标志物与肿瘤抗癌治疗的新靶点。
本研究筛选到了IL18、CHMP2B和CHMP3是PAAD的预后相关基因,其在胰腺癌中的表达显著增加,并且高表达与预后差显著相关。通过构建三个标志物的有效风险预测模型,提示这三个基因可能成为胰腺癌治疗和预后评估的靶点,它们的表达越高PAAD患者预后越差。IL18 (Interleukin 18)是一种炎性细胞因子编码基因,血清IL-18的水平可能与ENKTL患者的血液吞噬功能和不良生存结局有关 [21]。而有的研究表明炎症小体激活IL-18和IL-1β是一种结合先天性和适应性免疫应答系统的协同作用,因而产生抗病毒和抗肿瘤活性 [22]。人乳头瘤病毒感染的控制依赖于合适的免疫应答,并且需要通过炎症小体激活IL-18和IL-1β,以刺激先天免疫系统和适应性免疫系统之间的协调并产生有效的抗肿瘤反应 [23]。CHMP2B (Charged Multivesicular Body Protein 2B)编码异聚体ESCRT-III复合物的一个组成部分,该复合物在细胞表面受体的再循环或降解中起作用 [24]。虽然CHMP2B与肿瘤的关系研究较少,但也有文献报道CHMP2B的过表达提示较高的肿瘤分级 [25]。CHMP3 (Charged Multivesicular Body Protein 3)与肿瘤进展的关系与我们的结论相反,有研究表明过表达CHMP3可以抑制A549肺泡上皮细胞的上皮间质转化进程 [26]。也有研究称CHMP3在乳腺癌等多种人类癌症中显著下调,CHMP3表达越高,患者3年和5年生存率越高 [27],但是我们的研究发现CHMP3在胰腺癌中显著上调,其表达越高预后越差。
本研究利用生物信息学分析技术对来自于TCGA和GTEx的胰腺癌数据进行了系统分析,筛选出了与胰腺癌预后显著相关的细胞焦亡相关基因,尽管如此,本研究仍然存在一定的局限性,为了提高验证结果的准确性,还需要通过进一步的临床实验进行探究,因此后期将进一步通过体内或体外实验来验证IL18、CHMP2B和CHMP3的表达与胰腺癌进展的相关性,探索它们的预后价值,并深入探究其影响机制。
NOTES
*通讯作者。