1. 引言
乳腺癌是世界上最常见,且致死率较高的癌症之一。雌激素受体被证实同乳腺癌细胞的恶性增殖和侵袭转移存在直接的联系 [1]。雌激素受体α亚型(ERα)是细胞内响应雌激素刺激并且介导信号转导的关键因子,同时ERα也是临床上重要的药物靶点 [2]。能够拮抗ERα活性的化合物可能是治疗乳腺癌的候选药物。作为候选药物的化合物,需要具备良好的抗乳腺癌活性,在人体内具备良好的药代动力学特质和安全性,即ADMET性质。
在药物研发中,为了节约时间和成本,一般通过建立化合物活性预测模型的方法来筛选潜在活性化合物,常采用机器学习和深度学习等智能算法来提高模型的计算效率和预测精度。王正国 [3] 用支持向量机和神经网络两种机器学习方法建立选择性环氧化酶-2抑制剂的活性预测模型。胡珊珊 [4] 基于传统机器学习算法构建蛋白质热点残基的预测模型,以及基于深度学习的算法分别构建药物–靶标相互作用的预测模型和QSAR药物活性筛选模型。李伟 [5] 总结了深度学习在虚拟化合物库的生成,化合物活性、代谢和毒性的预测,以及有机合成反应预测中的应用。邓志罗 [6] 利用向量机筛选具有良好ADME/T性质及生物活性的化合物。张向东 [7] 应用支持向量机对拮抗药化合物的生物活性进行了预测,计算核函数及参数的选择和优化问题,建立了药物活性预测的数学模型。胡小英 [8] 采用自组织神经网络,支持向量机和聚类分析方法对水解酶催化的反应和氧化还原酶催化的反应进行了分类预测。
本文根据ERα拮抗药物的相关数据信息,利用神经网络建立化合物生物活性的定量预测模型,利用二次SVM建立ADMET性质的分类预测模型,以此可作为优化ERα拮抗剂的生物活性和ADMET性质的预测服务的手段。本文数据来自2021年华为杯中国研究生数学建模竞赛D题中1974个化合物对ERα的生物活性数据,1974个化合物的729个分子描述符信息和1974个化合物的5种ADMET性质的数据。
2. ERα生物活性的定量预测模型
2.1. 重要分子描述符的筛选
先通过随机森林对分子描述符进行数据一次降维,仅选取部分具有代表性的描述符,然后进行系统聚类分析结合斯皮尔曼相关性检验来进行二次筛选,得出最终的20个重要的典型分子描述符。
2.1.1. 随机森林特征重要度分析
本文基于随机森林的特征贡献度算法建立自变量特征筛选模型,进行分子描述符的贡献度分析。综合考虑求解运行时间和分析结果的准确度,使用Python应用随机森林特征重要度算法,设定算法的训练占比为0.75,决策树个数K = 300,决策树分类准则为MSE,决策树最大深度为20,分裂一个内部节点需要的最少样本数为2,每个叶子节点需要的最少样本数为2。通过运行程序可以得到729个自变量的贡献度排名,将贡献度由大到小排序,筛选出对生物活性pIC50值的贡献度总和超过90%的分子描述符,如图1所示。由图可知,选择累积贡献度达到90%的分子描述符,数量约为70个,说明选取贡献度排名前70名的变量已经可以有足够的信息来预测生物活性值pIC50。
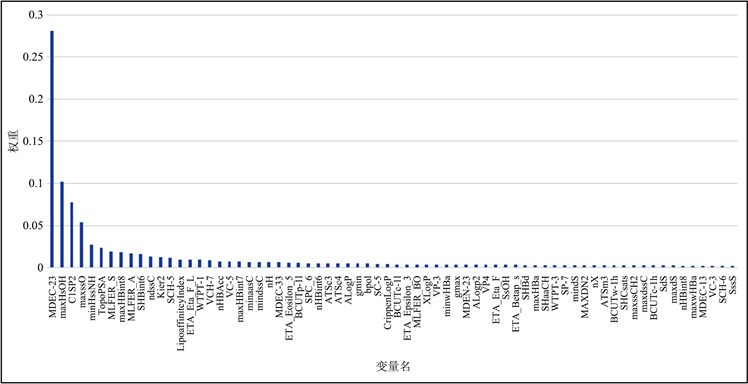
Figure 1. Random forest independent variable weight diagram
图1. 随机森林自变量权重图
2.1.2. 系统聚类和斯皮尔曼等级相关系数检验
为了更为精准的研究影响评价量的重要因素,对随机森林算法求得的重要自变量作为原数据进行数据标准化,即将其减去它的均值,再除以该变量的标准差,计算得到新的变量值,作为系统聚类的原数据。数据矩阵进行标准化处理的表达式如公式(1):
(1)
式中,
,
为矩阵
每一列的均值和标准差。
基于标准化后的70个新自变量值,采用SPSS软件对其进行系统聚类分析,采用系统聚类的合并算法通过计算两类数据之间的平方欧式距离,对距离最为接近的两类数据作为组合,反复迭代运算,直到70个变量全部完成距离计算,聚类完成 [9]。
对已有的聚类结果,通过斯皮尔曼等级相关系数检验自变量分子描述符和因变量生物活性值之间的相关性是否显著。表1为最终筛选的20个重要分子描述符的聚类类别和斯皮尔曼相关系数。

Table 1. Spearman correlation coefficient and clustering category of independent variables
表1. 自变量的斯皮尔曼相关系数及所属聚类类别
注:*表示显著性水平为0.1,则置信水平为90%,**表示显著性水平在0.05,置信水平为95%
从表1可看出,分子描述符minaasC在90%的置信水平下显著,其余19个分子描述符在95%置信水平下显著。故最终得到的20个分子描述符具有很强的代表性。
2.1.3. 典型变量的合理性验证
对确定的20个对生物活性值最具显著影响的分子描述符应用斯皮尔曼相关系数检验变量之间的相关性,图2为20个主要变量之间的斯皮尔曼相关系数矩阵热力图。

Figure 2. Spearman correlation coefficient matrix thermodynamic diagram of 20 variables
图2. 20个变量的spearman相关系数矩阵热力图
通过相关系数矩阵,颜色较浅的色块占据的矩阵中的绝大多位置,表明所得到的20个主要变量相互之间具有很强的独立性。又由于该主要变量通过了随机森林的贡献度和斯皮尔曼显著性检验,因此其具有很强的可靠性,能够很好地用于后续问题的处理。
2.2. 基于神经网络的ERα生物活性的定量预测模型
BP神经网络算法具有多输入多输出、非线性拟合能力强、准确率高、鲁棒性好等优点。基于贝叶斯(Bayesian Regularization, BR)算法训练的神经网络以最大后验概率为目标,将BP神经网络中的各网络权值看作随机变量,自主调节两个正则化系数的大小,能够在使得网络均方误差最小的基础上有效地控制网络的复杂程度 [10]。为了能够根据现有数据较为准确的预测生物活性pIC50的值,所以采用基于贝叶斯训练算法的BP神经网络方法。
2.2.1. 基于BP神经网络的定量预测建模
在设计神经网络时,设置输入节点数为20,即上面确定的20个描述符;设置输出节点数为1,即生物活性pIC50值,但目前没有完整的理论来支持直接设定中间层的节点数(一般通过人工经验获得,或经反复测试,根据预测效果选取较优值作为最终设定值)。选择70%的数据样本为训练集,30%的样本为验证集和测试集。经反复测试比较,最终选取隐含层神经元数为20。建立的神经网络结构图如图3所示。

Figure 3. Structure diagram of neural network
图3. 神经网络结构示意图
对于训练出来的结果,通常用神经网络结果图中的相关度R值或均方误差来判断神经网络模型的预测精度。均方误差(MSE)是预测值和真实值之差的平方和的平均值 [11]。其表达式如公式(2):
(2)
当R值越接近于1,MSE值越小,该模型的预测精度就越高,泛化能力越好。
2.2.2. 基于BP神经网络的定量预测建模
由贝叶斯算法神经网络模型计算预测值与真实值间的相关度R值、均方误差MSE值和模型预测的效果,如图4所示。
在本方法中,设定了70%的样本作为训练集,30%的样本作为测试集。训练集R = 0.92351,测试集R = 0.821,MSE值为0.29947。可看出预测精度还是比较可观的。
2.3. 定量预测模型的验证
将测试集中使用BR神经网络预测模型得到的生物活性pIC50预测值和实际值进行对比,根据实际数据样本,每间隔十个取一次样本。得到预测值与真实值的对比图5所示。
从图5预测值和实测值的拟合关系来看,BR神经网络预测模型能够在一定程度上较为准确的对生物活性pIC50值进行预测。总体而言,建立的BR神经网络预测模型是较为合理可行的。
为了更加直观地反映出BR神经网络预测模型的可靠性,即模型所得预测数据的准确率,进一步计算预测值和真实值之间的误差百分比,如图6所示。可看出平均误差为0.07,所以通过训练得到的BR神经网络预测模型的预测结果是较为准确的。
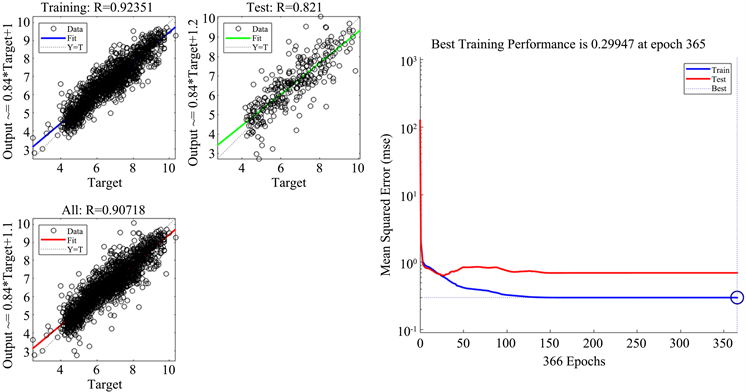
Figure 4. Prediction results of BR neural network model
图4. BR算法神经网络模型预测结果
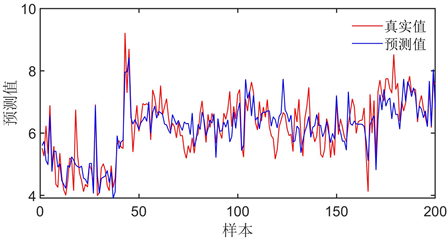
Figure 5. Comparison between real value and predicted value of pIC50
图5. pIC50的真实值与预测值的对比

Figure 6. Percentage of error between actual value and predicted value
图6. 实际值与预测值的误差百分比
3. 基于二次SVM的ADMET性质分类预测模型
在关注化合物生物活性的同时,还需要考虑其ADMET性质。分别是:1) 小肠上皮细胞渗透性(Caco-2),可度量化合物被人体吸收的能力;2) 细胞色素P450酶(Cytochrome P450, CYP) 3A4亚型(CYP3A4),这是人体内的主要代谢酶,可度量化合物的代谢稳定性;3) 化合物心脏安全性评价(human Ether-a-go-go Related Gene, hERG),可度量化合物的心脏毒性;4) 人体口服生物利用度(Human Oral Bioavailability, HOB),可度量药物进入人体后被吸收进入人体血液循环的药量比例;5) 微核试验(Micronucleus, MN),是检测化合物是否具有遗传毒性的一种方法。
对于分类模型,本文采用二分类法。Caco-2:“1”代表该化合物的小肠上皮细胞渗透性较好,“0”代表该化合物的小肠上皮细胞渗透性较差;CYP3A4:“1”代表该化合物能够被CYP3A4代谢,“0”代表该化合物不能被CYP3A4代谢;hERG:“1”代表该化合物具有心脏毒性,“0”代表该化合物不具有心脏毒性;HOB:“1”代表该化合物的口服生物利用度较好,“0”代表该化合物的口服生物利用度较差;MN:“1”代表该化合物具有遗传毒性,“0”代表该化合物不具有遗传毒性。对于二分类问题有很多解决方法,比如Fisher判别式,神经网络二分类,支持向量机(SVM)等等,各有优劣。考虑到样本数据量大,且变量之间的相关程度差距不同,若直接进行化合物ADMET性质分类预测模型的构建,这会影响预测模型的求解时间和预测数据的准确率,因此采用主成分分析法进行数据降维处理,然后再使用二次支持向量机构建预测模型。
3.1. 二次SVM建立分类预测模型
支持向量机(SVM)是一种判别分类器,由分类超平面所定义。SVM解决分类问题的重要环节是最优的决策边界的选择,而决策边界应分别远离两类数据点 [12]。
SVM的基本思路是寻找需要进行分类的样本数据集中的最优解超平面,然后通过输出最佳超平面来实现测试样本分类。SVM首先通过非线性目标函数将输入数据从低维数据空间映射到高维数据空间中,再在高维空间进行分类,取得在原空间进行分类的效果 [13]。利用极大化求解思想,得到分类决策函数表达式公式(3)。式中,
为与第i个样本相对应的Lagrange乘子;
为偏移量;
为核函数,表示映射到高维数据空间的两点的内积。预测过程中,核函数和惩罚系数的大小对算法的表现有非常关键的作用 [14]。
(3)
常用的核函数有线性核函数、多项式核函数、高斯核函数(RBF)等 [13]。
本文在传统支持向量机预测的基础上采用二次多项式核函数支持向量机来构建分类预测模型,以提高模型的预测精度,多项式核函数表达式如下:
(4)
支持向量机又有一对多的SVM算法和一对一的SVM算法。对于二分类问题,采用一对一的SVM算法,即由分子描述符和化合物的ADMET性质两类样本数据构成一个分类器。
3.2. 分类预测模型的求解分析
分析分类模型的性能的优劣时,常画出它们的ROC曲线图,考察它们的AUC值及测试集的准确度。也可以直接看ROC曲线离对角线(AUC = 0.5)的最远点的假正率和真正率。当AUC = 1时,是最完美的分类器,当0.5 < AUC < 1时,数值越大,分类效果越好。所以本文采取ROC曲线图对Caco-2、CYP3A4、hERG、HOB、MN的分类预测模型进行分析。统一选取70%的样本量作为训练集,30%的样本量作为测试集。
五种ADMET性质的分类预测结果ROC曲线如图7所示。
首先进行PCA降维,保留了35个成分,成分的解释方差和已经达到95%以上,可以作为数据分析使用。利用Matlab的分类学习机,五种ADMET性质(Caco-2,CYP3A4,hERG,HOB,MN)的测试集准确度分别为91.7%,93.2%,89.8%,86.7%,95.3%。从上面的ROC曲线图可看出,AUC值分别为:0.95,0.96,0.96,0.89,0.97。采用二次SVM方法的分类预测模型对五种指标的平均准确度达到了92%以上,预测准确度比较可观。所以该模型能够较准确地对ADMET性质进行分类。


Figure 7. ROC curves for five ADMET properties
图7. 五种ADMET性质的ROC曲线
4. 粒子群算法解决目标优化问题
本文设置的优化目标为:寻找并阐述化合物的哪些分子描述符,以及这些分子描述符在什么取值时,能够使化合物对抑制ERα具有更好的生物活性,同时具有更好的ADMET性质(五个ADMET性质中,至少三个性质较好)。
作为启发式算法之一的粒子群优化算法(PSO)能够有效地解决约束优化问题,即在一定的优化约束条件下,寻求目标函数的最大值或最小值 [15]。它源于对鸟类群体行为的研究,核心思想在于使用群体里面的个体对信息的共享,使得在问题求解区域内,整个群体的运动从无序到有序演化,从而得到问题的可行解 [16]。PSO算法具有很多显著的优点,它对问题的信息没有太大依赖,通用性很强,其原理相对简单,易于实施,而且收敛速度比较快,能够很容易的飞跃局部最优信息,擅长处理多变量全局最优解问题 [17]。基于PSO的约束优化是将粒子群的搜索区域设置在约束条件簇里面,也就是在可行解的范围内寻求最优值。由于粒子群算法具备众多优点,非常适合于解决目标优化问题,所以本文选择采用PSO来进行目标优化。
4.1. 双目标寻优转化为单目标寻优
上面已经建立了ERα生物活性的定量预测模型(采用确定的20个主要变量),但是这些变量对ADMET性质的影响是未知的。若能通过这20个变量准确预测出五种ADMET性质,那么就可以转化为双目标寻优问题。所以优先考虑这些变量对Caco-2、CYP3A4、hERG、HOB、MN分类预测模型预测准确度如何。通过MATLAB分类学习机中的袋装树分类方法,建立了五种ADMET性质的分类预测模型,结果显示虽然没有第3节中通过PCA降维再进行预测得到效果好,但是仍然取得了不错的效果。
ADMET性质预测模型的AUC值及测试集准确度如表2所示。可以看出,这五个分类模型的预测准确度都很好,平均可达90%左右,所以利用上面得到的20个主要变量来预测化合物的这五种ADMET性质是比较合理的。

Table 2. AUC value and test set accuracy of ADMET property classification prediction model
表2. ADMET性质预测模型的AUC值及测试集准确度
记这五个分类预测模型为
,i = 1, 2, 3, 4, 5。至此可以通过
以及ERα生物活性的定量预测模型的
来确定ADMET性质的好坏和pIC50的大小,将ADMET至少四个好的性质作为前提,尽可能使pIC50更大,至此就将双目标寻优问题转换为单目标寻优问题。
4.2. 优化目标及约束的设定
1) 决策变量
一共采用20个变量,均属于可变变量,所以决策变量的个数为20。记决策变量为:
(5)
2) 目标函数
优化目标为找出能够使化合物对抑制ERα具有很好的生物活性,且同时让化合物的ADMET的性质更好的分子描述符及其取值或取值范围。以pIC50值为优化目标,让pIC50值尽可能大。
目标函数的表达式为:
(6)
式中的net函数即为神经网络的ERα生物活性的定量预测模型。
3) 约束条件
对于该问题,已经得到了20个主要变量,在进行约束条件设置时,记样本中第i (i = 1, 2, 3, ∙∙∙, 20)个变量的数据值为Pi。那么变量xi的约束条件之一为数据样本中对应的最大值和最小值。此外,设定五种性质中有四个或五个好,即使通过分类预测模型对某一个性质预测出错,仍然有很大把握保证5个中至少有3个性质好,提高了模型的容错率。通过分析可知,五个指标分别为1,1,0,1,0时,ADMET的五个性质最好,为便于分析,对第三和第五指标取非,进行正向化。具体的约束条件表达式为:
(7)
4.3. 粒子群算法寻优求解分析
给出PSO算法的具体流程如下:
1) 初始化群体规模为m = 80的粒子群,设定所有粒子的初速度v0与初始位置x0,惯性因子在0.1~1.1中自适应,个体学习因子和社会学习因子为1.49,最大迭代次数1000次,维数为20;
2) 依据适应度计算方式,计算出每个粒子的适应度;
3) 比较当前每个粒子的适应度与其历史经过的最好位置的适应度作对比,如果当前更好,那就将它作为目前的最好位置
;
4) 比较当前每个粒子的适应度与其全局经历过的最好位置的适应度作比较,如果当前的更好,那就将它作为当前的全局最优位置
;
5) 更新每个粒子的速度和位置;
6) 如果达到了设定的最大迭代次数1000或最小误差1e−8,就输出解,否则返回步骤2。
当迭代次数达到300次左右时,达到函数容忍度1e−8,结束寻优过程,此时目标函数达到最小值,取反得net(X)的最大值。此时X取值即为20个分子描述符的最优值。目标函数的寻优过程图如图8所示,使用粒子群算法寻优得到的各分子描述符最终结果如表3所示。
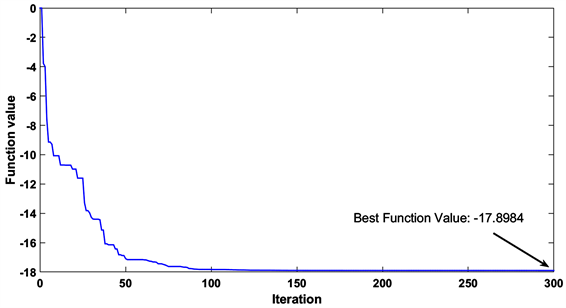
Figure 8. Objective function optimization process diagram
图8. 目标函数寻优过程图

Table 3. Molecular descriptors and their values
表3. 分子描述符及其取值
5. 结论与建议
本文根据ERα拮抗剂信息,采用了神经网络和二次SVM方法构建化合物生物活性的定量预测模型和ADMET性质的分类预测模型,为同时优化ERα拮抗剂的生物活性和ADMET性质提供预测服务,并且采用粒子群寻优算法进行多目标参数优化,寻找全局最优的pIC50值及其对应的分子描述符。主要得到以下结论:
1) 从729个分子描述符中筛选出了前20个对生物活性最具有显著影响的分子描述符。分别是MDEC-23、MDEN-23、ETA_BetaP_s等。
2) 对于大数据样本,基于神经网络建立的ERα生物活性的定量预测模型的预测值与真实值平均误差为0.07左右,该预测模型能够较为准确地对化合物的pIC50值进行预测。
3) 构建的ADMET性质的分类预测模型预测准确度达到92%左右,能够较准确地对化合物ADMET性质进行分类。本文采用的二次SVM方法构建的分类模型比常用的神经网络二分类和逻辑回归二分类准确度更高,具备一定的优势。
4) 在保证5种ADMET性质至少3个良好的前提下,pIC50值最大可达到17.8984左右,同时可得出与之密切相关的分子描述符的具体取值。
在后续的治疗乳腺癌药物筛选中,可使用参数更丰富的样本集对预测模型进行学习训练,不断优化预测模型,提高模型的准确度和可靠性。同时本文采用的方法和确定的数学模型为同类定量预测和分类预测问题提供了参考。
NOTES
*通讯作者。