摘要: 目的:以草菇为研究对象,对担子菌mating-type同源序列中的HD位点为靶标进行生物信息学初步分析。方法:应用在线生物信息学软件预测HD序列的理化性质、疏水性/亲水性、导肽、跨膜结构、三维结构、功能结构域,并构建了同亚门中其他物种该蛋白家族的系统进化树。结果:同源mating-type序列中HD基因位点的氨基酸不稳定性指数在56%左右,且无明显的信号肽、导肽和跨膜结构域,三维结构由3个α-螺旋和少数转角二级结构组成,七种食用菌在氨基酸序列呈现较低的相似性。结论:同源mating-type序列的HD1、HD2蛋白稳定性差,无信号肽、导肽、跨膜结构,无β-折叠,在N-J进化树中不同物种间该氨基酸序列相似性极低,表明HD位点具有丰富的多样性,且结构性简单。
Abstract:
Objective: The objective is to take Volvariella volvacea as the research object, and conduct a preliminary bioinformatics analysis on the HD site in the mating-type homologous sequence of Basidiomycetes. Methods: Using online bioinformatics software to predict the physical and chemical properties, hydrophobicity/hydrophilicity, guide peptide, transmembrane structure, three-dimensional structure, and functional domains of HD sequences, and construct a system of this protein family of other species in the same subphylum evolutionary tree. Results: The amino acid instability index of the HD locus in the homologous mating-type sequence is about 56%, and there is no obvious signal peptide, guide peptide, and transmembrane domain. The three-dimensional structure consists of 3 α-helices and a few turns in terms of secondary structure, and the seven edible fungi show low similarity in amino acid sequence. Conclusion: The HD1 and HD2 proteins with homologous mating-type sequences have poor stability, no signal peptide, guide peptide, transmembrane structure, and no β-sheet. The amino acid sequence similarity between different species in the N-J evolutionary tree is extremely low, indicating that HD sites are rich in diversity and simple in structure.
1. 引言
多数常见的食用菌都属于担子菌,如香菇、金针菇、糙皮侧耳、双孢蘑菇、草菇等,常见担子菌类食用菌的交配型系统都非常复杂,具有很多独特的生物学特征 [1]。担子菌的有性生殖方式可分成异宗配合和同宗配合两大类别,90%的担子菌属于异宗配合,其余10%属于同宗配合 [2]。异宗配合又可分为四极性和二极性交配型系统,其区别在于是由一个还是两个交配型位点而决定。四极性交配系统包含两个不连锁的交配型位点,通常称之为A位点和B位点,近年来,又被多学者建议根据交配型位点中基因的功能来命名这两个位点,即HD位点和PR位点 [3] [4] [5]。在典型的担子菌A交配型位点包含两类等位基因,因为可以编码含有一个嵌入同源结构域基序的同源域转录因子蛋白(HD蛋白),通常分别命名为HD1和HD2基因 [6]。HD蛋白序列中通常存在同源结构域、核定位信号序列等典型特征。使HD蛋白具有转录因子的功能,并在DNA双螺旋的大沟槽完成主要的蛋白质-DNA互作反应 [7] [8]。若能全面和深入了解交配型系统的分子结构,可有助于我们阐明交配型基因对子实体发育的调控及育种的相关科学问题。
目前,生物基因组序列数据正以爆发式增长,如何对这些序列数据进行科学的收集、管理和挖掘已经成为了研究的热点。随着生物信息学的发展,用生物软件预测蛋白质的结构变成现实,也成为一种趋势。本文利用比较基因组学和生物信息学的方法以草菇(Volvariella volvacea (Bull.) Singer)为研究对象,分析mating-type序列中HD位点的核苷酸序列以及所对应的氨基酸序列的理化性质、疏水性/亲水性、导肽、跨膜区域、二级结构、3D结构、功能结构域进行预测和分析,并与同亚门中不同物种的该蛋白质家族构建进化树,为下一步研究它在繁殖调控和开展生物大分子结构模型提供抛砖引玉的作用。
2. 材料与方法
2.1. 实验材料
因担子菌的有性生殖系统受交配型基因HD1和HD2调控 [2],根据担子菌基因的高度同源性,以草菇交配型基因序列进行分析。在NCBI (National Center for Biotechnology Information)数据库获得HD1、HD2的完整核苷酸及氨基酸序列(GI: 365940808, 365940807)。
2.2. 实验方法
本文在生物信息学分析中所需要应用的在线数据工具及其网址请参见表1。

Table 1. Website of bioinformatics online analysis tool
表1. 生物信息学在线分析工具网址
本文从数据库http://www.ncbi.nlm.nih.gov/ (NCBI)上阅读并下载HD1、HD2的核苷酸序列,同时也应用http://www.detaibio.com/tools/、http://www.cbs.dtu.dk/等网站提供的各类氨基酸在线生物软件工具及其他生物信息学软件进行了分析。蛋白质序列和理化结构性质的预测和分析主要应用于在线生物工具ProtParam;对氨基酸的同源比对及构建进化树的预测采用Mega 6.0软件,使用Bootstrap Replications算法,计算次数为1000;导肽的预测和分析主要应用于在线的工具TargetP 2.0;蛋白质序列的信号肽预测与分析应用SignalP 4.1工具;多肽链的亲水性与疏水性的预测和分析采用的工具为ProtScale;跨膜理化结构的预测和分析使用TMHMM Server 2.0;功能域的预测和分析使用NCBI数据库提供的CD-Serch Server工具;预测蛋白质3D结构用在线工具CPHmodels 3.2。
3. 结果与分析
3.1. HD1、HD2氨基酸序列的理化性质分析
利用在线工具ProtParam分析HD1、HD2氨基酸序列的特征。结果如表2,HD1中含有492个氨基酸,分子量达53.54 kDa,理论等电点(pI)为4.96;而HD2中含有404个氨基酸,分子量达46.20 kDa,理论等电点(pI)为9.73;HD1碱性氨基酸比例较多,HD2酸性氨基酸比例较多,两者蛋白质均不稳定,总平均疏水性数值中正值表示疏水性,负值表示亲水性,而数值介于−0.5~+0.5主要为两性氨基酸 [9],由上述序列特征可知HD1为两性氨基酸,HD2为疏水性氨基酸。
3.2. 信号肽的预测和分析
信号肽通常是位于氨基酸序列的N端,是一条短肽链,一般长度大约为5~30个氨基酸。信号肽的主要功能和作用之一是有效地促进细胞合成的蛋白质分泌转移到胞外,它在完成信号指引后会被细胞切除成为成熟蛋白 [10]。利用在线分析工具SignalP 4.1分别分析了HD1、HD2的N端的短肽链。每个位点中的氨基酸对应1个S值;C值为原始剪切位点分值;Y值则是综合考虑信号肽的S值和C值的一个重要参数,通常S值在陡峭区域且具有高C值的位点即为剪切点。由图1可以看出,HD1、HD2均无信号肽。

Table 2. Amino acid sequence composition and physicochemical properties of HD1 and HD2 genes
表2. HD1、HD2基因的氨基酸序列组成及理化性质

Figure 1. Prediction and analysis of HD1 (left) and HD2 (right) signal peptides
图1. HD1 (左)和HD2 (右)信号肽预测与分析
3.3. 导肽的预测和分析
导肽是通过引导肽链转运定位到对应细胞器的一段标签功能性序列,位于肽链N端,有助于蛋白质的亚细胞进行定位 [11]。本文利用最新的在线软件TargetP 2.0分析了HD1、HD2的氨基酸序列。序列含有线粒体目标肽(mTP)及分泌途径信号肽(SP)的分值如下表3,Loc表示所预测最有可能的细胞器定位,其中“_”表示除线粒体和细胞周质两处外的其他位置,即为定位到其他细胞器。RC代表的数值是可靠性的级别;TPlen可预测到剪切位点序列与氨基酸的个数;在表3中HD1、HD2预测剪切位点序列为均0个氨基酸,表示无氨基酸分裂位点。因此,HD1、HD2可能不具有导肽,且预测的可靠性级别较高。

Table 3. Prediction and analysis of amino acid sequence guide peptides of HD1 and HD2
表3. HD1、HD2的氨基酸序列导肽预测与分析
3.4. 跨膜结构域预测
跨膜结构的区域一般包含20~25个左右的疏水氨基酸及其残基形成的α-螺旋结构,在细胞磷脂膜双分子层中形成的跨膜通道 [12]。通过在线工具TMHMM Server 2.0对HD1、HD2进行预测,预测结果如下图2所示,HD1、HD2两者均没有明显的跨膜结构域。
Figure 2. Prediction of HD1 (top) and HD2 (bottom) transmembrane domains
图2. HD1 (上)和HD2 (下)跨膜结构域的预测
3.5. 疏水性与亲水性的预测和分析
蛋白质中间的疏水基团在维持整个蛋白质构象和折叠中发挥了很好的作用。根据疏水性或亲水性氨基酸的数值,可以推测整个蛋白质分子表面的疏水性或亲水性氨基酸残基的分布 [13]。利用在线的工具ProtScale对HD1、HD2进行分析,结果如下图3,HD1在492个多肽氨基酸中,亲水性与疏水性的氨基酸均匀分布,整个肽链表现为两性氨基酸。HD2在404个多肽氨基酸中,整体疏水性氨基酸较多呈现疏水性,该阶段HD1、HD2所预测的结果与3.1中总平均疏水性指数的结论相吻合。
3.6. 功能域的预测和分析
功能域是蛋白质三级结构中发挥功能的关键区域,是相对独立和保守的功能结构单位 [14]。利用在线工具Conserved Domain Search service分析HD1、HD2的氨基酸序列,搜索匹配结果(图4),在HD1中具有一个特定匹配(specific hits) Homeobox_KN域,是homeodomain超级家族中独有的成员,也是同源域转录因子蛋白的保守域。在HD2结果中(图4)发现具有一个特定匹配(specific hits homeodomain)同源异型结构域,属于homeodomain超级家族的成员,同源异型结构域是结合DNA序列的区域,同源结构域的存在可以表明HD蛋白具有转录因子的功能。

Figure 3. Prediction of hydrophilicity or hydrophobicity for HD1 (left) and HD2 (right)
图3. HD1 (左)和HD2 (右)亲水性或疏水性的预测
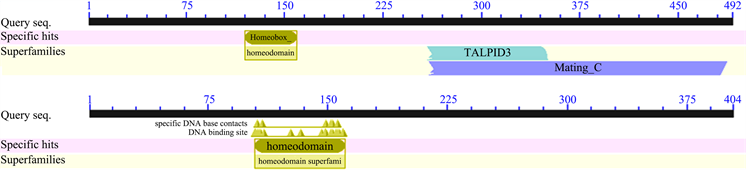
Figure 4. Prediction of HD1 (top) and HD2 (bottom) functional domains
图4. HD1 (上) HD2 (下)功能域的预测
3.7. 三维结构的预测和分析
蛋白质和酶只有在形成三级空间结构时,才更加接近天然构象,才能充分发挥生理活性。利用在线工具CPHmodels 3.2可以对现有多肽链进行3D结构预测。由下图5可知,HD1预测的三级蛋白结构由3个α-螺旋、1个转角结构,看上去是由3个松散泡影组成的正U型三维结构,含有3个脯氨酸(红色分子球)。HD2预测的三级蛋白结构由3个α-螺旋、3个转角结构,看上去是由5个松散泡影的二级结构组成的扭曲型团块三维结构,含有3个脯氨酸(红色分子球)。HD1、HD2同预测整体三维结构组成并不复杂且无β-折叠。可直观地观察到HD1、HD2两种蛋白性质不稳定这与3.1中蛋白质不稳定性指数的结论相吻合。

Figure 5. 3D structure of HD1 (left) and HD2 (right) amino acids
图5. HD1 (左)和HD2 (右)氨基酸三维结构图
3.8. 交配型基因的系统进化分析
将交配型基因编码的蛋白序列与其他同属担子菌亚门的6种食用真菌中已经鉴定的交配型基因编码的蛋白序列进行多重序列比对,登录NCBI下载各个真菌序列。其真菌GI登记号及真菌名称如下表4。

Table 4. Detailed list of protein sequences encoded by seven basidiomycete mating-type genes
表4. 七种担子菌交配型基因编码的蛋白序列详细表
利用MEGA 6.0软件对7种担子菌进行氨基酸序列比对,去除两端不齐序列,选择可视化的N-J树项,采用Bootstrap Replications算法,计算次数为1000,其他参数默认,分别构建HD1、HD2基因的有根系统发育树,得到如图6所示。

Figure 6. Phylogenetic tree of HD1 and HD2 amino acid sequences of seven basidiomycetes
图6. 七种担子菌HD1、HD2氨基酸序列的系统进化树
不同真菌的HD同源蛋白聚类,HD1、HD2明显可以分成两大群组,且金线菌属的金针菇HD1序列似乎脱离了HD1群,从而确定了树根。这与学者Van Peer表明金针菇HD-α亚位点的功能可能已经退化,不再具有调控交配过程的功能描述相符合 [15]。HD1群其余成员可以分为三小组香菇属为组I,鬼伞属和侧耳属为组II,蘑菇属、裂褶菌属和小包脚菇属为组III。HD2群可区分的小组较分散,各成员间遗传差异偏大。可见HD蛋白的遗传多样性较高。
4. 讨论
在大多数真菌中,无论是在子囊菌还是担子菌中均可探索到交配型基因的身影,近些年来大多数食用真菌和药用真菌的遗传育种研究开始泛热,研究人员们把在研究的目光逐渐投向了基因分子水平上,而在担子菌的异宗配合根据交配型系统中HD蛋白具有转录因子的功能,并可完成主要的蛋白质-DNA互作反应,在遗传和繁育有着一定影响力 [16]。本文从生物信息学的角度,以草菇作为主要研究对象,对其HD1、HD2基因的氨基酸序列进行理化性质、功能域作用、结构组成、细胞器定位和三维结构等预测和分析。本文预测的结果显示HD1、HD2两者均是一种不稳定性质的蛋白质,无信号肽、导肽及稳定的磷脂双分子层的跨膜结构,从氨基酸三维结构预测中可看到,HD1蛋白呈现正U形状,HD2蛋白为扭曲团块形状,从形态上HD2较比HD1相对保守,这可能与HD2螺旋III包含一个典型的、高度保守的DNA结合基序WFXNXR [17] 有关,另一方面,HD1蛋白螺旋III的DNA结合基序中的氨基酸序列具有更高的多态性,HD1蛋白较HD2蛋白更容易在DNA结合基序中发生DNA突变 [18] [19] [20]。同源结构域的氨基末端区域负责区分亲和与不亲和的相互作用,这个识别域内的关键残基的取代能够影响HD蛋白的特异性,若能对HD蛋白的关键残基进行一定的修饰或者调控,是否能对担子菌的有性繁殖过程起到深层次的认识及推动作用,其相关性研究还需待进一步研究。
NOTES
*通讯作者。