1. 引言
肾脏是人体内很重要的一个器官,它的主要作用是清除一些代谢废物,并且重吸收一些有用的物质和水分。肾实质病变是肾疾病中比较常见的病例,如果在小儿早期及早发现,就可以极大的预防肾实质的恶化,所以研究肾图具有重要意义。我们现在临床上主要是通过静脉注射荧光标记物示踪,然后经过肾图仪器监控,得到左右两肾的医学影像。随后通过临床医生的经验,对得到的影像进行标注,并对肾的健康与否做出诊断。医学影像的手工标注是十分精细的一份工作,一个具有丰富经验的医学影像科医生对于肾图的标注速度大约在二十至三十张左右每天,而且医生人工标注的精细程度也会随着工作量的增加而变差,显然这样的效率与精度是满足不了我们的使用的。下图1所展示的为小儿肾图的原始图像,从图像我们可以明显的看出边界是十分模糊的,手工的标注的难度还是十分大的。
医学图像分割就是对于医学图像中感兴趣对象的每个像素进行标记的任务,它通常是临床应用的关键任务。随着深度学习的发展,计算机辅助诊断系统 [1] [2] (computer aided diagnosis, CAD)开始逐渐走进医学图像领域。分割精度和不确定性的量化对于评估其它应用程序的性能至关重要,现阶段医学图像分割的关键挑战是缺乏大量注释、缺乏用于训练 [3] 的高质量标记图像、图像质量低、缺乏标准分割协议,以及患者与 [4] 患者之间的图像变化较大。也因此我们需要一种自动化的、可泛化的、高效的语义图像分割方法。卷积神经网络(CNN)在2012年的图像处理中显示出卓越的性能 [5]。在2014年CNN开始应用于图像分割领域,Jonathan Long等人 [6] 提出的全卷积神经网络(FCN),也是历史上首次把CNN解码部分由原来的全连接层转换成为卷积层,为图像的语义分割开启新的纪元,并且为图像分割提供了新的解决方案。同年U-Net [7] 更加精细的扩充了FCN网络的解码部分,构造了一种新型的端到端训练像素级预测图像分割架构,也正是这种编解码的结构为医学图像分割任务开启了新的篇章,为医学图像语义分割领域做出了重要的贡献。
医学图像分割有助于临床医生对于疾病的辅助诊断,可以更加便捷高效的提取详细的信息,以获得更快更精确的诊断。正是这种优越的特性,帮助医生快速定位病灶区域,彻底解放了医生的双手,让医生可以把精力投入到相关病情分析与诊断中,得到了医学界的广泛青睐,为患者和医生之间搭建了诊断的桥梁。所以医学图像分割的应用将会给医学界带来极大地便利,但是对于肾图 [8] 分割还是一个未被广泛研究的领域。
目前市面上对于肾实质的标注都是基于临床医生手动标注,没有一种肾实质自动化标注的方法,所以亟需一种有效的方法来解决这个问题。当卷积神经网络在图像领域独据一方时,transform在自然语言处理领域同样占据着主导地位,正是这两种各具特色的结构,所以现在研究人员都在研究着把这两种结构结合起来以观察结果。也正是受到这种启发,本文在U-Net [7] 的基础上把它的编码层用transform架构替代,提高了肾实质标注的精度,同时降低了医生用于肾图标注的成本,使得诊断的速度大幅度的提升,帮助医生对患者进行更加准确的处理患者肾病灶区。最后我们通过实验与一些比较流行的方法(FCN [6]、U-Net [7]、Deeplab-v3+ [9]、SegNet [10] )。
本文的主要贡献如下:
1) 在医院提供的小儿肾图的基础上,对图像做了预处理,并制作了小儿肾实质的数据集。
2) 提出了基于transform的编码模块,使网络的解码部分由传统的卷积模块改变为新型的结构,提高了模型分割精度。
3) 提出基于transform与卷积神经网络(CNN)结合的新型网络该网络,并将该结构用于语义分割。本网络改变了传统编解码网络并将解码部分用transform代替,实现了与U-Net [7] 网络的首次结合,并且首次将图像分割任务运用在肾图数据集上。
2. 相关工作
2.1. 图像分割网络
语义分割在当今的计算机视觉领域占有重要的地位 [11],从现实意义上讲,语义分割是一项高层次的分割任务,而这其中的图片解析作为计算机视觉领域的一个核心问题,为越来越多通过图像来获得信息的应用提供理论基础。就比如当下很热门的自动驾驶汽车、人机交互、虚拟现实和医学图像分割的任务等等,就很流行用语义分割网络架构来解决这些问题。图像的语义分割任务通俗的理解就是将图像的每个像素进行分类,分为不同实体,其中的每个实例都对应分割的一个类。这个任务目的是为了更好地解释图像的全局上下文,它是场景理解概念的一部分。在医学图像分析领域,医学图像的语义分割可以用于图像的引导治疗、放射治疗或改进的放射诊断。不过,由于当前医学图像数据量小,人工标注成本费时费力,目前大多数研究都是基于某个器官,建立相应的网络架构,然后通过改进网络的模块来提高分割精度。
首先采用语义分割的是2014年Long J等人提出的FCN [6],它是语义分割的开山之作。这也是首次提出的一种端对端的分割网络,也是自全卷积网络(FCN)开始,语义分割不在使用之前精度很低的TextonForest和基于随机森林分类器等方法,从而开始使用基于卷积的编解码结构。从而开始使用基于卷积的编解码结构。随后,Olaf Ronneberger等人在2015年提出的有着“医学影像分割基石”之称的U-Net [7],U-Net网络的提出更是大大提升了分割精度,并且为后来新的网络的革新提供了思路。同年,Badrinarayanan V [10] 等人提出了在语义分割领域也有开山之作的轻量级网络SegNet,该文章提出了经典的SE-Block模块以及编码–解码的思想影响着后来的很多模型。随后就是Liang-Chieh Chen [9] [12] 等人提出的Deeplab其衍生网络,在DeepLabv3+中,使用了encoder-decoder结构和空间金字塔池化模块,进一步的探索改进Xception和深度分离卷积,提升了模型在语义分割任务上的性能,提出一种新型的网络框架。并且该网络具有一定的鲁棒性,为后来网络的性能比较提供了一个很好地标杆。本文就是借鉴了医学影像基石U-Net网络的设计思想,并结合了当下最热门的结构transform所形成的新型网络。
2.2. Transform模块
众所周知,上下文信息是提升语义分割性能的关键因素,而感受野的大小就大致决定了网络可以利用多少有用的信息。理论上,通过堆叠足够深的卷积层,网络的感受野便能够覆盖到输入图像的全局领域,然而何凯明等人通过在文章Resnet [13] 实验发现,当残差网络达到150多层,这时候再下采样,识别精度不提升反而下降。这也表明,过多的下采样操作会导致目标的一些边缘细节信息缺失,甚至会完全丢失,从另外一方面我们也可以知道此时网络实际的感受野会远远小于其理论的感受野。对于这个问题,现在主流的解决方法主要有两种,第一种是改进原始的卷积操作,通过提取更多的目标像素从而捕获更多的上下文信息。如Deeplab-v3+ [9] 网络通过使用atrous/dialted convolution (过建立spatial attention (空间注意力)模块提升感受野大小;CCNet [14] 通过建立 self-attention (自注意力)模块提升感受野的大小。但是这两种方法其间都穿插了卷积,没有摆脱传统的卷积结构,此外卷积中引入注意力机制会势必会损失一部分感受野,这意味着缺乏对低级特征使用,导致表示学习结果不是最优,这样的下采样操作势必会降低网络感受野提取空洞卷积)提升感受野的大小;再比如PSPNet [15] 网络通过image/feature pyramid (特征金字塔)模块提升感受野的大小。第二种方法是网络中引入attention (注意力机制),通过从不同维度建立长距离的依赖从而捕获全局的上下文信息,提升感受野大小。如PSANet [16] 网络通过建立PSA结构已经在自然语言处理领域的很多任务中取得了非常不错的效果,DANet [17] 网络通的信息,也出于这样的考虑,transform应运而生。2017年谷歌实验室Juraj Juraska等人写的一篇名为《Attention Is All You Need》 [18] 的论文,提出了一种基于attention (注意力机制)的结构来处理序列相关问题的一种新模型,并且命名为transformer。它在解决文本分类、机器翻译,阅读理解等任务时,该模型可以高度同时高效的工作,训练速度十分快,此外该模型可以自适应的捕捉输入序列在不同位置之间的相对关联,很擅长处理长文本。transformer摒弃了卷积神经网络(CNN)和循环神经网络(RNN)架构的固定形式,它虽然不具备平移不变性和局部性,但是Transform不像卷积操作那样有固定且有有限的感受野,它的核心self-attention可以直接提取整体的感受野。也正是这样丰富的感受野,以及它在各项挑战赛中优异的性能,吸引了很多图像领域的研究学者。
受到transformer网络 [18] 以及 [19] 中transform结构的启发,本文提出了一种新的架构,它的编码部分不再像传统的卷积网络,而是采用了transform模块,这样本网络就能在编码层提取到图片全部的上下文的语义信息,从而不丢失感受野,保留图片每一帧的细节信息。我们通过在肾图数据集上充分验证了本网络的有效性。
3. 网络结构
在传统的网络中,解码部分是由一层一层的卷积进行下采样的,然而根据军妓提取特征的机理,随着卷积层数逐渐的增多,虽然特征图包含的类别信息逐渐增多,但是相应的一些细节和边界信息会减少。在医学上,有时候一些边缘细节信息很重要,甚至会直接影响到评价结果,所以我们需要重视这些细节与边缘信息,也是出于这种目的,编码部分用了新型的一种从序列到序列角度的架构——transformer,而抛弃了传统的卷积结构。下面图2是网络结构。
(图片2流程解析:首先把输入的每张图片均分成9小块,提升数据处理的效率,随后通过展平处理把二维的图片转换成的一维的序列,并且嵌入position encoding,对不同的位置进行一定程度的编码,这样得到的每一个词,在不同位置的编码会有所不同,也就可以更好地得到位置信息的相关性。随后通过transformer编码层提取特征信息,并传入解码层上采样得到相应的掩码图。)
程序开始时,由于图片是二维的,我们需要对图片进行预处理,通过一种算法把一个二维的图片转换成一维的序列,并且把这个图片均分成9份,然后记录下它对应的位置信息,并且把这些信息输入到下面的编码层。
3.1. Transformer编码层
以一维序列作为输入,采用基于纯transformer的编码器来学习特征,这也就意味着每个变压器层都有一个全局的接受域,这样就可以解决使用传统卷积操作会瞬时部分感受野的问题。Transformer编码层主要由有多头自注意力(Multi-Head Self-attention) (MSA)、多层感知器(MLP)和layer norm层(LN)组成。图3是编码层transformer的示意图。
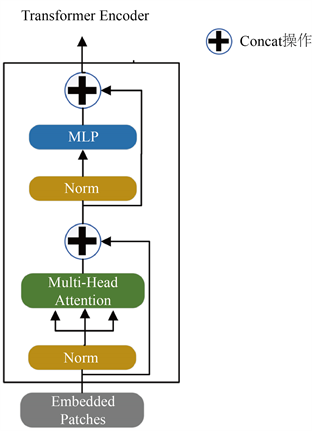
Figure 3. Transformer schematic diagram
图3. Transformer示意图
本文中的transformer层,每层输出结果即{
},多头注意力机制(Multi-Head attention)的输入是多个自注意力相关的运算,一组(q, k, v)矩阵经过一系列的运算即可代表一个SA (Self-attention)即自注意力的运算,将这多个矩阵拼接起来后再乘以一个参数矩阵
,即可得到最终的输出。
三元组(q, k, v)的计算公式如公式(1),其中
是随机矩阵,L代表输入的层数,
代表L行C列的矩阵,L代表层数,C代表列数,
代表参数矩阵。
(1)
自注意力SA(Self-attention)计算公式如公式(2)
(2)
多头注意力机制(Multi-Head Self-attention)计算公式如公式(3)
(3)
L层的最终输出结果如公式(4)
(4)
3.2. 解码层(decoder)
我们把前面transformer编码层得到的特征图大小进行调整,随后通过4层上采样,并且在每层上采样中加入跳跃连接来补充丰富上采样过程中的语义信息,然后把得到的特征信息进行融合,最后通过双线性插值恢复至原图片大小,下面图4是我们解码层的结构示意图。

Figure 4. Schematic diagram of decoding layer structure
图4. 解码层结构示意图
4. 实验结果与分析
选取了语义分割领域经典的深度神经网络FCN、SegNet、U-Net和Deeplab-V3+与本文提出的网络分别在我们制作的肾图数据集上做了对比实验,以下介绍具体实验过程和实验结果。
4.1. 实验软硬件环境
本文进行试验所使用的实验平台如下表1所示:
4.2. 小儿肾图数据集介绍与预处理
本文中的肾实质数据是由上海交通大学医学院附属新华医院影像科提供的原始肾图共(700)套,我们在提供的原始数据上进行了预处理,它的基本原理是静脉注射能被肾实质摄取,然后迅速随尿液排出的显影剂,通过肾图仪进行采集。正如我们图一所看到的,陨石肾图仪所形成的图片是很模糊不清的灰度图,所以就需要我们实验前预先处理下数据。每个人通过肾图仪能得到大概136张图片。肾实质数据集是在专业影像科医生的指导下进行的,在数据整理过程中,由于刚开始显影剂没到达指定位置,所以我们除去前一分钟的所采集的灰度图。然后我们对随后的灰度图片,每七张按时间顺序进行融合。一般一个人能融合得到11张可用于标记的图片。随后我们对正11张图片利用OpenCV进行伪彩色处理,生成的伪彩色的图片经更有利于我们的标记和训练。我们把每组图片中的第2张和第三张进行图片标注,并且把原图片和标签收录到我们收集的数据集中,通过这几步操作,我们一共得到了肾实质数据集(共700张),随后我们对得到的数据集进行适当的剪裁,最终得到的图片像素大小为384 * 384。具体数据集制作流程如下图5所示:
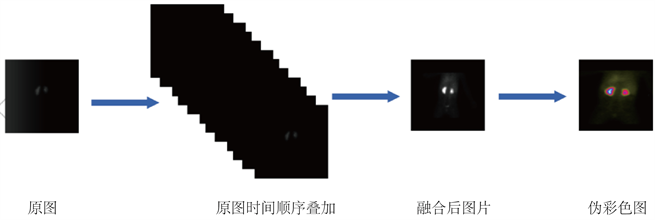
Figure 5. Renal parenchyma data set production process
图5. 肾实质数据集制作过程
4.3. 损失函数与评价指标
实验采用语义分割领域常用的评价标准:以Precision (精确率)、Dice_coeff (集合相似性)、Recall (回归率)为评价指标对本文提出的模型进行评价。
本文使用的损失函数为经典的交叉熵损失函数,函数公式如下所示:
(5)
其中,
表示真实标签类别,当n为变化类像素时
取值为1,否则,
取值为0;
代表预测n为变化类像素的概率且
。N是一个样本中总的像素数,n是样本中一个像素。
图像分割任务是对每个像素进行的分类预测,通常是可以通过从模型预测的混淆矩阵中获取四个基本指标,他们分别是真阳性(True Positive, TP),假阳性(False Positive, FP),真阴性(False Negative, TN),以及假阴性(True Negative, FN),然后通过基本的指标运算,就可以得到Precision (精确率)、Dice_coeff (集合相似性)、Recall (回归率)、正确率(Accuracy)、Jaccard系数这几个衡量参数的指标。其中正确率为正确预测的样本数占总预测样本数的比值;精确率Precision为正确预测的正样本数占所有预测为正样本的数量的比值;回归率Recall为正确预测的正样本数占真实正样本总数的比值;集合相似性系数Dice_coeff用于描述模型预测出的预测值与真实值的相似性,Dice系数为2倍预测结果与样本标签交集与其和的比,Jaccard系数为预测结果与样本标签的交并比。这几个衡量指标的计算公式为:
(6)
(7)
(8)
(9)
(10)
实验中我们设置的batch-size为8,训练超参数学习率设置为0.002,训练迭代次数设置为最多1000个epoch,这是一个考虑到医学数据集数据量比较小的防过拟合的机制。即当没有达到最优的精度(最佳权重),会一直训练,最多迭代到1000次,训练结束;当在1000次内的某一次达到最佳精度时,他会继续再迭代100个epoch,如果最佳还是100个批次前的那个精度,则保存这个最佳精度。这样做可以既可以有效地防止数据量过小导致实验结果不精确,又能防止训练次数过多所导致过拟合。
4.4. 实验结果
实验过程中,我们对本文制作的700张伪彩色图片进行随机取样,其中取五分之四的图片作为训练集,剩下的作为测试集。下面具体介绍所做的实验。
传统网络对比实验:为了验证网络的效果,在本实验中,我们比较了五种不同网络对于肾图数据集的分割性能,包括FCN,SegNet,U-Net,Deeplab-v3+和本文提出的网络,实验结果见表2:

Table 2. Comparison of different network segmentation effects
表2. 不同网络分割效果对比
由实验结果得知,使用本文提出的网络进行肾图分割,得到的分割结果在precision、recall和dice_coeff上均优于运用其余四种经典网络得到的分割结果,其中比U-Net在dice_coeff方面高出4.1%,在precision方面高出2.5%,在recall上也提升了2.54%,总体效果提升明显。
由图6不同网络分割效果对比图也可以很明显的看出,本文所提出的网络相比其他四种网络分割效果要更加好,尤其体现在边界细节方面。
随后,针对肾实质数据集数据量比较小的缺点,我们把所得到的数据集进行sample pairing数据增强处理,即对现有的图像进行随机翻转、对称,最终得到了2520张图片,然后我们用这2520张图片作为训练集,验证集和测试集不变,下面是用数据增强后的数据训练的结果。(带*即为使用数据增强后的网络)

Figure 6. Comparison of different network segmentation effects
图6. 不同网络分割效果对比图

Table 3. Comparison of segmentation effects of networks after data enhancement
表3. 数据增强后网络分割效果对比

Table 4. Comparison of segmentation effects of networks after data enhancement
表4. 数据增强后网络分割效果对比
从表3和表4五个指标的数据结果可以看出,数据增强后测试集的精度并没有我们实际猜想中的那么高,我们猜想可能是过多数据导致的过拟合引起的精度下降。因为就类似FCN,U-Net,他们都是轻量级的网络,经过数据增强后的数据,很多图片经过了翻转、对称等操作,这时针对不同肾图上的细节特点,在训练的时候被倍数放大,导致与测试集过度拟合,然后导致了测试精度相比起原数据集的测试精度比较差的结果。
5. 结论
本文提出了一种新型肾实质分割网络。该网络结合当下流行的transform模块与当下最流行的语义分割网络,也是首次将transform型的语义分割网络应用在肾图数据集上,也正是transform模块优越的提取特征的特点,使得该模型很适合肾图数据集的肾病变区域分割,最终得到的特征图包含了图像的各个尺度特征,充分补充了网络的一些细节信息。实验结果表明,所提出的语义分割网络优于其他网络,并且本网络有效的减小了卷积操作产生的语义间隙,提升了网络的感受野,提高了网络图像分割的效果。减轻了医生的工作量,在辅助治疗领域具有很好地研究价值。