1. 引言
胎盘对哺乳动物胚胎的存活和发育有着重要的作用,子宫内胎儿独特的生长潜能是由怀孕期的胎盘决定的,胎盘是母体与胎儿营养物质和代谢物质交换的重要场所,具有免疫学和内分泌的功能 [1]。研究共识高效率胎盘被认为是较小的胎盘能维持相对较大的胎儿正常生长,益于子宫容量和幼崽大小,相反,低效率胎盘导致胚胎流产、死亡、出生胎儿低重和胎儿断奶前死亡 [2]。因此,胎盘功能遗传机制的揭示,将更好地调控宫内胎儿的生长发育,为提高哺乳动物繁殖率奠定理论基础。
大量的印记基因以剂量敏感的方式调节胎儿生长和胎盘发育,这种调控方式至关重要 [3]。印记基因特点是源于亲本等位基因的单一表达,它能影响母体与胎儿之间营养的分配和胎盘响应外界环境改变时营养供给的变化 [4]。营养假说认为父本等位基因的表达促进胎儿的生长,确保父本基因组成功的遗传到下一代,反之,母本等位基因的表达抑制胎儿的生长,有利于自身的生存和繁殖 [3] [4]。另外,发现印记基因还参与到其它的过程途径调控,包括温度调控、哺乳行为、糖和脂肪代谢调控,对出生后幼崽的生存和发育起到相当重要的作用 [3]。
研究报道,PHLDA2 (pleckstrin homology-like domain, family A, member 2)在人和鼠中被证实为胎盘生长和分化的变阻器,属于母本表达印记基因,分别位于人、小鼠的11p15和7号染色体,大量表达于胎盘滋养细胞层细胞,维持胎盘功能的稳定和胎儿的生长发育 [5] [6]。大量研究人胎儿生长受限(Fetal growth restriction, FGR)的胎盘中,发现PHLDA2异常的升高表达和出生后体重急剧增加的现象,表明PHLDA2异常表达影响胎盘功能 [7] [8]。同样,过表达Phlda2的小鼠,表现明显的胎盘萎缩和FGR,相反,敲除phlda2的小鼠,胎盘过度生长 [9] [10]。尤为重要的是Phlda2能刺激小鼠胎盘糖原的储存,其功能的丢失和获得导致海绵体滋养层发育异常 [6] [11]。PHLDA2母源等位基因表达方式在人、鼠、牛、猪、山羊中也得到证实 [5] [12] [13] [14]。
DNA甲基化是DNA复制后调控方式之一,是一种表观遗传修饰,通过改变基因启动子区域的DNA甲基化频率来影响染色质结构,DNA稳定性以及转录因子与DNA相互作用等,从而导致基因转录水平的变化。DNA甲基化在甲基转移酶作用下,使DNA序列中CpG位点的胞嘧啶变为5-甲基胞嘧啶 [15] [16] [17] [18]。如果一个特定的序列中含有大量的CpG二核苷酸重复序列,这个地区被称为CpG岛,CpG岛为约100~200 bp或几千个碱基,将近60%的基因通过DNA的甲基化来改变基因的染色质结构调控表达和转录 [16]。如果特定的基因启动子区域CpG岛被甲基化,该基因的表达可能被下调或转录沉默。相反,如果启动子区域CpG的岛未被甲基化,该基因通常高度表达,这也是基因组印记表达方式的调控机制之一 [19] [20]。
近年来,PHLDA2基因启动子片段相继在人、牛中被克隆 [21] [22]。有关山羊PHLDA2基因转录调控机制研究的报道尚少。本研究旨在克隆山羊PHLDA2启动子,分析山羊胎盘、心脏、脾脏和肺脏等各组织中的PHLDA2启动子区域甲基化频率对其转录水平的影响,为探明PHLDA2基因对山羊胎盘、心脏和脾脏等组织发育的调控机制提供分子依据。
2. 材料
2.1. 样品
山羊来自西南大学实验羊场。分别采集出生3 d仔黑山羊的9种组织:心脏、肝脏、脾脏、肺脏、肾脏、肌肉、脂肪、舌、大脑,另采集孕产期胎盘组织,屠宰后迅速采集样品并放置于液氮中暂存,后转移至−80℃保存备用。
2.2. 主要试剂
RNAisoTM Plus、PrimeScript@RT reagent Kit with gDNA Eraser、DL-2000 Marker、TaKaRa Ex Taq® DNA Polymerase、PMD19-T Vector Cloning Kit,均购自宝生物工程(大连)有限公司;琼脂糖、Gold view,均购自北京鼎国生物有限公司;E. coli DH5α感受态细胞,购自北京擎科生物科技有限公司;Gel Extraction Kit (100) D2500-01,购自OMEGA bio-tex公司;EZ DNA methylation gold Kit,购自北京Zymo公司。
2.3. 主要仪器
PCR仪(型号为T100 BIO-RAD)、凝胶成像系统(型号为ChemiDoc XRS+),均购自Bio-Rad公司;核酸蛋白检测仪(型号为Thermo Scientific NanoDrop 2000),购自美国Thermo Fisher公司;高速台式离心机(型号为5418R),购自德国Eppendorf AG公司;核酸电泳仪(型号为DYY-6B),购自北京六一生物科技有限公司。
3. 方法
3.1. 引物设计和合成
利用生物信息学方法,在GenBank检索同源性相近的牛PHLDA2基因启动子区域(登录号为NC_007330),根据保守性较高区域设计引物,上游引物为PHF1和PHF2,下游引物设计在已克隆山羊PHLDA2基因(登录号为KF994922.1)区域内,为PHR1和PHR2,其组织间表达引物为F3和R3,内参基因GAPDH引物为F4和R4,所有引物用Primer Preimer 5.0软件设计。根据已扩增得到的PHLDA2启动子序列和其基因序列,利用Methylation Primer Express v1.0软件设计启动子CpG岛引物MSPF1/MSPR1、USPF1/USPR1和第一外显子内的引物CpG岛引物MSPF2/MSPR2、MSPF2/MSPR2。引物委托北京华大基因公司合成,引物序列信息见表1。
3.2. 组织表达检测
取山羊心脏、肝脏、脾脏、肺脏、肾脏、肌肉、脂肪、舌、大脑和胎盘的组织各100 mg。
利用RNAisoTM Plus试剂盒提取RNA,使用10 g/L琼脂糖凝胶电泳及紫外分光光度计检测RNA浓度和纯度,参照反转录试剂盒(PrimeScript@RT reagent Kit with gDNA Eraser)说明书对总RNA进行反转录合成cDNA,置于−20℃保存,备用。以山羊cDNA为模板进行PCR扩增,PCR扩增体系:上下游引物各1 μL,模板cDNA 1 μL,10 × Ex buffer (Mg2+) 2.5 μL,dNTP Mixture (2.5 mM each) 2.5 μL,Ex Taq 0.25 μL,补充灭菌双蒸水至25 μL。PCR扩增程序:94℃预变性4 min;94℃变性30 s,60.0℃退火30 s,72℃延伸30 s,共35个循环;72℃再延伸2 min。PCR扩增产物通过1.5%琼脂糖凝胶电泳并凝胶成像系统拍照。
3.3. 启动子PCR扩增及测序分析
取山羊心脏、肝脏、脾脏、肺脏、肾脏、肌肉、脂肪、舌、大脑和胎盘的组织各100 mg采用常规的苯酚–氯仿抽提法各组织基因组DNA。使用核酸蛋白测定仪测定DNA样品浓度及纯度并进行凝胶电泳,最后将样品置−20℃冰箱中保存。采用巢氏PCR进行启动子扩增。PCR扩增体系:上下游引物各1 μL,模板DNA (25 ng) 1 μL,10 × Ex buffer (Mg2+) 2.5 μL,dNTP Mixture (2.5 mM each) 1.0 μL,Ex Taq 0.25 μL,补充灭菌双蒸水至25 μL。PCR扩增程序:94℃预变性4 min;94℃变性30 s,退火温度30 s (各退火温度见表1),72℃延伸2 min,共35个循环;72℃再延伸2 min。将第一次扩增PCR产物稀释50倍为模板,进行第二次PCR扩增,PCR扩增体系和扩增程序同第一次相同。第二次PCR扩增产物经1%琼脂糖电泳检测,拍照,并回收纯化以及连接转化到大肠杆菌DH5α感受态细胞中,将阳性克隆菌液送至北京华大基因公司测序。
3.4. PHLDA2启动子序列分析
对获得的启动子序列进行生物信息学分析,所用软件及其在线网址见表2。
3.5. 亚硫酸氢盐修饰基因组DNA及PCR扩增
严格按照DNA甲基化转化试剂盒说明书对样品各组织DNA进行亚硫酸氢盐修饰处理,再用核酸蛋白测定仪测定修饰处理后样品的浓度及纯度,最后置−80℃冰箱中保存,备用。PCR扩增体系:上下游引物各2 μL,模板DNA 2 μL,10 × Epi buffer (Mg2+) 5 μL,dNTP Mixture (2.5 mM each) 6 μL,Epi HS Taq 0.25 μL,补充灭菌双蒸水至50 μL。PCR扩增程序:94℃预变性4 min;94℃变性30 s,50.0℃退火30 s,72℃延伸30 s,共35个循环;72℃再延伸10 min。PCR扩增产物经2%琼脂糖电泳检测,拍照,并回收纯化以及连接转化到大肠杆菌DH5α感受态细胞中,分别挑取阳性菌落30组送至华大基因检测。甲基化频率:F = CpG位点胞嘧啶甲基化数 × 100%/CpG位点胞嘧啶总数。
3.6. 统计分析
运用Origin 8.0软件绘图,用SPSS16.0软件分析所得数据,当P < 0.05认为差异显著,P < 0.01表示差异极显著。
4. 结果与分析
4.1. 山羊PHLDA2基因组织表达分析
PCR扩增得到PHLDA2和GAPDH基因片段分别为159 bp和119 bp片段(见图1),表明PHLDA2基因在山羊的心脏、肝脏、脾脏、肺脏、肾脏、大脑、肌肉、脂肪、舌和胎盘组织中均表达,其中在胎盘中表达量最高,心脏、肾脏、大脑、肌肉、舌、脂肪、肝脏、肺脏和表1引物序列信息。

Table 1. Primer sequence information
表1. 引物序列信息

Table 2. Analysis software and its online address
表2. 分析软件及其在线网址
脾脏中均有表达(见图1)。
4.2. 山羊PHLDA2基因启动子序列获得及生物信息学分析
PCR扩增及测序结果显示获得长1842 bp片段,包括5’ UTR区域20 bp和启动子区域序列1822 bp。通过NCBI,与山羊基因组数据库比对,发现该序列存在山羊29号染色体。
同源性分析发现山羊PHLDA2基因启动子与绵羊、牛的启动子同源性较高,分别为93.2%和87.9%, 与人、猪和鼠PHLDA2启动子同源性较低。
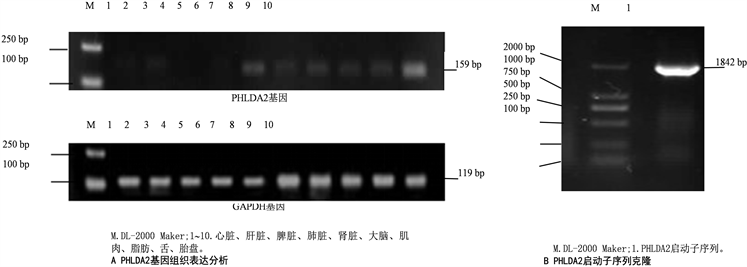
Figure 1. Expression analysis of PHLDA2 gene in goat tissues and amplification of its promoter sequence
图1. 山羊PHLDA2基因组织表达分析和其启动子序列扩增
为了充分了解不同物种PHLDA2基因启动子结构特征及物种间的同源关系,对NCBI数据库上可以搜索到其它物种PHLDA2基因启动子进行聚类分析,包括人(Homo sapiens)、牛(Bos taurus)、家犬(Canis lupus familiaris)、斑马鱼(Danio rerio)、鸡(Gallus gallus)、家鼠(Mus musculus)、绵羊(Ovis aries)、黑猩猩(Pan troglodytes)、褐家鼠(Rattus norvegicus)和猪(Sus scrofa)共搜索到10条进行分析。将筛选出来的序列用phylogeny (Bootstrap = 1000)构建PHLDA2启动子进化树。结果表明山羊与绵羊、牛PHLDA2启动子亲缘关系最近(见图2)。
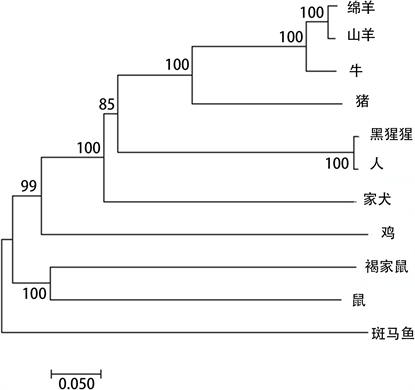
Figure 2. Phylogenetic tree construction of goat PHLDA2 promoter sequence
图2. 山羊PHLDA2启动子序列系统进化树构建
4.3. PHLDA2启动子预测和结合位点分析
对山羊PHLDA2基因起始密码子上游2000 bp的序列进行3种不同的在线软件预测,分析启动子结构。FPROM软件分析得到启动子可能位于−52 bp附近,−30 bp附近存在TATA框(TATAAA),−75 bp附近存在GC框(GGGCGGG)。Neural Net Work Promoter Prediction (NNPP)软件预测出启动子可能位于−51~−2 bp。用Promoter Scan在线软件分析,且cut-off值设置为0.8,预测PHLDA2启动子,结果显示在启动子区−51~−2 bp, Promoter Score为80.47,Promoter Cutoff为53.0,综合3个软件的预测结果,PHLDA2基因启动子可能位于−2~−51 bp区域内(见表3)。利用PROMO在线软件预测山羊PHLDA2基因启动子转录因子结合位点时,发现CpG岛内存在YY1转录因子结合位点(见图3)。

Table 3. Prediction of core activity areas of gene promoter PHLDA2 in goat
表3. 山羊PHLDA2基因启动子的核心活性区域预测
4.4. CpG岛的分析
利用CpG Island Searcher软件,以(G + C)s/Total bases > 55%、CpG observed/CpG expected > 0.65、CpG length = 200~2000 bp作为检测依据,发现山羊PHLDA2基因启动子区和第一外显子区存在一段典型的CpG岛(−631~379 bp) (见图3),分别选取启动子和外显子CpG位点密集区域−302~−88 bp和49~333 bp,依次包含23个和29个CpG位点。
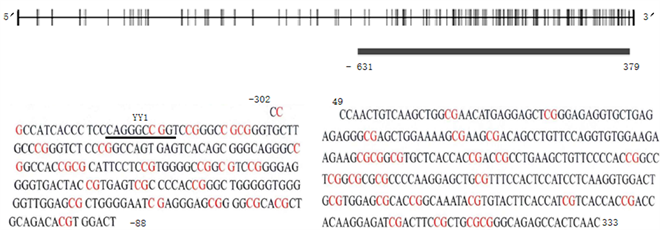
Figure 3. CpG island prediction of goat PHLDA2 gene promoter and its first exon
图3. 山羊PHLDA2基因启动子和其第一外显子CpG岛预测
4.5. 甲基化位点分析
通过亚硫酸盐处理技术检测CpG岛内CpG位点胞嘧啶的甲基化水平,包括PHLDA2启动子区(−302~−88 bp)和其第一外显子区(49~333 bp)。结果分析发现:胎盘PHLDA2第一外显子区域甲基化率1.28%,显著低于其他组织第一外显子甲基化水平(P < 0.05),其启动子区的甲基化水平为22.6%,同其他组织甲基化水平无明显差异(P > 0.05) (见图4),但其启动区CpG岛内第三位点CpG的甲基化频率为0.03%,显著低于其它组织该位点的甲基化频率(P < 0.05) (见图5)。
 注:字母完全不同表示差异显著(P < 0.05);无标注表示差异不显著(P > 0.05)。
注:字母完全不同表示差异显著(P < 0.05);无标注表示差异不显著(P > 0.05)。
Figure 4. Methylation analysis of goat PHLDA2 promoter region and its first exon region
图4. 山羊PHLDA2启动子区域和其第一外显子区域甲基化分析
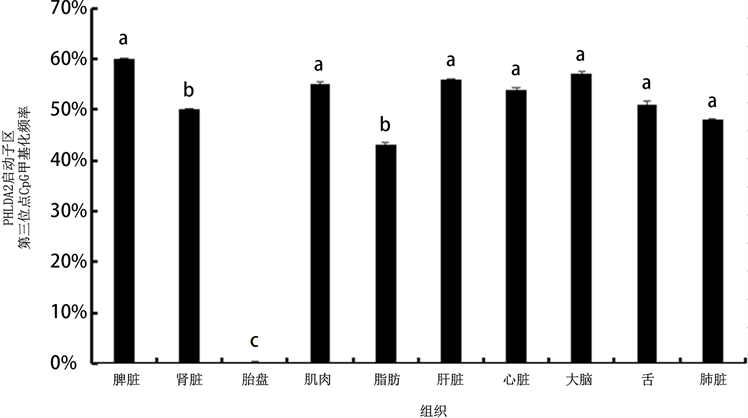 注:字母完全不同表示差异显著(P < 0.05)。
注:字母完全不同表示差异显著(P < 0.05)。
Figure 5. Methylation analysis of PHLDA2 promoter region in the third CpG site
图5. 山羊PHLDA2启动子区第三位点CpG甲基化分析
5. 讨论
本研究根据PHLDA2启动子在人、鼠、牛和猪等物种间的保守性,利用比较基因组的方法,首次克隆了山羊PHLDA2启动子。核苷酸序列分析表明山羊PHLDA2启动子与绵羊、牛的序列相似性较高,同源性分别为93.2和87.9%,进一步证明山羊PHLDA2启动子在物种间具有一定的保守性。
PHLDA2基因在山羊的心脏、肝脏、肺脏、脾脏中表达量较少,大脑、肌肉、脂肪、肾脏、舌和胎盘组织中均表达,其中胎盘中表达量最高。
基因的启动子序列和启动子转录因子的预测,为了解基因功能和调节机制提供强有力的重要基础,预测软件发现启动子TATA框位于−30 bp区域,基因启动子位置可能位于起始密码子上游48~97 bp区域内,预测启动子CpG岛存在YY1转录因子结合位点(见图3)。YY1是一种锌指蛋白,泛表达在哺乳动物细胞,而且进化相当保守,调控一系列基因的表达,包括印记基因 [23] [24] [25]。因此,组织间转录因子表达量的差异变化可能影响山羊PHLDA2基因的表达,影响山羊胚胎的发育。
同时发现PHLDA2基因的启动子附近存在CpG岛,DNA甲基化常常发生在哺乳动物CpG岛胞嘧啶的第5个碳原子上,研究证明这种修饰能阻碍转录复合体与DNA相互结合和改变染色体的构型,从而基因的转录受到抑制 [26]。YY1转录因子结合位点刚好位于CpG岛内第三个CpG位置,所以认为此CpG位点胞嘧啶的甲基化抑制了YY1转录因子的结合,导致山羊PHLDA2基因的表达减少,同时,间接证明YY1转录因子对PHLDA2基因的表达可能起促进作用。另外,也有报道发现基因内部的CpG胞嘧啶甲基化与基因表达存在着负相关性 [27]。本研究脾脏等其它组织启动子和第一外显子区域CpG甲基化程度明显高于胎盘组织,RT-PCR结果显示PHLDA2在胎盘中大量表达,但在脾中几乎不表达,推测山羊PHLDA2基因启动子和第一外显子的高甲基化抑制其转录。此外,CpG岛胞嘧啶的甲基化与基因组印记密不可分,印记基因是指亲本等位基因在下一代细胞中差异表达或不表达,这种差异表达的机制之一是基因启动子区CpG岛内存在差异甲基化区域(Different methylation regions, DMRs)。前期研究推断山羊PHLDA2基因在胎盘中表达母本等位基因,在其它组织双等位表达,但是这种表达的机制仍不清楚。本研究发现PHLDA2基因在山羊胎盘显著高于其它组织,在脾脏组织中甚至不表达,可能是由于PHLDA2基因启动子或第一外显子CpG岛甲基化的影响,结果表明胎盘PHLDA2基因启动子区第三CpG位点和第一外显子区的甲基化率显著低于脾脏等其它组织(P < 0.05)。
本文成功的克隆了山羊PHLDA2基因启动子序列,并预测了潜在的转录因子结合位点,进一步分析其启动子和第一外显子甲基化状态,可为了解山羊PHLDA2基因的功能和调节机制提供了重要依据。
6. 结论
PHLDA2的mRNA相对表达量水平与启动子区CpG岛(−302~−88 bp)内第三个CpG位点甲基化频率和第一外显子CpG岛(49~333 bp)甲基化水平呈负相关。
基金项目
江汉大学科研机构培育基金;江汉大学学科特色方向协同创新团队项目(03100074);重庆市科委自然科学基金项目(2009BA1052);中央高校基本科研业务费专项(XDJK2011C022)。