1. 引言
2021年11月9日,南非豪登省在采集的标本中检测出第一个奥密克戎变异毒株(Omicron, B.1.1.529) [1],奥密克戎变异株的传染率是Delta变异株的3至6倍。奥密克戎变异株中的基因序列积累了大量的突变 [2],使得奥密克戎对宿主ACE受体有很高的亲和性,能够更紧密的黏附在人体细胞上,并对人体内的抗体有更高的抵抗力,与疫苗接种者或感染者产生的大多数中和抗体不匹配 [3],变异株有更强的感染性,感染的症状也出现改变,在治疗过程中使用的药物以及接种的疫苗都受到很大的影响。
通过对变异株的基因序列进行监测分析和追踪,将基因序列分段处理和对比分析,构建图形来解析复杂的基因序列,提取更多有用的基因信息,检测出各个基因段的特征,以及确定基因段的排序和位置,有助于更全面的了解基因的特性和功能,利于治疗药物的研制和疫苗的研发。
基因序列分段测量可视化是将基因序列转化为字符来进行处理,利用测量模型,将处理的样本转化为可以进行观察的图示 [4],形成二维、三维或者多维基因序列可视化图,得到的可视化结果可以用于映射样本基因组的非线性逻辑关系,显示研究样本的特征分布。
本文使用新型冠状病毒变异株奥密克戎全基因序列作为样本,将样本进行处理,建立一套展示奥密克戎基因序列分段测量特征分布的可视化模型 [5] [6] [7],并对可视化图示进行分析。
2. 研究设计
2.1. 数据概述
本次研究中使用的序列为新型冠状病毒变异株奥密克戎(omicron, B.1.1.529)全序列,序列长度为29,740,数据来源为NCBI (美国国家生物技术信息中心)官网(https://www.ncbi.nlm.nih.gov/) [8],数据可靠度较高。
2.2. 架构
基因序列分段测量可视化系统整体架构如图1所示,包括输入、计数、三维直方图、投影、输出五个部分。
2.3. 计数模块
计数模块(如图2所示)是针对输入的奥密克戎全基因序列进行处理,将导入的基因序列以我们设定的某个碱基数量自动分段,将序列N分割成几个长度为m的子序列,这样就有M = N/m个子序列,本次研究m值为30,共992个子序列。然后计算出每段中相应的四种碱基(A, C, G, T)数量并进行输出。
2.4. 三维直方图模块
三维直方图模块(如图3所示)是将奥密克戎全基因序列分组后,基因序列中A,C,G,T碱基及空
集形成16种不同组合并转置为矩阵,将其中任意两个作为X、Y轴,提取各频数生成256幅三维直方图,利用三维频数直方图,通过不同颜色的变化来直观呈现不同组合碱基频数分布。
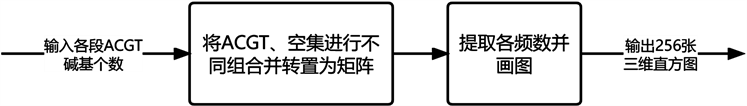
Figure 3. Three-dimensional histogram module
图3. 三维直方图模块
2.5. 投影模块
投影模块(如图4所示)是将奥密克戎全基因序列分段后,基因序列中A,C,G,T碱基及空集形成16种不同组合并转置为矩阵,将其中任意两个作为X、Y轴,提取各频数生成256幅投影图像,并使用不同的颜色进行区分,通过不同颜色的变化在投影图上来直观呈现不同组合碱基频数分布。
3. 结果分析
3.1. 分段测量图
本文主要对奥密克戎全基因序列进行分段测量可视化及分析,从而对有关奥密克戎变异株的生物科学研究提供一定的基础。本次我们选择了一组奥密克戎(B.1.1.529)全基因序列,此组序列共29740个碱基,我们将其分为30个一组,共992组。分别计算每一组四种碱基(A, C, G, T)不同组合(Φ, A, C, G, T, AC, AG, AT, CG, CT, GT, ACG, ACT, AGT, CGT, ACGT)共16种组合的碱基总和数,并且又将16种组合两两组合统计碱基数量的频数(即组数)。图5为奥密克戎基因序列256种组合投影的分段测量图,可以看到投影图趋近对称图形,具有规律性。
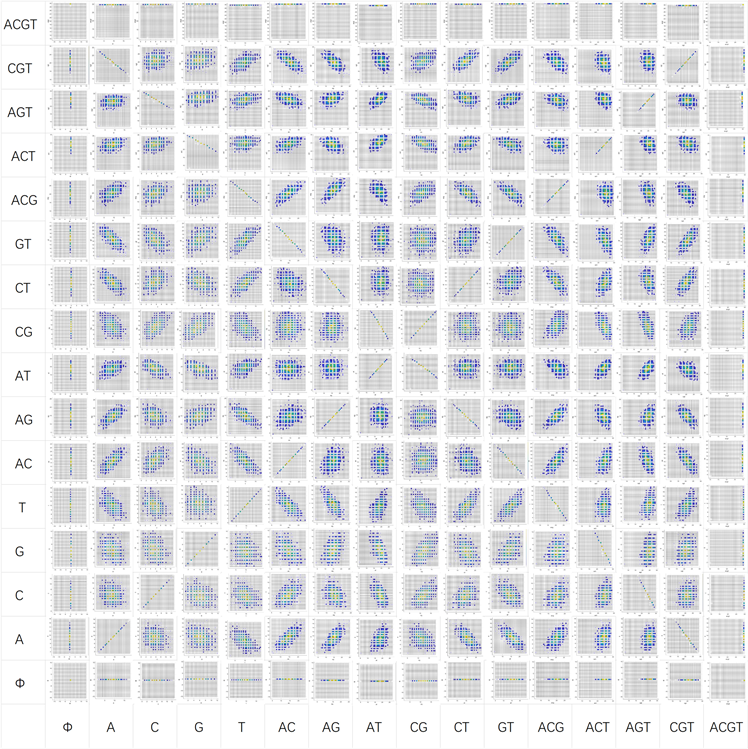
Figure 5. Variable value measurement map of 256 combinations projection of Omicron
图5. 奥密克戎基因序列256种组合投影的变值测量图
3.2. 可视化分析
如图5所示,整个图主要被分为四个部分,这两条“分界线”即是碱基中互补组合和相等组合的图像,互补组合包括A-CGT、C-AGT、G-ACT、T-ACG、AC-GT、AG-CT、AT-CG、CG-AT、CT-AG、GT-AC、ACG-T、ACT-G、AGT-C、CGT-A共14种组合,相等组合即A-A、C-C、G-G、T-T、AC-AC、AG-AG、AT-AT、CG-CG、CT-CT、GT-GT、ACG-ACG、ACT-ACT、AGT-AGT、CGT-CGT共14种组合。
我们将各自从这两种组合中选取一张图像进行分析,如图6所示为A-A三维分布及其投影图,如图7所示为CGT-A三维分布及其投影图,图像左图为组合的三维分布图,右图为左图的投影图像。
如图6所示,由于是相同碱基的两两组合,所以组数主要分布在投影图的对角线,即x = y这条线上。如图7所示,由于每段分组共30个,故组数主要分布在投影图的对角线,即x + y = 30这条线上。此时我们观察各投影点颜色情况,根据色卡可知颜色越接近黄色频数越大,两张图像颜色都由两端向中心逐渐变黄(其中白色部分为频数为0),最大值(z轴)大概在160左右。

Figure 6. A-A three-dimensional distribution and its projection diagram
图6. A-A三维分布及其投影图

Figure7. CGT-A three-dimensional distribution and its projection diagram
图7. CGT-A三维分布及其投影图
投影图的四个定点分别为Φ-Φ、Φ-ACGT、ACGT-Φ、ACGT-ACGT四种组合的图像。Φ-Φ数据集中在点(0,0),Φ-ACGT数据集中在点(0,30),ACGT-Φ数据集中在点(30,0),ACGT-ACGT数据集中在点(30,30)。四个点色块颜色都为黄色,为全部组数的数量992组。如图8所示为Φ-Φ三维分布及其投影图。
投影图的四周由“横线”和“竖线”的图像组合而成。这些图共有四类组合,第一类为y = 0的组合,共有14种组合,分别为A-Φ、C-Φ、G-Φ、T-Φ、AC-Φ、AG-Φ、AT-Φ、CG-Φ、CT-Φ、GT-Φ、ACG-Φ、ACT-Φ、AGT-Φ、CGT-Φ,如图9左上为A-Φ三维分布及其投影图。第二类为y = 30的组合,共14种组合,分别为A-ACGT、C-ACGT、G-ACGT、T-ACGT、AC-ACGT、AG-ACGT、AT-ACGT、CG-ACGT、CT-ACGT、

Figure 8. Φ-Φ three-dimensional distribution and its projection diagram
图8. Φ-Φ三维分布及其投影图
GT-ACGT、ACG-ACGT、AGT-ACGT、ACT-ACGT、CGT-ACGT,如图9右上为A-ACGT三维分布及其投影图。第三类为x = 0的图像,共14种组合,分别为Φ-A、Φ-C、Φ-G、Φ-T、Φ-AC、Φ-AG、Φ-AT、Φ-CG、Φ-CT、Φ-GT、Φ-ACG、Φ-ACT、Φ-ACT、Φ-CGT,如图9左下为Φ-A三维分布及其投影图。第四类为x = 30的图像,分别为ACGT-A、ACGT-C、ACGT-G、ACGT-T、ACGT-AC、ACGT-AG、ACGT-AT、ACGT-CG、ACGT-CT、ACGT-GT、ACGT-ACG、ACGT-ACT、ACGT-AGT、ACGT-CGT,如图9右下为ACGT-A三维分布及其投影图。图9的四个图色块都从两端由深蓝逐渐变黄,最大值在160左右。

Figure 9. Four-dimensional distribution and its projection diagram
图9. 四类三维分布及其投影图
在投影图的主要图像部分,主要有两种形态的图像。第一种是偏圆形聚集的三维图像,在投影图的内两圈部分,图像都趋近此形态分布,分别有AT-CT、CG-CT、AG-CG、AG-AT、AT-AG、CG-AG、CT-CG、CT-AT、AC-CT、AC-CG、AC-AT、AC-AG、AG-AC、AT-AC、CG-AC、CT-AC、GT-CT、GT-CG、GT-AT、GT-AG。可以发现这24中组合都是A、C、G、T两个碱基组合分别作x轴和y轴的三维图像,其中每组的x轴和y轴都有重复的一个碱基,比如AT-CT便出现了重复的碱基T。如图10所示为AT-CT三维分布及其投影图,可看到图像最大值即浅黄色部分主要集中在整个图像相对中心的位置,并且颜色从四周逐渐向中心变浅,最大值在30左右。
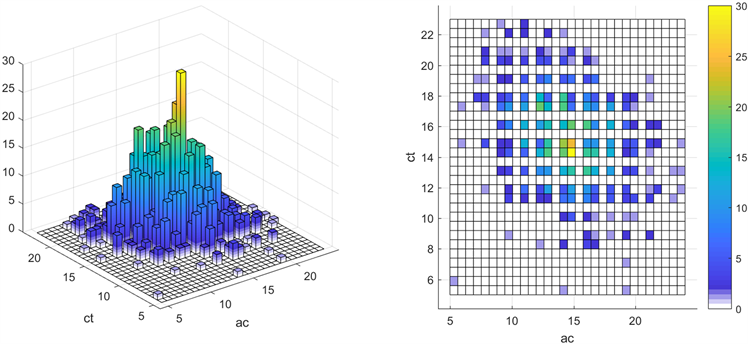
Figure 10. AT-CT three-dimensional distribution and its projection diagram
图10. AT-CT三维分布及其投影图
第二种是相对于第一种偏长条形的分布形态的三维图像。比较典型的分布是在投影图的从内向外的第三圈部分。即AC-ACG、AG-ACG、AT-ACG、CG-ACG、CT-ACG、GT-ACG、ACG-GT、ACG-CT、ACG-CG、ACT-AT、ACG-AG、ACG-AC、GT-T、CT-T、CG-T、AT-T、AG-T、AC-T、T-AC、T-AG、T-AT、T-CG、T-CT、T-GT共24种组合。在所有长条形分布的图像里,比较典型的是以下四种,分别
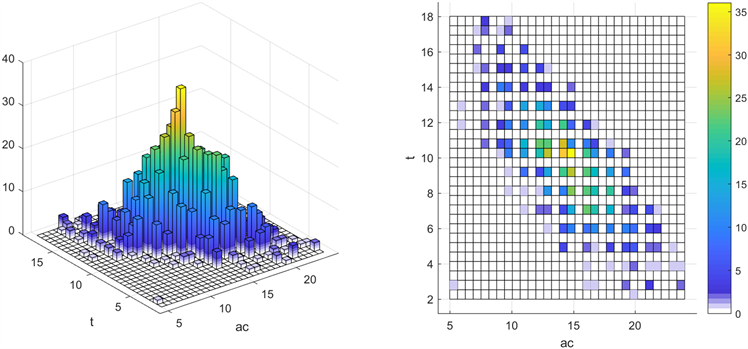
Figure 11. AT-T three-dimensional distribution and its projection diagram
图11. AC-T三维分布及其投影图像
是西南-东北走向的细条形,西北-东南走向的细条形,西南-东北走向的宽条形,西北-东南走向的宽条形。如图11所示为AC-T三维分布及其投影图像,其呈典型的西北东南走向的细条形分布,最大值点位于图像中心,最大值为35以上。
除上面所说的从内第三圈之外,呈条形状分布的图像还分布在图5四个红框区域(区域包括上述提到的从内第三圈部分),图12中以下区域图像都为上述四种类型中的一种,在此不再列举。
4. 总结
本文实现了对奥密克戎变异株的基因序列进行分段测量的可视化展示,可对其进行放大分析。在实现过程中可以通过调整颜色卡使其可视化更加具有针对性以及更直观,也可以通过调整分段数量获取不一样的可视化结果。通过对奥密克戎基因序列分布进行可视化展示,相对于传统生物研究,更具直观性并且计算方便,能够为生物科学研究提供一定的研究基础,加快研究进程。
致谢
感谢郑智捷教授的悉心指导,感谢云南大学软件学院对本项目的支持。