1. 引言
长链非编码RNA (Long non-coding RNA, lncRNA)是一类不编码蛋白质且长度不小于200 nt的转录物序列 [1] [2] [3]。长期以来,lncRNA被视作没有生物学功能的“噪声”,但是最新的研究表明,lncRNA与基因表达,蛋白质翻译和稳定性等细胞机制有关 [4]。除此之外,lncRNA还和许多疾病密切相关,例如癌症、糖尿病等 [4] [5] [6]。因此,研究lncRNA具有十分重大的意义。近些年来,随着高通量测序技术的发展,产生了大量的新的序列数据,这其中就有许多长链非编码RNA,而研究其功能的第一步就是准确识别出长链非编码RNA,所以研究出能够准确快速识别出lncRNA的工具是迫切的需求。
目前已经发展出了一些识别lncRNA的生物信息学方法。第一大类是基于比对的方法。Kong等在2007年提出了CPC,其使用六个具有生物学意义的特征,并基于支持向量机模型预测转录本序列的编码能力 [7]。这六个特征包括三个开放阅读框(open reading frame, ORF)特征以及和已知蛋白质的序列相似性特征。CPC具有较高的蛋白质编码转录本识别能力,但也存在速度缓慢等不足之处。Lin等在2011年提出了PhyloCSF [8]。PhyloCSF充分利用多重比对并产生非编码RNA和编码RNA的似然比。上述基于比对的方法都依赖于数据库,这限制了它们的预测能力。第二大类是不基于比对的方法,这类方法依赖于序列本身固有的信息,这提高了其预测转录本序列编码能力的效率。Wang等在2013年提出了CPAT,其使用开放阅读框的最大长度、开放阅读框覆盖率、Fickett分数以及hexamer分数作为特征构建了逻辑回归模型去识别lncRNA [9]。Hexamer得分是CPAT所使用的特征中最有识别力的特征 [10]。Li等提出了PLEK去识别lncRNA [11]。PLEK使用校准的k-mer作为特征并使用支持向量机算法来区分长链非编码RNA和信使RNA。Tong等在2019年提出了CPPred [12]。CPPred基于支持向量分类器预测转录本序列的编码能力。近些年来,深度学习在诸多领域得到了广泛应用,随之也出现了一些基于深度学习技术去识别lncRNA的工具,例如DeepCPP [13]、LncRNAnet [14]、LncADeep [15] 等。
在本文中,我们提出了一种新方法去识别lncRNA。我们使用k-mer作为原始特征,利用主成分分析法对k-mer特征进行降维。我们选择累积方差贡献率比较大的一些主成分作为新的特征向量,并将其作为我们构建的多层感知机神经网络的输入来预测转录本序列的编码能力。最后,我们使用灵敏度、特异度和准确率来评估我们提出方法的性能。
2. 材料和方法
2.1. 数据集
我们使用人类的编码RNA序列和长链非编码RNA序列去训练模型。得到模型后,使用人类、小鼠、斑马鱼物种的转录物序列来评估我们所提出方法的性能。我们从Ensembl数据库中收集上述物种的转录物序列来建立各个物种的数据集。
我们从人类数据集中的长链非编码RNA序列和编码转录物序列中分别随机抽取三分之二的序列作为训练集,剩余的序列作为人类物种测试集,共得到了16068条编码转录物序列以及19606条长链非编码RNA序列作为训练集,8034条编码RNA和9804条长链非编码RNA序列作为测试集。
当在其他物种数据集上测试模型性能时,将上述所有人类物种转录物序列作为测试集来训练模型。具有小开放阅读框的长链非编码RNA (sORF)是长度不超过300 nt的长链非编码RNA,对其进行正确预测比较困难,还有提升的空间。我们仍然构建了各个物种的sORF类型的测试集来评估我们所提出的方法,表1展示了各个数据集的详细信息。

Table 1. The data amount used for evaluating the performance of the method proposed in this work
表1. 用于评估本文提出方法性能的数据集所含样本数目
2.2. 特征提取
K-mer是一种从DNA序列和RNA序列中提取信息的常见且有效的方法,在生物信息学的许多领域中有着广泛的应用。对于一条给定的转录物序列,我们以k为滑动窗口的大小,每次滑动1个步长去分割该转录物序列,并计算4k种子序列出现的频率,滑动窗口的长度为k,且每个位置的碱基有四种(A:腺嘌呤,T:胸腺嘧啶,C:胞嘧啶,G:鸟嘌呤),因此共有4k种子序列。因此,我们得到了一个表示转录物序列的维度为4k的向量。可以注意到,随着k的不断增大,k-mer向量的维度呈指数级增长,这可能会引起“维度灾难”。在本研究中,我们将k设置为3、4、5,因此我们可以将一条转录物序列表示为一个1344维的向量。
2.3. 主成分分析
特征的维度并不是越高越好,高维特征向量中可能会存在信息冗余及噪声,进而影响模型的精度和运算速度,导致过拟合问题,因此在将其输入到模型之前进行降维是非常有必要的。常见的降维方法有主成分分析,方差分析,因子分析等。本文选择主成分分析来进行特征降维。主成分分析使用方差来作为衡量信息量大小的标准,在信息损失尽可能小的情况下,将高维空间上的数据映射到低维空间,将含有冗余信息的多个变量映射成数量较少的不相关的新变量,也就是主成分。使用主成分分析法进行降维的步骤如下 [16] [17]:
1) 对数据集进行中心化处理
2) 计算样本的协方差矩阵
3) 计算协方差矩阵的特征值与特征向量
4) 取最大的i个特征值所对应的特征向量,记作
,,因此得到投影矩阵:
其中i为选取的主成分的个数。
2.4. 多层感知机模型
近些年来,深度学习技术迅速发展,在许多领域有着广泛的应用,在生物信息学中也得到了越来越多的应用。多层感知机是一种经典的深度学习模型,它包括输入层、输出层和若干隐藏层,这显著提高了神经网络拟合非线性数据的能力。在本文中,我们构建了多层感知机模型去从转录本序列中识别lncRNA。我们构建的多层感知机模型包括输入层,输出层以及若干个隐藏层。本文构建的多层感知机模型将累积方差贡献率比较高的主成分作为输入,输出该转录本序列是lncRNA和编码转录本的概率。激活函数有着非常重要的作用,它是非线性变换,若没有激活函数,多层神经网络实际上和单层神经网络没有区别。使用激活函数为神经网络加入了非线性变换,使得其可以拟合更加复杂的数据。常见的激活函数有sigmoid、tanh和relu函数。我们在本文中构建的多层感知机模型的每个隐藏层的神经元节点数都被设置为相同的,并使用relu函数作为激活函数,输出层使用softmax函数作为激活函数来输出转录本是lncRNA和编码转录本的概率。随机丢弃在深度学习中是一种常见且有效的技术。随机丢弃是指在神经网络训练的过程中,随机忽略一部分神经元,这有利于简化神经网络、缓解过拟合问题。本文在构建多层感知机时,在每一个隐藏层都使用随机丢弃方法随机忽略一半的神经元来减轻神经网络过拟合问题。本文提出的方法流程如图1所示:

Figure 1. The flow chart of methods proposed in the paper
图1. 本文方法流程图
2.5. 评估准则
我们使用准确率(ACC),灵敏度(SN)和特异度(SP)等常见的机器学习评估指标来评估我们所提出方法的性能:
(1)
(2)
(3)
其中TP代表被正确识别的阳性样本的数量,TN,FP,FN分别代表真阴性,假阳性,假阴性样本的数量,在本文中,lncRNA为阳性样本。
3. 结果
3.1. 参数对模型性能的影响
特征数量过多时会造成计算效率低下,过拟合等问题,因此我们利用主成分分析来降低特征维度。我们选择前333个主成分,其累积方差贡献率在95%左右。
在本文中我们使用k-mer作为原始特征向量,利用主成分分析进行降维,得到维度为333的特征向量,将其作为多层感知机的输入,进而得到转录本为lncRNA和编码转录本的概率,选取概率大的类别作为最后的预测结果。
K-mer是生物信息学中常见且有效的特征提取方式,我们在测试集上验证了使用不同k-mer作为特征搭建的模型的性能。图2展示了不同k-mer作为特征时,不同测试集上模型的性能。我们可以发现在选择3-mer,4-mer和5-mer组合特征时,在相应的测试集上ACC达到最大,尤其是在HumansORF testdataset上的ACC的提高非常明显,因此我们选择3-mer,4-mer和5mer为原始特征。

Figure 2. The effect of different values of k on the performance of the model
图2. k不同的取值对模型性能的影响
多层感知机中隐藏层的层数和每一层的节点数是神经网络中非常重要的参数,我们在本文中所搭建的模型每一个隐藏层中含有相同的节点个数。为了选取最好的参数以及展示不同参数对性能的影响,我们分别选择不同的隐藏层的层数和不同的节点个数来构建模型并使用人类物种数据来测试其性能。
当固定节点个数时,我们将隐藏层的个数分别设置为1,2,3,4,5搭建模型来测试其性能。可以发现在Human normal test dataset上时,不同的隐藏层的个数对应的ACC大体相同,但是在Humans ORF test dataset上不同的隐藏层层数对应的ACC变化较大,当隐藏层的层数为5时对应的ACC明显高于其他隐藏层层数对应的ACC,因此我们将隐藏层的个数设置为5。图3展示了隐藏层的层数对模型性能的影响。
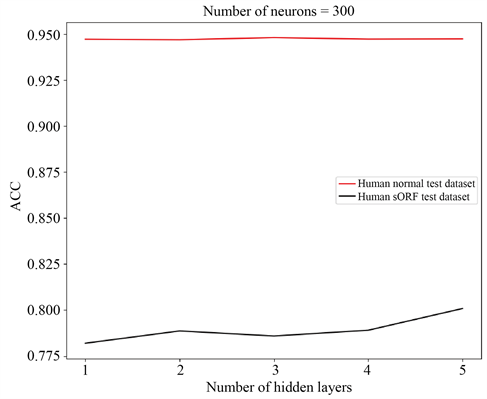
Figure 3. The effect of the number of hidden layers on model performance
图3. 隐藏层的层数对模型性能的影响
当固定隐藏层的层数时,我们将节点数设置为50、100、150、200、250和300来构建不同的模型。在Human normal test dataset上,不同的节点数对应的ACC差别不大。在Humans ORF test dataset上不同的节点数对应的ACC波动较为明显,当节点数设置为300时,所搭建的模型在Humans ORF test dataset对应的ACC达到最大,因此我们将每一个隐藏层所含有的神经元个数设置为300。图4展示了不同的节点数对模型性能的影响。

Figure 4. The effect of the number of neurons on model performance
图4. 节点数对模型性能的影响
3.2. 模型性能
我们在人类、小鼠和斑马鱼的不同类型测试集上测试我们提出的方法的性能,就准确率而言,我们提出的方法在Human normal test dataset上最高,为94.74%,另外两个物种的normal类型数据集即Mouse normal test dataset,Zebrafish normal test dataset次之,分别为93.25%和93.04%,并且normal类型数据集上的准确率高于sORF类型的数据集上的准确率。
对于灵敏度指标,Human normal test dataset最高,为95.24%,Mouse normal test dataset次之,为91.12%,其余数据集的灵敏度均在90%之下,Human sORF test dataset,Mouse sORF test dataset上的灵敏度均在80%之上,分别为82.30%,84.97%,而斑马鱼物种的测试集上的灵敏度均较低,Zebrafish normal test dataset上的灵敏度75.64%,Zebrafish sORF test dataset上的灵敏度为64.36%。
就特异度而言,Human normal test dataset、Mouse normal test dataset和Zebrafish normal test dataset的特异度均超过了90%,其中最高的为Zebrafish normal test dataset的95.28%,Zebrafish sORF test dataset的特异度最低,为74.04%。表2展示了我们所搭建模型在各个测试集上的性能。

Table 2. The performance of the proposed method on each test set
表2. 本文提出方法在各个测试集上的性能
4. 讨论
我们在本文中提出了一个从转录本序列中识别lncRNA的新方法,我们使用3-mer,4-mer,5-mer作为原始特征,并使用主成分分析对特征进行降维得到了333维的新特征向量,然后基于多层感知机模型来预测其是否为lncRNA。我们提出的方法是不基于比对的方法,这利用了序列本身的信息。我们使用主成分分析对原始特征向量进行降维,这大大缓解了维度过高可能会导致的过拟合以及计算效率低下等问题。我们使用训练集训练模型后,使用不同物种的不同类型测试集评估了本文所提出的方法。
我们在本文中提出的方法不同于以往直接将特征送入神经网络的方法。我们首先使用主成分分析对原始特征进行了一次降维,这相当于对原始特征进行初步的提取有效信息,除此之外,这还有利于提高后续训练效率以及缓解过拟合问题。
5. 总结
本文提出了一个用于区分lncRNA和编码转录本的新方法。我们将3-mer,4-mer和5-mer作为原始特征,使用主成分分析法来降低原始特征的维度,进而得到最终的333维特征向量。我们搭建了包含5个隐藏层的多层感知机,并将降维后的333维特征向量作为其输入来识别lncRNA。我们构建的模型在人类、小鼠和斑马鱼的normal类型测试集上的准确率较高,分别达到了94.74%,93.25%和93.04%。