1. 介绍
胃癌是最常见的消化道恶性肿瘤之一,在全球癌症发病率中排名第五,STAD占所有胃恶性肿瘤的95%以上 [1]。尽管目前已经对胃癌患者采取了手术联合化疗、免疫治疗等多学科治疗,但中位生存期不到12个月,治疗效果因人而异 [2]。因此,建立一个准确的风险模型对STAD患者进行分层,对设计个性化的治疗方案至关重要。
铜是生物体内的重要辅助因子,参与广泛的代谢过程,包括有氧呼吸、色素沉着、铁转运、超氧化物歧化和细胞外基质的生物合成等 [3]。但当细胞内铜积累过量后,可以通过与三羧酸(Tricarboxylic acid, TCA)循环的酯酰化成分直接结合导致细胞死亡,即铜死亡 [4]。据报道,已在多种癌症中观察到了铜的积累。铜积累与肿瘤细胞生长、转移以及肿瘤血管生成密切相关 [5] [6]。因此,寻找与铜死亡相关的治疗靶点可能为癌症的治疗提供新的方向。
LncRNA是一种长度超过200个核苷酸且不具备蛋白质编码功能的RNA。其通过调节癌症相关基因的转录调控、转录后调控、翻译后修饰等在癌症的发生、进展和代谢重编程的过程中发挥重要作用 [7] [8]。并且最近有研究开发了lncRNA作为癌症患者的新的预后指标 [9]。一项研究建立了细胞焦亡相关lncRNA风险模型来预测STAD患者的预后 [10]。然而,在STAD中,铜死亡相关的lncRNA的预测作用仍然未知。因此,该研究旨在利用生物信息学构建并验证铜死亡相关的lncRNA风险模型,用于预测STAD患者的预后。
2. 资料和方法
2.1. 数据采集
STAD患者的转录组表达数据和临床数据来自于TCGA (https://portal.gdc.cancer.gov/)数据库。其中包括375个肿瘤样品和32个正常组织样品。使用Perl编程语言(Strawberry-Perl-5.30.0版;https://www.perl.org),以已标准化的每千碱基百万片段(FPKM)格式提取转录组表达数据。同时用Pearl对临床数据进行预处理,获得临床数据完整的信息。
2.2. 筛选铜死亡相关lncRNA
根据之前的研究 [4] [11] [12],确定了19个铜死亡相关基因(NFE2L2, NLRP3, ATP7B, ATP7A, SLC31A1, FDX1, LIAS, LIPT1, LIPT2, DLD, DLAT, PDHA1, PDHB, MTF1, GLS, CDKN2A, DBT, GCSH, DLST)。采用Pearson相关分析研究铜死亡相关基因与lncRNA的相关性,并使用Pearson相关系数 > 0.4和p < 0.001作为标准进行筛选铜死亡相关lncRNA。使用“dplyr”、“ggalluvial”和“ggplot2”包,绘制了铜死亡基因和铜死亡相关lncRNA之间的桑基图。
2.3. 构建和验证预后风险模型
使用R“caret”包将来自TCGA数据库的STAD患者按1:1的比例随机分为训练集和测试集。训练集用于构建铜死亡相关的lncRNA风险模型,测试集和整个集用于验证风险模型。首先使用单因素Cox回归分析生成森林图以揭示STAD中与铜死亡相关的预后lncRNA基因。然后使用最小绝对收缩和选择算子(LASSO)回归来选择预测变量并避免过度拟合。然后进行多因素Cox回归分析以确定参与风险模型构建的lncRNA。基于铜死亡相关的lncRNA构建了一个风险模型,以预测STAD患者的预后。风险评分计算如下:
公式中的Expr(i)代表每个lncRNA的表达值,Coef(i)表示每个lncRNA的多重Cox回归分析的回归系数。根据中位风险评分,将患者分为高风险组和低风险组。Kaplan-Meier生存曲线分析用来探索高低风险组之间的总生存率和无进展生存率是否存在差异。对患者年龄、性别、组织学分级、临床分期、风险评分分别进行单因素和多因素Cox独立预后分析,从而观察风险评分是否可以独立于其他临床特征作为新的预后因子。通过受试者工作特征(ROC)曲线评价预后模型的预测性能。
2.4. 构建列线图
使用rms包,建立了列线图用来预测STAD患者1、3和5年生存率。采用校准曲线检验预测的存活率是否与实际存活率一致。
2.5. PCA、GO富集分析、KEGG富集分析
为了获得不同风险组中铜死亡相关lncRNA的表达分布模式,使用散点图进行了PCA来显示高风险和低风险样本的空间分布。此外,对于低风险和高风险人群的差异基因,使用了GO分析,它由三个组成部分组成:生物过程(BP)、细胞成分(CC)和分子功能(MF)。此外,分析了两组中差异表达基因的KEGG通路。
2.6. 免疫相关功能分析和肿瘤突变负荷
通过“limma”、“GSVA”和“GSEABase”软件包对STAD中的浸润免疫细胞和免疫相关功能进行单样本GSEA (ssGSEA)评分,并以热图的形式呈现。从TCGA网站下载体细胞突变数据后,应用Perl提取突变数据。然后,使用“maftools”包检查和整合TCGA数据,并分析了高风险组和低风险组之间TMB的差异。
2.7. 统计学分析
在本研究中使用R4.2.0进行了统计分析。Pearson相关分析用于分析铜死亡相关基因与铜死亡相关lncRNA的相关性。Kaplan-Meier分析用于比较亚组之间的总生存期。进行单因素和多因素Cox回归分析以确定总生存期的独立预测因子。P < 0.05被认为具有统计学意义。
3. 结果
3.1. 鉴定对STAD患者具有预后价值的铜死亡相关lncRNA
根据之前的研究,确定了19个铜死亡相关基因。通过共表达分析,筛选出430个与铜死亡相关基因有共表达关系的lncRNA (图1(A))。然后,将371名STAD患者被随机分配到训练集(N = 186)或测试集(N = 185)。然后,通过单因素Cox分析(P < 0.05)筛选出18个差异表达的预后相关lncRNA:AC011632.1,AC016737.1,LINC00494,AC008915.2,AC234775.2,DIRC1,BX890604.1,PINK1-AS,STX18-AS1,HAGLR,OGFRP1,DPP4-DT,AC016394.2,AC009336.1,AP005057.1,VCAN-AS1,AL513165.1,AC022113.1 (图1(B))。
3.2. 铜死亡相关lncRNA预后预测模型的构建
通过LASSO回归分析进一步筛选预后相关的lncRNA,获得了15个lncRNA (图1(C),图1(D))。根据多因素Cox分析结果,其中8个lncRNA被引入到预后模型当中(图1(E))。风险得分 = AC016737.1 × (0.4278) + AC008915.2 × (0.5498) + AC234775.2 × (0.3267) + DIRC1 × (0.6624) + STX18-AS1 × (−1.4439) + HAGLR × (0.2656) + AL513165.1 × (−0.4177) + AC022113.1 × (0.8877)。在训练集和测试集以及所有患者中,高风险组的生存期显著缩短(图2(A)~(L))。同样,与低风险组相比,高风险组的无进展生存期显著降低(图2(M))。
3.3. 风险评分可作为独立的预后因素并指导STAD患者的临床结果预测
使用单因素和多因素Cox回归分析探究在STAD患者中铜死亡相关lncRNA对预后的预测价值。结果表明,铜死亡相关lncRNA构建的风险评分与患者生存期显著相关,是STAD患者的独立预后因素(图3(A),图3(B))。并且,1年,3年和5年的ROC曲线下面积(AUC)分别为0.681,0.684和0.732 (图3(C))。该模型的1年ROC风险评分的AUC为0.681,与其他临床特征相比具有更强的预测能力(图3(D))。同样,风险模型的C指数高于任何其他临床特征的C指数(图3(E))。
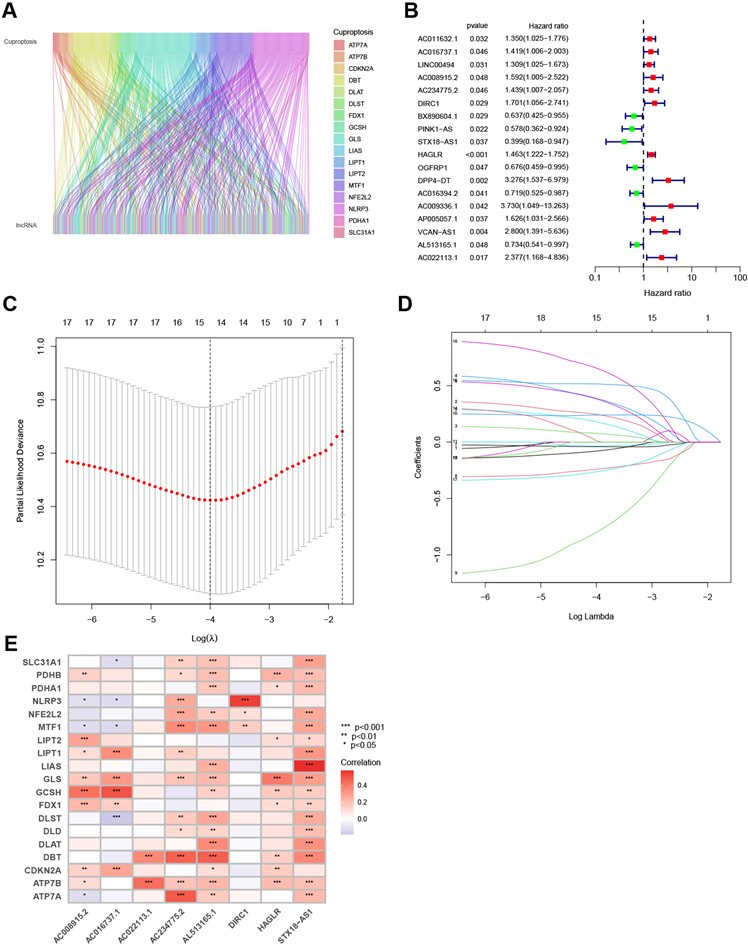 (A) 铜死亡基因与铜死亡相关lncRNA的桑基图。(B) 森林图显示了铜死亡相关的预后lncRNA。(C) 铜死亡相关lncRNA的LASSO系数分布。(D) 最小绝对收缩和选择算子(LASSO)算法中变量选择的10倍交叉验证。(E) 风险模型中铜死亡相关基因与lncRNA的相关性。
(A) 铜死亡基因与铜死亡相关lncRNA的桑基图。(B) 森林图显示了铜死亡相关的预后lncRNA。(C) 铜死亡相关lncRNA的LASSO系数分布。(D) 最小绝对收缩和选择算子(LASSO)算法中变量选择的10倍交叉验证。(E) 风险模型中铜死亡相关基因与lncRNA的相关性。
Figure 1. Identification of cuproptosis-related lncRNA with prognostic value in patients with STAD
图1. 鉴定对STAD患者具有预后价值的铜死亡相关lncRNA
 训练集、测试集及整个集的风险评分分布(A)~(C)、生存时间和生存状态(D)~(F)、8个lncRNA表达热图(G)~(I)及Kaplan-Meier生存曲线(J)~(L)。(M) STAD患者整个集的无进展生存期的Kaplan-Meier生存曲线。
训练集、测试集及整个集的风险评分分布(A)~(C)、生存时间和生存状态(D)~(F)、8个lncRNA表达热图(G)~(I)及Kaplan-Meier生存曲线(J)~(L)。(M) STAD患者整个集的无进展生存期的Kaplan-Meier生存曲线。
Figure 2. Prognosis of the risk model in different groups
图2. 不同组的风险模型的预后
 (A) (B) 铜死亡相关lncRNA的单因素和多因素的独立预后分析。(C) 模型对1、3、5年生存率预测性能的AUC值。(D) 与年龄、性别和分期等临床病理特征相比,风险模型的预测准确性。(E) 风险模型的C指数曲线。
(A) (B) 铜死亡相关lncRNA的单因素和多因素的独立预后分析。(C) 模型对1、3、5年生存率预测性能的AUC值。(D) 与年龄、性别和分期等临床病理特征相比,风险模型的预测准确性。(E) 风险模型的C指数曲线。
Figure 3. Evaluate the prognostic ability of cuproptosis-related lncRNA risk model in patients with STAD
图3. 评估铜死亡相关lncRNA风险模型对STAD患者的预后预测能力
3.4. 列线图的构建和验证
创建了结合临床特征和风险评分的列线图,来预测STAD患者在1年,3年,5年的预后(图4(A))。校准曲线显示列线图与预测结果之间具有良好的一致性(图4(B))。
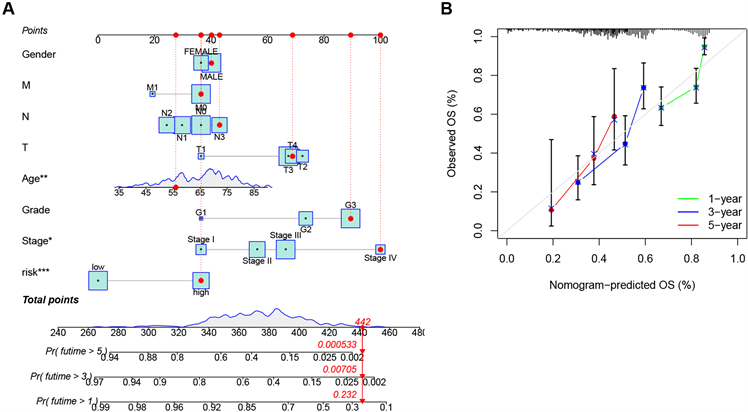 (A) 构建列线图预测STAD患者1年、3年和5年的生存期。(B) 校准曲线检测预测存活率与实际存活率的一致性。
(A) 构建列线图预测STAD患者1年、3年和5年的生存期。(B) 校准曲线检测预测存活率与实际存活率的一致性。
Figure 4. Construction and validation of the nomogram
图4. 构建并验证列线图
3.5. PCA、GO富集分析、KEGG富集分析
PCA用于探索高风险组和低风险组之间的差异,总基因、铜死亡基因、铜死亡相关lncRNA的表达谱分类的高低风险组之间无明显差异(图5(A)~(C)),而8种与铜死亡相关的lncRNA具有最好的区分低风险和高风险人群的能力(图5(D))。为了检测生物学功能的差异,比较了高风险组和低风险组患者的基因

 (A) 所有基因的PCA。(B) 铜死亡相关基因的PCA。(C) 铜死亡相关lncRNA基因的PCA。(D) 风险模型中lncRNA的PCA。
(A) 所有基因的PCA。(B) 铜死亡相关基因的PCA。(C) 铜死亡相关lncRNA基因的PCA。(D) 风险模型中lncRNA的PCA。
Figure 5. PCA of patients in high-risk group and low-risk group
图5. 高低风险组患者的PCA
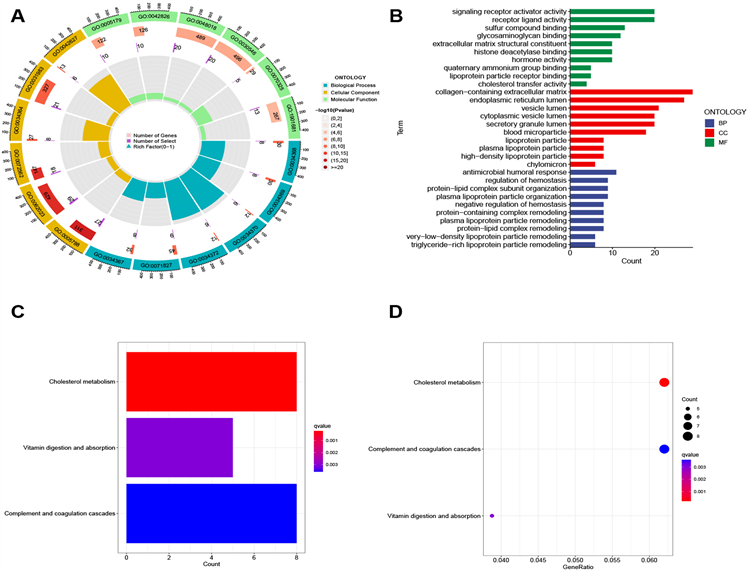 (A) (B) 高低风险组之间差异表达基因的GO富集分析结果。(C) (D) KEGG富集分析显示差异表达基因的显著富集的通路。
(A) (B) 高低风险组之间差异表达基因的GO富集分析结果。(C) (D) KEGG富集分析显示差异表达基因的显著富集的通路。
Figure 6. GO and KEGG enrichment analysis
图6. GO和KEGG富集分析
表达,获得了357个差异表达基因。GO富集分析表明差异基因与胶原蛋白(包括细胞外基质)以及内质网腔密切相关(图6(A),图6(B))。KEGG富集分析表明差异基因主要涉及胆固醇代谢(图6(C),图6(D))。
3.6. 免疫相关功能分析和肿瘤突变负荷
免疫相关功能分析热图显示,MHC I类通路、抗原提呈细胞的共抑制、T细胞的共抑制和细胞毒性的激活在高风险组与低风险组之间具有明显差异(图7(A))。从TCGA网站下载体细胞突变数据,分析高风险组和低风险组患者的体细胞突变变化。突变率最高的10个基因是TTN、TP53、MUC16、LRP1B、ARID1A、SYNE1、CSMD3、FAT4、FLG、PCLO (图7(B),图7(C))。并且,两组之间的肿瘤突变负荷具有差异(图7(D))。并且,与其他组相比,高肿瘤突变负荷和高风险组的患者预后最差(图7(E),图7(F))。
 (A) 高低风险组之间的免疫相关功能的差异。(B) (C) 高低风险组之间基因突变的瀑布图。(D) 两组之间TMB的差异。(E) 高TMB组和低TMB组之间的Kaplan-Meier生存曲线。(F) 四组之间的Kaplan-Meier生存曲线。
(A) 高低风险组之间的免疫相关功能的差异。(B) (C) 高低风险组之间基因突变的瀑布图。(D) 两组之间TMB的差异。(E) 高TMB组和低TMB组之间的Kaplan-Meier生存曲线。(F) 四组之间的Kaplan-Meier生存曲线。
Figure 7. Evaluation of immune related function and tumor mutational burden
图7. 评估免疫相关功能及肿瘤突变负荷
4. 讨论
STAD是胃癌的主要组织学类型,但目前的治疗方法使STAD的疗效仍不能令人满意,患者的生存期因人而异。因此,建立一个预测模型对STAD患者进行个性化的预后分析对于改善患者预后至关重要。
越来越多的证据表明,各种程序性细胞死亡方式与癌症的发展密切相关 [13] [14] [15]。铜死亡作为一种新的程序性细胞死亡方式,在2022年3月份被提出。细胞内铜的积累导致了脂酰化蛋白质聚集,和铁硫簇蛋白质丢失,进而引发蛋白质毒性应激,并最终致使细胞死亡 [4]。据报道,铜的摄取、转运及与铜依赖蛋白的结合对癌症的发展有重要作用 [16] [17] [18]。因此,铜死亡的深入研究有助于促进对肿瘤发病机制的新认识。
lncRNAs的功能和调控作用在胃癌中越来越得到认可。据报道,LncRNA MIR503HG通过miR-224-5p/ TUSC3级联信号调节未折叠蛋白反应的ATF6分支来抑制胃癌的进展 [19]。lncRNA MAGI2-AS3通过海绵化miR-141/200a和维持胃癌中ZEB1的过表达来促进肿瘤进展 [20]。细胞焦亡以及坏死性凋亡相关的lncRNA在胃癌患者的预后预测中的作用也有报道 [10] [21]。然而,迄今为止还没有研究评估铜死亡相关的lncRNA在胃癌中的作用。在这里,该研究构建了一个与铜死亡相关的lncRNA风险模型来预测STAD患者的预后。在该研究中,通过分析获得了18个与预后相关的铜死亡相关lncRNA。然后通过LASSO回归、多因素Cox回归分析筛选并鉴定了8个lncRNA (AC016737.1, AC008915.2, AC234775.2, DIRC1, STX18-AS1, HAGLR, AL513165.1, AC022113.1)引入到模型当中。其中,AC016737.1,DIRC1,STX18-AS1已被报道与胃癌密切相关 [22] [23] [24]。HAGLR则被报道在多种癌症的发展中发挥重要作用 [25] [26] [27]。而AC008915.2,AC234775.2,AL513165.1以及AC022113.1则是被首次报道。Kaplan-Meier生存曲线、ROC曲线以及c指数评估了STAD患者铜死亡相关lncRNA的预测准确性。可以发现风险评分比其他临床特征具有更强的预测能力。接下来,构建了一个列线图来预测STAD患者的预后。校准曲线显示列线图与预测结果之间具有良好的一致性。然后PCA结果表明,8个与铜死亡相关的lncRNA具有最好的区分低风险和高风险人群的能力。GO富集分析表明,高低风险组之间差异表达的基因与胶原蛋白(包括细胞外基质)以及内质网腔密切相关。KEGG分析表明,差异基因主要涉及胆固醇代谢。此外,免疫相关功能分析结果显示,MHC I类通路、抗原提呈细胞的共抑制、T细胞的共抑制和细胞毒性的激活在高风险组与低风险组之间具有明显差异。这些结果表明,铜死亡相关lncRNA可能与STAD肿瘤免疫微环境相关,通过抑制免疫而促进癌症的进展。此外,瀑布图显示,大多数基因在低风险组中的突变频率要高于高风险组。肿瘤突变负荷显示高低风险组之间的肿瘤突变负荷具有差异,低风险组的肿瘤突变负荷更高。并且,高风险与低TMB的叠加效益会使STAD患者的生存期进一步缩短。
该研究确定了8个具有预后价值的铜死亡相关lncRNA,并利用它们建立了预测STAD患者预后的模型,为STAD患者的个体化治疗提供了重要依据并为STAD的靶向治疗提供一定的科研依据和理论基础。
这项研究仍有一定的局限性。首先,该研究仅在TCGA数据库中验证了该模型,还需要更多的临床数据对其进行验证以确定它是否可以应用于临床患者。其次,铜死亡相关的lncRNA在癌症中发挥作用的机制尚不明确。因此,还需要进一步的研究以验证铜死亡相关lncRNA的价值。