1. 引言
乳腺癌作为全球最常见的肿瘤疾病,其治疗方式和药物研发进程一直备受关注。国际癌症研究机构(IARC)发布的数据显示,截至2020年中国女性新增癌症人群中,乳腺癌患者占五分之一,乳腺癌已经成为了女性中发病率最高的癌症。相关研究表明,乳腺癌的疾病越早被发现,患者存活的机会就越大,因此提高抗乳腺癌药物的生物活性是药物研发的关键问题。
经研究发现,雌激素受体α (ERα)作为转录因子,通过调节各种基因的转录来控制恶性肿瘤的发展 [1]。因此ERα被认为是乳腺癌增殖的重要受体,也是乳腺癌中一个重要的预后因素,能够拮抗ERα的药物对于乳腺癌的治疗是必要的 [2]。其次,ADMET性质也是药物研发过程中最关键的问题之一 [3],即吸收(Absorption)、分布(Distribution)、代谢(Metabolism)、排泄(Excretion)、毒性(Toxicity)。然而在药物研发期间,分析化合物分子的生物活性和ADMET性质费时且费力,因此通常采用建立化合物生物活性预测模型(QSAR)的方法来筛选潜在活性化合物,国际上已有很多学者依靠数据挖掘和机器学习方法解决这一问题。Zang Q. [4] 等(2013)使用随机森林特征选择方法提取与Erα拮抗剂活性最相关的结构特征,并使用SVM结合RF算法从大量描述符中筛选识别,构建QSAR模型。Zekri A. [5] 等(2020)基于多元线性回归MLR方法,设计了一个稳健可靠的QSAR模型来预测抗癌药物的活性。Chang K. [6] 等(2021)筛选出12个分子描述符,分别构建了基于BP神经网络、决策树和随机森林的复合ERα生物活性定量预测模型。认为随机森林模型可用于预测具有更好生物活性的新化合物分子。Babiker S. [7] (2020)等使用逻辑回归模型分析导致沙特妇女患乳腺癌的危险因素。
目前,针对抗乳腺癌候选药物的研究大多数倾向于预测活性,对药物活性与ADMET性质综合分析的文献较少;虽然近年有学者利用机器学习方法进行预测建模,但预测精度还存在提升空间。因此,本文构建集成学习XGBoost模型,建立化合物生物活性的定量预测模型,并采用粒子群算法,以药物活性达到最佳且至少满足三种ADMET性质为条件,求解化合物分子描述符的最优解。
2. 数据来源与处理
2.1. 数据来源
本文数据来源于DrugBank药物分子数据库,数据集包括1974个化合物的729个分子描述符和生物活性pIC50值。使用train_test_split函数来将1974个化合物以4:1划分为训练集和测试集,训练集样本数为1579,测试集样本数为395。在训练集上训练模型,再用测试集的数据来考察模型的预测效果。
2.2. 数据处理
由于数据表中分子描述符信息变量过多,在筛选并提取主要变量之前,需要将数据降维,弱化潜在的相关性。具体处理方法如下:
1) 缺失值处理:首先对数据进行预处理,将全为0的变量进行剔除,这相当于第一次的变量选择。最终剔除分子描述符变量225个,同时进一步针对化合物进行数据检测,发现没有其他缺失值。此时的数据为1974个化合物的504个分子描述信息。
2) 变量筛选:经过综合考虑各种降维方法的特点,本文采用随机森林实现对变量的筛选。随机森林在处理小样本量、高维特征空间和复杂数据结构方面具有独特的优势。对于每一个变量,随机森林的决策树都可以度量由该变量所导致的分裂准则函数(残差平方和或基尼指数)的下降幅度,然后针对此下降幅度,对每棵决策树进行平均,即为对该变量重要性的度量。最终依据随机森林方法进行变量重要性排序,选取了前20个变量作为建立ERα生物活性的定量预测模型的变量,所筛选的20个分子描述符见表1。

Table 1. Results of variable screening
表1. 变量筛选结果
3) 变量合理性评价:从变量降维流程来看,先删除数值为空的分子描述符变量,共删除225个变量,再采用随机森林法得到在对化合物的生物活性影响性排名前20的分子描述符。可视化结果见图1,颜色越深表明相关程度越大,容易导致模型估计失真和预测结果无效等问题。由图可知,20个变量之间的自相关性程度较低,表明化合物分描述变量具有较好的独立性,有利于下一步分析。
3. 预测模型构建
3.1. XGBoost活性预测
XGBoost是一种基于GBDT并进行了许多优化的极致梯度提升决策树算法 [8]。XGBoost能够通过对每个附加的新树进行优化,将弱学习器变成强学习器(提升)。将树修剪到定义的最大深度,然后再向后修剪,直至损失函数低于设定的值,从而使分类模型生成更少的误报、轻松标记数据和准确分类数据。其目标函数为:
其中:
,
,且
为样本 的一阶偏导数,
为样本
的二阶偏导数。
本文选取上文所筛选出的20个分子描述符变量的数据与变量pIC50的数据进行合并,形成一个1974 × 21的矩阵,再从中选取90%数据作为二次划分的训练集,而其余197组数据用来测试。用XGBoost算法进行拟合,并针对模型中的部分参数进行了改进:max_depth表示“树的最大深度”,设置为8;eta学习效率默认0.3,在每次提升后起到收缩步长的作用,得到新特征的权重,使提升过程更加具有鲁棒性;其他参数按照模型初始值设置。利用R软件编程实现的预测值和误差曲线如图2所示。
将得到的197组pIC50预测值与同组样本实际值做比较。图2表明,拟合值大体上与真实情况相近,虽然存在着个别偏离真实值较严重的样本点,但整体趋势一致,出现这种情况的原因可能是因为存在试验误差或受到了参数设定的影响。图3描绘了残差波动的情况,拟合误差在0值上下波动,模型误差分布比较集中,大部分样本的误差仅在[−2, 2]之间,说明XGBoost模型比较稳定。
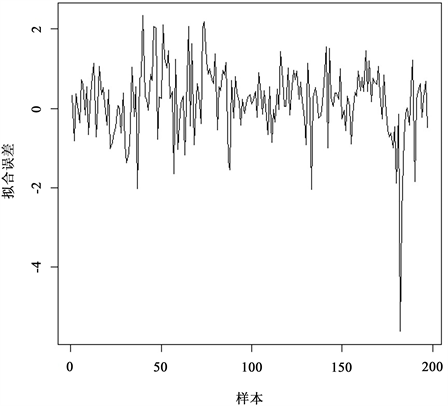
Figure 3. Residual plot of XGBoost fitting
图3. XGBoost拟合残差图
3.2. 模型评价
为了探究XGBoost算法的拟合效果,本文选取测试集中的30组样本,用上述方法计算了模型的拟合误差和精度,计算公式如下:
其中,
表示第
个样本的估计值,
表示第
个样本的实际值。通过计算每个测试样本的误差值,计算了模型的预测精度,具体数值如表2所示。通过验证测试集数据,预测趋势与真实趋势一致,误差在[0, 0.8]之间,XGBoost模型的预测效果较为良好,且预测精度较高。


Table 2. Evaluation table of algorithm prediction effect
表2. 算法预测效果评价表
4. 优化建模
4.1. ADMET性质样本选择
对1974个化合物依据Caco-2 (化合物的小肠上皮细胞渗透性)、CYP3A4 (化合物的代谢稳定性)、hERG (化合物的心脏毒性)、HOB (人体口服生物利用度)、MN (化合物的遗传毒性)这五个药代动力学性质进行筛选。
数据中CYP3A4为“1”代表该化合物能够被CYP3A4代谢,为“0”表示该化合物不能被CYP3A4代谢,而化合物能够被CYP3A4代谢表示药代动力学性质差;同时数据MN为“1”代表该化合物具有遗传毒性、药代动力学性质差,为“0”则代表该化合物不具有遗传毒性、药代动力学性质好。因此对数据进行处理,表3为满足不同ADMET性质个数的化合物样本数。其中,在给定的五种ADMET性质中,有543个化合物样本至少满足了三种性质。

Table 3. Properties of compound ADMET
表3. 化合物ADMET性质满足情况
4.2. 优化目标及约束设定
1) 决策变量
本文所建立的基于XGBoost算法的化合物对ERα生物活性的定量预测模型的主要变量主要有20个,因此该粒子群算法模型中共有20个决策变量,记为:
2) 目标函数及约束条件
本文的优化目标在于得出化合物的哪些分子描述符,以及这些分子描述符在什么取值或者处于什么取值范围时,能够使化合物对抑制ERα具有更好的生物活性,同时具有更好的ADMET性质。化合物的筛选满足了好的ADMET性质,因此本文以生物活性pIC50为主要的优化目标,即要求化合物的生物活性尽可能大。同时,考虑到提出的20个分子描述符在操作时需要给定范围,不能无限大或无限小,因此设定约束为:
另外,需要保证经过XGBoost预测的生物活性大于原来的生物活性值,于是提出约束:
所以目标函数及约束条件为:
其中,
表示化合物分子描述符与生物活性的XGBboost算法预测模型。
4.3. 粒子群算法参数设定
粒子群算法是一种受到群居动物集体行为的启发而产生的优化方法。假设一群粒子进入了函数的搜索空间,每个粒子在搜索时,需要考虑个人搜索的历史最佳位置和其他粒子搜索的全剧最佳位置。每一次搜索中,各粒子通过迭代来更新自己的位置和速度生成一个新的序列,再用新序列中的最优的粒子替换某个粒子的位置,从而搜寻全局最优解。主要变量设定及相关参数如表4所示。
4.4. 模型求解
针对上述建立的复杂多约束优化模型,本文使用R语言编写粒子群算法程序进行求解。在演化过程中,样本的平均迭代次数为16次,并未超过预设的参数,表明模型参数设置合理,模型求解性能较好,比较准确快速。本文对543个数据样本求解生物活性最大时的主要变量对应的取值。可求得在满足约束条件下,每个化合物分子描述符最优取值统计结果如表5所示。

Table 5. Optimal values of molecular descriptors
表5. 分子描述符最优取值表
在满足至少三种ADMET性质较好的前提下,pIC50活性值达到最大时,20个变量的最优解如表5所示,其中ATSp2的数值应为3535.7,ECCEN的数值为206,minHsOH、SsOH、maxsOH、nHBAcc四个变量的最优解为0,其余14个分子描述符的最优解分布在[0, 50]范围内,数值较接近。
通过观察每个化合物分子描述符最优取值,一方面可以为抗乳腺癌药物的设计提供理论支撑,另一方面也可为ERα拮抗剂的优化提供参考依据。该优化结果从理论上具备了可靠性和实用价值,能够减少研发成本和资源浪费,未来可用于设计具有更好生物活性的新化合物分子,对乳腺癌药物的研制具有一定的现实意义。
5. 结论
针对抗乳腺癌药物研发过程中的生物活性预测和求解化合物最优值问题,本文首先用随机森林方法降解数据维度,并对所有化合物的重要性排序,筛选出前20个对生物活性有显著影响的分子描述符,建立了分子描述符和生物活性之间的数学关系。基于选定模型,在保证较高生物活性和良好药物性质的前提下,确定各分子描述符的最佳取值。全文主要研究结果如下:
1) 本文自变量为候选药物的生物活性,因变量为构成化合物的20个分子描述符,用XGBoost模型拟合二者关系,通过观察预测精度的高低来检验模型是否合适。根据表2,选取的预测方法对数据拟合能力较强,估计误差在0值上下平稳波动,因此变量选择的结果较为合适且模型合理。
2) 以化合物生物活性最佳为目标,通过设定约束条件和粒子群参数值,计算了化合物分子描述符的具体值。取值结果如表5所示,其中ATSp2取值最大,为3535.7。大部分描述符的范围在[0, 50]区间内取值,此时能够使化合物在保证良好药代动力学性质下,对抑制ERα具有更好的效果。