1. 引言
Adcy3是一种在人体组织分布广泛的膜相关蛋白,催化腺苷三磷酸形成环磷酸腺苷,环磷酸腺苷是第二信使。腺苷酸环化酶家族由10个成员组成,在催化位点显示出高度的一级序列相似性 [1]。Adcy3基因最初在嗅神经上皮细胞和人胰岛细胞中被鉴定 [2] [3]。Adcy3基因突变小鼠表现出外周和行为性嗅觉障碍 [4]。
2018年1月,在《Nature Genetics》同一期上登出了三篇Adcy3基因的研究论文。Adcy3基因以往被认为是下丘脑调节机制中的一部分,可以帮助调节多种激素,而这些激素则参与多种生物功能的调节 [5]。在关于小鼠的研究表明发现,黑色素皮质素受体(melanocortin 4 receptor, MC4R)与Adcy3共定位在初级纤毛上。特异性地阻断含有MC4R神经元纤毛上的Adcy3,会导致小鼠肥胖,预示着人类肥胖可能也与Adcy3蛋白有关。而另外两项人类遗传学的研究另外两项人类研究证实了关于小鼠实验的研究结果,Adcy3功能缺陷的遗传变异会增加个体患肥胖和2型糖尿病风险,是导致单基因遗传严重肥胖的主因,在特定研究人群(巴基斯坦地区的儿童)中有超过30%的肥胖和Adcy3基因的隐形突变有关 [6]。一项关于胃癌组织样本基因转录本分析提示,在癌组织中的Adcy3表达同样发生了特异性改变。之后作者从细胞学水平上研究了Adcy3的过度表达对肿瘤的促进作用,并证实了Adcy3的上调与Lauren的肠型胃癌之间的显著相关性。Adcy3的过度表达增加了HEK293细胞的迁移、侵袭、增殖和克隆性;相反,SNU-216细胞中Adcy3的沉默表达降低了这些表型 [7]。这些结果都预示着深入了解Adcy3对肿瘤发生和发展机理的研究中也极为重要。目前很多疾病没有有效治疗药物的重要原因在于,对大多数疾病缺乏分子生物学机制的认识以及缺乏有效的药物作用靶点。无论是肥胖还是肿瘤都是目前非常热门的研究领域,可以预计,在今后的一段时间内Adcy3将会成为一个研究热点。
基因修饰小鼠是目前研究人类基因功能最重要的模式生物。本研究采用CRISPR/Cas9基因编辑技术开发一种新的Adcy3基因小鼠模型,为Adcy3基因在代谢性疾病、肿瘤发生发展中作用及机制奠定基础。
2. 材料与方法
2.1. 试剂、材料和动物
2.1.1. 质粒和动物
pX330-U6-Chimeric_BB-CBh-hSpCas9质粒(Plasmid #42230)、H2B-mCherry (Plasmid #20972)由美国Addgene公司提供、C57BL/6小鼠由南京医科大学动物中心提供。
2.1.2. 主要试剂
MESSAGE mMACHINE T7 Ultra Kit试剂盒(AM1345)购自美国Ambion公司,HiScribeTM T7 High yield RNA Synthesis Kit试剂盒(E2040S)、各种限制性内切酶、T4DNA连接酶购自英国NEB公司,长链Taq酶、高保真酶、胶回收及片段回收试剂盒由日本takara公司提供,小鼠组织DNA抽提试剂盒购自南京Vazyme公司。
2.2. 实验设计
2.2.1. CRISPR敲入方案的设计
针对小鼠Adcy3基因结构(图1),将mCherry基因敲入到小鼠Adcy3基因的3'端,通过Adcy3基因的3'端与mCherry的5'端连接,形成Adcy3-mCherry融合蛋白。通过在线sgRNA设计软件(http://crispor.tefor.net),针对最后一个外显子在靠近终止密码子TGA的位置附近设计sgRNA识别靶点(sgRNA序列和PAM序列见表1)。

Figure 1. Gene editing strategy for mouse Adcy3
图1. 小鼠Adcy3基因编辑策略

Table 1. Target Information for mouse Adcy3
表1. Adcy3基因目标靶点信息
2.2.2. mCherry Donor的设计
根据H2B-mCherry质粒中mCherry CDS区的完整序列,针对sgRNA2的作用位点,在mCherry CDS区5’和3’末端分别增加36 bp的同源臂,得到mCherry Donor序列。
2.2.3. 鉴定方案的设计
在Donor同源臂外侧50 bp以外,利用Vector NTI软件分别生成2对PCR巢式扩增引物。在mCherry CDS区内部生成一对短链鉴定引物。鉴定示意图见图2。

Figure 2. Mouse identification strategy for Adcy3
图2. Adcy3基因编辑小鼠鉴定方案
2.3. 实验方法
2.3.1. sgRNA的合成
针对3个sgRNA靶位点,选取靶点特异性MIT Specificity Score和CFD Spec. score平均值最高的sgRNA2合成相应的Oligo DNA,CFD off-target score显示,该靶点在12号染色体的外显子上不具备脱靶位点。采用重叠pcr制备sgRNA的T7转录模版,用在线软件(http://crispor.tefor.net)生成sgRNA2对应的重叠pcr扩增引物(sgRNA2-T7fw和T7rev common),见表2。PCR条件为:95℃预变性5 min;95℃变性20 s,60℃退火复性20 s,72℃延伸20 s,扩增35个循环。PCR产物用2%琼脂糖凝胶电泳检测。利用PCR纯化试剂盒纯化PCR扩增产物,溶于无核酸酶的水中作为sgRNA T7转录模板。使用HiScribe TM T7 High Yield RNA Synthesis Kit试剂盒转录及回收sgRNA,浓度测定后浓度调整到1000 ng/ul,分装保存于−80℃备用。
2.3.2. Cas9 mRNA体外转录
以pX330-U6-Chimeric_BB-CBh-hSpCas9质粒为模版,用长链Taq酶(takara)以引物CAS9-T7F和CAS9-T7R扩增,PCR条件为:94℃预变性5 min;94℃变性30 s,55℃退火复性30 s,72℃延伸5 min,扩增35个循环,产物大小4289 bp。切胶回收目的片段以无酶水洗脱获得Cas9 mRNA体外转录模板。体外转录并回收Cas9 mRNA,浓度测定后调整到1000 ng/ul,分装保存于−80℃备用。
2.3.3. mCherry Donor的设计与制备
用高保真酶(PrimerSTAR Max DNA Polymerase,Takara公司)以引物Adcy3-mCherry-F和Adcy3-mCherry-R PCR扩增H2B-mCherry质粒(美国Addgene公司提供),产物大小781 bp。PCR条件为:98℃变性10 s,55℃退火复性10 s,68℃延伸1 min,扩增35个循环。切胶回收目的片段,溶于无核酸酶的水,浓度测定后调整到1000 ng/ul,分装保存于−80℃备用。
2.3.4. 小鼠受精卵显微注射
将体外转录好的Cas9 mRNA、sgRNA和mCherry Donor加入无酶水中,分别调整至终浓度50 ng/ul、20 ng/ul和5 ng/ul。通过显微注射法将混合物注射到小鼠受精卵胞质中,次日将存活的受精卵移植到假孕受体母鼠内,至母鼠生产。
2.3.5. F0代小鼠的基因型鉴定
受精卵移植的假孕母鼠单笼饲养20 d后,观察出生情况。小崽长至7天,趾标法进行小鼠编号。剪下的脚趾以鼠尾组织标本试剂盒(Vazyme)提取基因组DNA。以mCherry-HK13 F和mCherry-HK14 R进行短链初筛,PCR条件为:95℃预变性5 min;95℃变性30 s,55℃退火复性30 s,72℃延伸1 min,扩增35个循环,小鼠扩增产物为555 bp。出现目的片段的样品以外侧引物(Adcy3-Outer-F和Adcy3-Outer-R)和内侧引物(Adcy3- Inner -F和Adcy3- Inner -R)进行巢式pcr,PCR条件为:94℃预变性5 min;94℃变性30 s,55℃退火复性30 s,72℃延伸1 min,扩增35个循环,正确的基因编辑基因型终产物大小为965 bp (wt为259 bp)。扩增产物以Adcy3-Inner-F和Adcy3-Inner-R送测序公司进行双向测序,确认插入序列的正确性。
2.3.6. F2代纯合子小鼠的获得和基因型鉴定
F0代首建小鼠性成熟后与C57BL/6野生型小鼠交配,获得可稳定遗传的Adcy3-mCherry F1代杂合子小鼠,再通过杂合子小鼠自交获得F2代Adcy3-mCherry基因敲除纯合子小鼠模型。鉴定引物与F0代小鼠的基因型鉴定方法相同。判定F2代纯合子的标准为巢式pcr无法扩增得到259 bp的目的片段。
3. 结果
Adcy3-mCherry基因小鼠的获得
Adcy3基因位于12号染色体上,包含21个外显子;4.41 kb mRNA转录物能够被翻译成129 kda蛋白。Adcy3基因小鼠构建策略如图1所示,通过显微注射方式将Cas9 mRNA、sgRNA和mCherry Donor注射到C57BL/6小鼠受精卵中,用体外转录获得的 Cas9 mRNA和sgRNA共注射了120枚受精卵,注射后的受精卵移植到5只假孕鼠中,其中3只小鼠妊娠并出生14只小鼠(出生率为11.6%),其中1只小鼠pcr鉴定正确。对应的Sanger测序显示Cas9/sgRNA复合物可将Adcy3基因终止密码子删除,细胞在mCherry Donor的同源臂为模版进行同源重组修复后,内源基因Adcy3的3'端在终止密码子与mCherry的5'端(ATG)正确连接,同时mCherry整个CDS区未见突变。当Adcy3-mCherry基因首建鼠生长至10周龄时与2至3月周龄C57BL/6雌鼠交配, F1代阳性小鼠Adcy3-mCherry基因型分析结果均和F0代相同。F1代杂合子小鼠经自交获得F2代纯合子小鼠,F2代小鼠基因型鉴定结果如图3所示。当检测不到野生型目的片段时(野生型为259 bp)可判断为纯合子,其中编号为80、83、87的三只小鼠为纯合子小鼠。
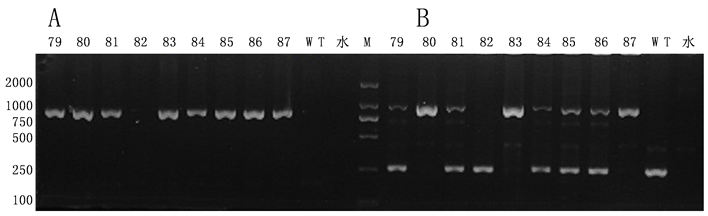 A:F2代小鼠短链筛选电泳图,B:F2代小鼠纯合鉴定电泳图。
A:F2代小鼠短链筛选电泳图,B:F2代小鼠纯合鉴定电泳图。
Figure 3. Identification results of Adcy3-mCherry gene knock in mice
图3. Adcy3-mCherry基因敲入小鼠鉴定结果
4. 讨论
随着生物学研究的发展,基因组编辑技术为探究特定基因功能提供了强有力的技术手段。CRISPR/Cas9基因编辑系统以绝对的优势取代了锌指核酸内切酶(zinc finger endonuclease, ZFN)和类转录激活因子的效应物核酸酶(transcription activator-like effector nuclease, TALEN) [8]。本研究应用CRISPR/Cas9技术,在制备sgRNA时采用重叠PCR的方法快速扩增得到转录模版,相对于传统方法,这种方法简单、快捷。传统的构建sg载体方法,经过构建载体、抽质粒测序及质粒模版扩增等一系列繁琐的操作,制备转录模版最快也要3~4天。而采用重叠PCR扩增,仅需3个小时就可以制备好转录模版进行转录操作 [9]。
在应用CRISPR/Cas9技术时,设计靶位点的软件有很多,本研究选用的是加州大学提供的在线sgRNA设计软件,这款软件的靶点输出界面支持靶点特异性分值(依据MIT特异性及CFD特异性算法预测)和切割效率进行分别排序,而且还可以非常简洁的方式筛选出靶点所在染色体的外显子上是否存在脱靶情况。关于特异性的预测,大多数的设计软件采用的是2013年麻省理工大学的算法,而根据Doench和Tycko等分别在2016年和2019年发表的论文显示,CFD特异性分值比MIT特异性峰值具有更高的准确性 [10] [11] [12]。本研究中,sgRNA2的CFD特异性得分也是三条候选靶点中最高的。
利用CRISPR/Cas9技术在靶点处引入精准插入目的序列的实验策略,需要考虑的一个重要因素就是donor该如何设计。根据Alexandre等人的研究结果,研究人员发现长度为33~38 nt的同源臂的PCR片段可以作为小鼠胚胎和人类细胞基因组编辑的有效供体,最佳的同源臂长度大约为35 nt [13]。作者指出线性DNA片段更适合作为供者DNA,而且它们在人细胞中编辑DNA的效率比环状质粒DNA高2~5倍,而且线性DNA通过PCR扩增法就可以直接制备,方法简单。对长度范围在57~993 nt的供者DNA序列测试得出的编辑成功率在10%~50%,相对而言短的供者DNA要比更长的供者DNA得到更高的编辑成功率,插入片段超过1000 nt供者DNA具有非常低的插入成功率。本研究插入的外源基因片段满足长度低于781 nt,采用36 bp的同源臂设计供体DNA,阳性率7.14% (1/14),与Alexandre等人的研究结果一致。本研究通过选用得到了1只首建鼠用于其后的建系并成功繁育得到纯合子后代,这一结果不仅为Adcy3基因在代谢性疾病、肿瘤发生发展中作用及机制的研究提供了动物模型,更为构建其他类似基因编辑动物模型提供了可借鉴的成功案例。
基金项目
江苏省自然科学基金(BE2019730),国家重点研发计划(2021YFF0702500)。
NOTES
*通讯作者。