1. 引言
重叠延伸PCR (overlap extension PCR, OE-PCR)是基因定点突变的传统方法 [1],已被广泛使用了三十多年。OE-PCR定点突变法的最大缺陷是操作繁琐,前后两轮PCR与电泳检测和胶回收操作还会增加野生型污染风险。虽然目前已开发出几种新的基因定点突变方法,如滚环扩增法和Gibson组装法等 [2] [3],但这些方法也各有弊端 [4]。滚环扩增法中若不能对原始质粒模板进行彻底消化则极易产生假阳性干扰;Gibson组装试剂盒价格昂贵,且克隆形成数偏少;故OE-PCR法并未被完全取代。
很多研究者尝试对传统OE-PCR法进行简化改进,提出了不少行之有效的优化方案 [5] [6] [7]。Andreas Urban等人曾提出一种基于平行模板的简化方案 [8]。利用含同一基因的两个不同质粒混合物作为平行模板,采用分别匹配两个质粒的正、反向引物和一对含目标突变的中介引物进行一步法OE-PCR (One-step overlap extension PCR, OOE-PCR)即可扩增出定点突变DNA (图1(a))。其理论基础是“无中介引物时,由于平行模板末端不互补,野生型DNA不会被扩增”;那么添加中介引物后扩增得到的DNA片段一定含有目标点突变。该简化方案也得到了很多科研工作者的支持 [9]。然而,我们在实验中发现无中介引物的OOE-PCR负对照往往能扩增出单一、明亮且大小正确的目标DNA条带 [10]。对于这一现象,我们曾分析了其可能原因,即不完全延伸DNA片段交叉互补,互相作为模板和引物合成完整基因进而继续扩增(图1(b))。该结论与OOE-PCR理论基础有所冲突,尽管如此,并不能彻底否定OOE-PCR法的价值。
常规OOE-PCR产物中目标基因突变型比例如何,它受哪些因素影响及如何控制等都有待进一步研究。为此,我们对中介引物浓度、基因长度、DNA聚合酶,PCR延伸时间等诸多OOE-PCR影响因素进行了对比分析,在此基础上提出一种两步法OE-PCR (two-step overlap extension, TOE-PCR)优化策略,并以克隆细胞周期蛋白E (cyclin E)的一个定点突变为例进行了验证。本研究旨在找出OOE-PCR定点突变法的效率制约因素并制定相应优化策略,以期提供一种更加高效的基因定点突变优化方案。
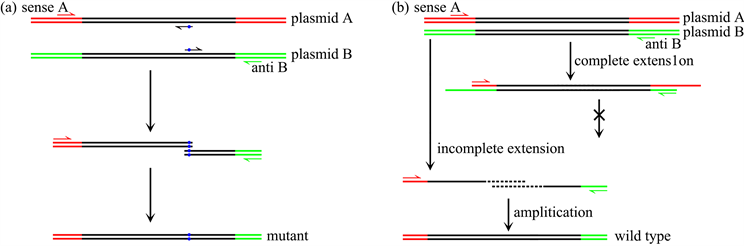
Figure 1. Schematic diagram of OOE-PCR based on parallel template. (a) Amplification of mutant gene by OOE-PCR in a single tube with the aid of parallel template and intermediate primers. (b) Wild type gene could be amplified with the aid of cross-complemented incomplete extension fragments in the absence of intermediate primers
图1. 基于平行模板的OOE-PCR原理示意图。(a) 中介引物辅助下利用平行模板在一个小管内一步法重叠延伸PCR扩增突变型基因。(b) 无中介引物下不完全延伸片段交叉互补可使野生型基因扩增
2. 材料与方法
2.1. 材料
感受态大肠杆菌DH5α为本实验室自制与保存 [11];pcDNA3.1质粒(V800-20)购自Invitrogen;pEGFP_C2质粒(#6083-1)购自Clontech;pcDNA3.1~与pEGFP_C2~系列质粒由本实验室构建。
Taq DNA Polymerase (AP111)购自北京全式金生物技术有限公司;2 × PCR Taq MasterMix (G013)购自abm;LA-Taq (RR02MQ)、Pyrobest DNA Polymerase (R005A)、rTaq DNA Polymerase (R001)、DNA Ligation Kit Ver.2.1 (6002)购自Takara;限制性内切酶AgeI (R3552S)、BamHI (R3136V)、EcoRI (R3101V)及XhoI (R0146V)购自NEB;DNA回收试剂盒(BSC02M1/BSC03M1),质粒小量抽提试剂盒(BSC01M1)购自杭州博日科技有限公司。
2.2. 引物设计与合成
引物采用Primer Premier 5.0软件设计,由北京奥科鼎盛生物科技有限公司合成,经PAGE纯化。引物序列见表1。

Table 1. Primers sequences for PCR
表1. PCR引物序列
下划线指示酶切位点。
2.3. 基因扩增与检测
常规PCR:4 μL 10 × buffer,4 μL dNTP,0.4 μL正向引物/0.4 μL反向引物,0.4 μL DNA聚合酶,2 ng质粒模板,补水至40 μL。94℃预变性5 min;94℃变性30 s,54℃退火30 s,72℃延伸1~3 min,30循环;72℃再延伸5 min。
OOE-PCR:4 μL 10 × buffer,4 μL dNTP,0.4 μL T7 Promoter/0.4 μL pEGFP_C3',0.4 μL正/反向中介引物,0.4 μL DNA聚合酶,2 ng pcDNA3.1~&pEGFP_C2~质粒模板混合物,补水至40 μL。94℃ 5 min;94℃变性30 s,54℃退火30 s,72℃延伸1~3 min,30循环;72℃再延伸5 min。
TOE-PCR:A管:2 μL 10 × buffer,2 μL dNTP,0.2 μL T7 Promoter/0.2 μL反向中介引物,0.2 μL Pyrobest DNA Polymerase,1 ng pcDNA3.1~质粒模板,补水至20 μL。B管:2 μL 10 × buffer,2 μL dNTP,0.2 μL正向中介引物/0.2 μL pEGFP_C3',0.2 μL Pyrobest DNA Polymerase,1 ng pEGFP_C2~质粒模板,补水至20 μL。94℃ 5 min;94℃变性30 s,54℃退火30 s,72℃延伸1~3 min,0~30循环;A、B管混合后94℃ 5 min;94℃变性30 s,54℃退火30 s,72℃ 1~3 min,30循环;72℃再延伸5 min。
从琼脂糖凝胶中回收目标DNA,根据条带亮度将其洗脱至20~50 μL纯水中。酶切:7.5 μL DNA回收物,2 μL 10 × buffer,0.5 μL内切酶,补水至20 μL;37℃孵育2 h。
2.4. cyclin EM2突变体的克隆
取目标DNA凝胶回收物17 μL (同时取pEGFP_C2质粒1 µg稀释到17 μL纯水中),加2 μL 10 × buffer,0.5 μL EcoRI/0.5 μL SalI,37℃孵育4 h。酶切产物直接回收至40 μL纯水中。采用DNA Ligation Kit Ver.2.1进行连接:DNA片段4.5 μL,载体0.5 μL,solution I 5 μL;16℃孵育30 min。连接产物直接转化感受态大肠杆菌DH5α,涂布于卡那霉素-LB平板。从过夜平板上随机挑取20个菌落,置于3 mL卡那霉素-LB培养基中继续培养7 h后提取质粒进行酶切检测:质粒DNA 7.5 μL,加2 μL 10 × buffer,0.25 μL AgeI/0.25 μL BamHI,补水至20 μL;37℃孵育2 h后1%琼脂糖凝胶电泳检测。任取1个阳性克隆进行序列测定,DNA测序由北京奥科鼎盛生物科技有限公司完成,采用DNAMAN 4.0软件进行序列比对。
3. 结果与分析
3.1. 中介引物浓度对OOE-PCR产物的影响
关于以平行模板为基础的OOE-PCR定点突变简化策略,既然无中介引物下依然可以扩增出目标基因,那么在添加中介引物时,终产物是其野生型还是突变型?为此我们以克隆CDK4629E突变体为例进行研究。正向引物T7 Promoter仅匹配pcDNA3.1-CDK4质粒,反向引物pEGFP_C3'仅匹配pEGFP_C2-CDK4质粒。中介引物在CDK4基因629位引入一个EcoRI位点,以便于后续对PCR产物进行快速酶切分析(图2(a))。
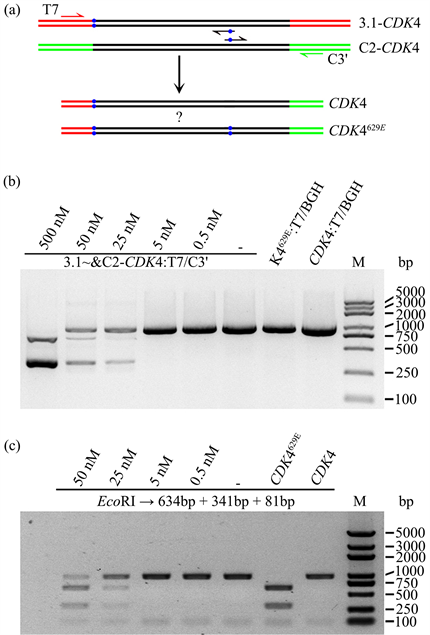
Figure 2. Effect of intermediate primers concentration on OOE-PCR products. (a) Gene map and primer matching sites. The blue dot indicates the EcoRI restriction site. (b) Amplification of CDK4629E by OOE-PCR with different concentrations of intermediate primers. The last two lanes were positive control with conventional PCR amplification of wild type CDK4 and its mutant. (c) EcoRI digesting result of the extraction from figure b. The expected result of CDK4629E mutant was shown in the up of the figure. M represents DNA marker
图2. 中介引物浓度对OOE-PCR产物的影响。(a) 基因图谱及引物匹配位点。蓝色圆点表示EcoRI酶切位点。(b) 添加不同浓度中介引物OOE-PCR扩增CDK4629E结果。最后2道为常规PCR扩增野生型CDK4及其突变体作对照。(c) b图目标条带回收物EcoRI酶切检测结果。CDK4629E突变体酶切预期变化如图所示。M为DNA标志物
电泳检测发现高浓度中介引物下只得到两个稍小片段,根据其大小推测为突变位点上、下游基因片段;随着中介引物浓度降低,上、下游片段产量随之减少,并出现全长CDK4基因产物;无中介引物时亦能扩增出全长目标基因(图2(b))。CDK4基因通过EcoRI/SalI双酶切插入载体,EcoRI位点会保留在PCR产物DNA前段。目标突变在629位新增一个EcoRI位点,故突变型基因被EcoRI酶切为三个片段,而野生型只会产生两个片段。对上述全长基因片段回收物作酶切分析,发现高浓度中介引物下的OOE-PCR产物大部能被切开,但仍保留一定量全长基因片段,提示该PCR产物中仍含有少量野生型基因。降低中介引物浓度,则突变型比例减少,野生型比例增加;至5 nmol/L (即常规PCR引物浓度1/100)时,已不见突变型基因存在(图2(c))。经反复调整PCR反应条件,仍未能得到任何一道彻底去除野生型干扰的终产物(结果未显示)。
3.2. 延伸时间对OOE-PCR的影响

Figure 3. Effect of gene length on OOE-PCR products. (a) OOE-PCR results with different DNA polymerase in the absence of intermediate primers. (b) OOE-PCR results in different elongation time in the absence of intermediate primers. (c) Amplification of genes in different length with OOE-PCR in the absence of intermediate primers. (d) Amplification of p16 and p21 genes with OOE-PCR in the presence of intermediate primers. All their primers were T7 and C3', M stands for DNA marker
图3. 基因长度对OOE-PCR产物的影响。(a) 不同DNA聚合酶进行无中介引物OOE-PCR结果。(b) 不同延伸时间下无中介引物OOE-PCR结果。(c) 不同长度基因进行无中介引物OOE-PCR结果。(d) p16与p21两基因进行有中介引物OOE-PCR结果。以上引物皆为T7/C3',M表示DNA标志物
OOE-PCR中野生型基因产物的干扰在于不完全延伸形成的DNA片段交叉互补,找到影响不完全延伸的原因并加以解决即可改善OOE-PCR定点突变效率。这里涉及到DNA聚合酶体外持续合成能力、PCR反应延伸时间、DNA长度等影响因素。选用5中常见PCR专用DNA聚合酶进行对比分析,发现无中介引物下它们都能顺利扩增出目标基因(图3(a))。DNA聚合酶体外合成速度可达2 kb/min以上 [12],常按1 kb/min保守估计PCR反应延伸时间。选用高保真DNA聚合酶pyrobest扩增CDK4基因,在不添加中介引物的情况下,延伸时间增加到3 min仍能扩增出目标基因(图3(b))。采用其他DNA聚合酶亦得到同样结果(结果未展示),说明延长PCR反应延伸时间不能削弱平行模板的交叉互补。
如果不完全延伸片段是DNA聚合酶随机中止的结果,那么很容易推测长基因OOE-PCR更容易出现平行模板交叉互补。选用7个长度各异的细胞周期相关基因(表2)进行无中介引物OOE-PCR检测,发现p16与p21两个小基因组未见目标产物,p27以上长基因组皆能扩增出目标片段(图3(c))。在p16与p21组添加中介引物,当其浓度增加至50 nmol/L (正、反向引物浓度1/10)时出现目标产物,并伴有少量上、下游基因片段(图3(d))。该结果与OOE-PCR原构思相符,只是中介引物浓度略高。似乎短基因不易交叉互补,而更适合直接OOE-PCR克隆定点突变。
3.3. 两步法对OOE-PCR的优化效果
为减少OE-PCR过程中不完全延伸片段产量,尝试进行TOE-PCR,即先在两个小管中扩增突变位点上、下游片段,再将两管直接混合继续运行PCR程序扩增基因突变体。首先进行无中介引物验证,发现第一轮PCR循环15次时,目标基因产量明显降低;至20循环以上时,已看不到明显的目标产物主带(图4(a))。以此为基础,添加适量中介引物对比TOE-PCR与OOE-PCR差异,其中TOE-PCR第一轮运行15次循环。电泳检测发现低中介引物浓度下,TOE-PCR产物量较少;至50 nmol/L时两者产物量相当(图4(b))。回收两种方案下目标产物进行酶切检测,发现低中介引物浓度下,两种方案的产物均为基因野生型;至50 nmol/L时,出现被EcoRI切开的突变体,且TOE-PCR方案下几乎不见野生型残留(图4(c))。对比上述结果,发现TOE-PCR方案添加50 nmol/L (即常规PCR引物浓度1/10)中介引物,第一轮PCR运行15次循环可得到理想目标产物,称之为“优化的TOE-PCR方案”。
采用OOE-PCR与优化的TOE-PCR方案在1/10中介引物下扩增其它基因定点突变,发现对于p16和p21两个较短的基因,两种方案的产物相差无几;对于稍长一些的其它基因,TOE-PCR方案上、下游片段量少,而目标产物量略多(图4(d))。说明优化的TOE-PCR方案可以减少副产物的干扰,增加目标DNA产量。对各组目标产物进行酶切检测鉴定其构成,发现短基因p16和p21在两种方案下均为突变型;其它基因在OOE-PCR方案下仍有一定量野生型,而TOE-PCR方案下野生型比例有明显减少(图4(e))。说明对于长基因定点突变,优化的TOE-PCR方案优势更加明显。

Figure 4. TOE-PCR is better than OOE-PCR. (a) Amplification of CDK4 gene by TOE-PCR. The number of PCR cycles in of first round PCR was shown on top of figure. (b) A comparison of OOE-PCR and TOE-PCR in the amplification of CDK4 gene. (c) EcoRI digesting result of the extraction from figure b. (d) Amplification of other gene mutants with different lengths by OOE-PCR and TOE-PCR. (e) The restriction results of the extraction from figure d. Wild type cyclin E could be cut off by AgeI, while the mutant of other groups cut off by XhoI
图4. TOE-PCR优于OOE-PCR。(a) TOE-PCR扩增CDK4基因,第一轮PCR循环次数如图所示。(b) OOE-PCR与TOE-PCR扩增CDK4基因对比。(c) b图对应目标产物EcoRI酶切检测结果。(d) OOE-PCR与TOE-PCR扩增相关基因突变体的对比效果。(e) d图相应目标产物的酶切检测结果。cyclin E组野生型可被AgeI切开,其它组均为突变型可被XhoI切开
3.4. 两步法OE-PCR克隆cyclin EM2的实践
为进一步检测优化的TOE-PCR方案在基因定点突变中的可行性,以克隆cyclin EM2突变体为例进行验证。cyclin EM2在其CDK结合结构域内发生FDRYM [156~160] AAAAA突变,从而失去CDK2结合能力,常用于研究cyclin E/CDK2复合物功能 [13]。
首先采用优化的TOE-PCR方案扩增cyclin EM2基因片段,同时以不同浓度中介引物进行OOE-PCR对比分析。电泳检测发现中介引物浓度增至50 nmol/L后目标DNA产量降低,而TOE-PCR明显增加了同等条件下的目标DNA产量(图5(a))。由于cyclin EM2将原序列TTTGACCGGTATAT突变为GCTGCCGCGGCTGC,破坏了其中的AgeI位点,因此可通过酶切鉴定PCR产物构成。对上述PCR产物作AgeI酶切分析,发现TOE-PCR产物中能被切开的野生型片段明显减少,提示其中绝大部分都是突变型(图5(b))。将TOE-PCR产物插入pEGFP_C2载体,筛选出上百个菌落(图5(c)),与常规分子克隆经验值相符 [14]。从平板上任意挑取20个菌落,小提质粒作AgeI/BamHI酶切检测。目标突变质粒切出888 bp与1132 bp两个小片段,而野生型质粒切出401 bp、731 bp与888 bp三个小片段。结果显示得到18个突变克隆,1个野生型克隆,另有1个推测为空载体(图5(d))。阳性率90%,结合经验值及对比其它基因定点突变方案,仍属于优秀水平 [4] [15] [16]。取#1号克隆作序列测定,序列比对结果显示其突变位点与目标序列完全相符,且其它位置无非预期突变(图5(e))。说明成功克隆了cyclin EM2突变体,并已将其插入pEGFP_C2载体。

Figure 5. Clone cyclin EM2 mutant with TOE-PCR. (a) Amplification of cyclin EM2 mutant by OOE-PCR or TOE-PCR. (b) AgeI digesting result of three designated PCR products from figure a. (c) Bacterial colonies on plate after transformation of recombinant plasmid. (d) Restriction results of selected clone from plate C. (e) Multiple alignments between #1 positive clone and its expected sequence. The red boxes show the target sequence
图5. TOE-PCR方案克隆cyclin EM2突变体。(a) OOE-PCR与TOE-PCR扩增cyclin EM2突变基因。(b) a图3个关键PCR产物的AgeI酶切检测结果。(c) 重组质粒转化大肠杆菌后形成的菌落。(d) c图平板菌落酶切检测结果。(e) #1号阳性克隆与目标序列比对结果。红色方框显示目标序列
4. 讨论
利用含有同一基因的两个不同质粒混合物作为平行模板,采用OOE-PCR方案是对常规OE-PCR定点突变法的简化。而实际上由于PCR过程中产生的不完全合成片段交叉互补,导致无中介引物时也能扩增出全长基因,即OOE-PCR方案负对照不能完全成立 [10]。添加含突变位点的中介引物有助于合成目标基因突变体;但中介引物必须稀释,否则只会(或主要)合成突变位点上、下游DNA片段副产物。调整中介引物稀释比至1/10时,得到较理想的目标产物主带,但其中仍含有一定比例野生型。故单纯调整中介引物浓度虽可改善产物构成,但无法根本克服该方案的缺陷。
消除或减少反应过程中不完全延伸DNA片段可以提高OOE-PCR终产物中基因突变型比例,这里涉及多种影响因素。常见热稳定性DNA聚合酶有快速型与高保真型两类,从选用的几个代表性酶来看,两者效果差异不大。高保真型PCR酶在无中介引物OOE-PCR中似乎更容易导致交叉互补,这对于基因定点突变是不利的。单纯延长PCR反应延伸时间也毫无作用,但是基因长度对结果却有显著影响,其中短基因更不易交叉互补。这种现象可能与DNA聚合酶的持续合成能力有关。虽然PCR酶体外持续合成能力可达数十kb [17],但仍有可能在更短范围内中止,由此形成不完全延伸片段。基于上述实验结果,很容易推测PCR过程中DNA聚合酶存在随机中止,则对于长基因更容易形成不完全延伸片段,故而发生无中介引物下的平行模板交叉互补。
TOE-PCR是一种不错的改进策略,既可以增加重叠延伸之前含点突变的上、下游片段含量,也可以“补齐”不完全延伸片段副产物。关于中介引物浓度和第一轮PCR循环次数两点,我们发现中介引物作1/10稀释效果较好,而后者可视基因长度而调整。对于1 kb左右的基因,虽然无中介引物时第一轮PCR循环20次以上产物主带才会消失,但添加中介引物后循环15次时TOE-PCR产物中已几乎不见野生型基因。短基因可减少第一轮PCR循环次数,甚至直接OOE-PCR即可;长基因则应适当增加第一轮PCR循环次数。
我们以优化的TOE-PCR方案克隆cyclin EM2定点突变,筛选得到90%克隆阳性率;在克隆其它基因定点突变时,往往也有相当高阳性率(结果未显示)。实际上,各种基因定点突变方法都存在多种原因的野生型干扰风险。如滚环扩增法中原始质粒未能彻底消化,或OE-PCR法回收物中污染的痕量原始质粒在第二轮PCR时扩增等,都会造成一定比例的野生型干扰。相较而言,我们认为TOE-PCR方案阳性率并不亚于其它定点突变方法。相比常规OE-PCR定点突变,TOE-PCR方案操作步骤有所简化。虽然需要两轮PCR反应,但第二轮PCR反应物只是第一轮产物的简单混合,中途无需电泳检测和胶回收。前提是实验室准备有插入同一目标基因的不同载体以及匹配不同载体的通用引物。我们认为TOE-PCR方案在克隆某基因的多个定点突变时优势较明显。
基金项目
湖北文理学院大学生创新创业训练计划项目(X202210519015, X202210519065)资助。
NOTES
*通讯作者。