1. 引言
随着大数据时代的到来,以及计算能力和其他现代技术的快速发展,在当代许多研究中经常出现复杂的高维数据。在遗传学、生物学等各种科学研究领域存在大量高维数据。对于传统的学习模型,由于使用大量的特征,模型往往会过拟合,导致模型性能下降。而且过多的数据也会显著增加数据分析的内存需求和计算成本。降维是解决前面描述的问题的最强大的工具之一,它主要可分为特征提取和特征选择两个主要组成部分 [1] 。特征提取和特征选择都具有提高学习性能、提高计算效率、减少内存存储、建立更好的泛化模型等优点,因此都被认为是有效的降维技术。相对于特征提取,特征选择保持了原始特征的物理意义,并给模型提供了更好的可读性和可解释性,因此,特征选择在各种数据挖掘和分析中有更多的应用。通过特征选择去除这些特征可以减少存储和计算成本,同时避免了信息的重大损失或学习性能的下降。
特征选择方法根据数据集中可用的信息,可分为监督、半监督和无监督 [2] 的方法。监督的方法需要标记数据来识别和选择相关特征,半监督方法只要求一些数据被标记,而无监督特征选择方法则不需要被标记的数据集。对于无监督的特征选择方法,常用的有三种方法,即包装方法、嵌入式方法和过滤方法 [3] 。包装的特征选择方法的选择取决于分类器的性能,该方法直接利用分类算法对特征选择得到的特征子集进行评价,目的是选择最有利于给定分类器性能的特征子集。对于包装的方法,Hancer [4] 等人提出了一种基于变串长度的多目标差分进化方法(MODE-CFS)来同时进行聚类和特征选择,为了评估解的优劣,MODE-CFS在最小化WB指数的同时最大化Silhouette指数和所选特征的数量。嵌入式的特征选择方法也取决于分类器,它将特征选择过程与学习者训练过程相结合,两者在同一优化过程中完成,即在分类器训练过程中自动进行特征选择。对于嵌入式的方法,Zhu [5] 等人提出了判别嵌入无监督特征选择方法(DEUFS),该方法通过最大化聚类之间的异质性来获取聚类标签,从而对数据的聚类结构进行建模。过滤的特征选择方法独立于特定的分类器,该方法首先选择数据集的特征,然后训练分类器。对于过滤的特征选择方法,Solorio-Fernández [6] 等人介绍了一种用于混合数据的单变量无监督光谱特征选择方法,称为USFSM(混合数据的无监督光谱特征选择方法)。USFSM通过分析当每个特征分别从整个特征集中排除时,归一化拉普拉斯矩阵的第一个非平凡特征值的谱分布(谱间隙)的变化来评估特征,最后特征根据各自的光谱间隙值按降序排序。由于包装器特征选择方法是针对给定的分类器进行直接优化的,因此包装器特征选择方法的性能优于过滤的方法,但包装器的计算成本通常远高于过滤的方法,且过滤的方法很容易扩展到高维数据集,因此具有计算简单、快速,计算成本低和可拓展性等优点。
Haindl [7] 等人提出了一种基于过滤的特征选择方法,引入了一种基于互相关性(Mutual Correlation)的特征选择方法。该方法首先采用皮尔逊相关系数计算数据中特征之间所有可能的成对相关性,然后删除与其他特征平均依赖性最高的特征,对于所有特性,重复此过程,直到选择需要的特征数量,他们的实验结果表明,虽然筛选效果不如包装的方法,但也实现了较好的筛选,且训练更快。然而皮尔逊相关系数只能度量变量间的线性相关性,会忽略变量间的非线性相关性。互信息是对一个随机变量包含另一个随机变量的信息量的度量,也可以描述为在了解另一个随机变量的情况下,降低了原始随机变量的不确定性,互信息可以很好的度量特征间的线性和非线性关系。本文利用互信息的概念来度量特征和特征之间的关系,建立基于互信息的无监督特征选择方法。
2. 特征选择方法
2.1. 互信息的估计
互信息 [8] 是两个随机变量之间的相互依赖性度量,对于任意两个连续随机变量X和Y,其互信息为:
(1)
其中
分别为x,y的概率密度函数,
是x和y的联合概率密度函数。对于任意两个离散随机变量X和Y,互信息则为:
(2)
的值总是非负的,当且仅当X和Y独立时
。
设
是来自样本量为的随机向量
的样本。假设
具有连续的概率密度函数,记
分别为x,y的概率密度函数,
是x和y的联合概率密度函数。由于对于两个连续随机变量X和Y的互信息计算时既含有积分是运算,又含有未知的密度函数,因此,直接对其估计是十分困难的,再由随机变量的数学期望公式,对于两个连续随机变量X和Y的互信息可以写成如下形式。
(3)
因此,当
存在时,两个连续随机变量X和Y的互信息可以考虑采用下式来进行估计:
(4)
其中
,
和
分别是
和
的非参数核密度估计,具体的有:
(5)
另外有:
(6)
(7)
其中,
和
为核密度估计的窗宽,控制着其光滑程度,本文根据拇指法则选取
,其中
为数据集的样本标准差。
为核函数,本文取高斯核函数为:
(8)
2.2. 基于互信息的方法
设有n个p维的特征向量来自k个类别,矩阵表示为:
(9)
两个变量
和
的互信息量定义为:
(10)
该互信息值可由上述介绍方法进行估计,即
(11)
然后估计所有特征对的所有互信息值,并计算一个特征在特征上的平均绝对互信息:
(12)
计算平均互信息值最大的特征:
(13)
在特征选择算法的每个迭代步骤中每次的平均互信息值最大的特征将被删除。当特征从特征集中移除时,它也从剩余的平均相关性中被丢弃,即:
(14)
2.3. 无监督的特征选择方法
根据上述描述,建立基于核密度互信息(Kernel Density Mutual Information, KMI)的无监督特征选择方法,所提出的方法的基本步骤如下:
Step 1. 初始化
,计算变量间的互信息值矩阵I;
Step 2. 根据(13)式计算平均最大互信息值;
Step 3. 丢弃平均互信息值最大的特征;
Step 4. 更新
,如果
,则返回生成的个特征子集D并停止,否则,执行Step 5;
Step 5. 根据式(14)计算剩余特征的平均互信息。
Step 6. 返回Step 3。
该算法从原始数据集中产生最优的维特征子集
:
3. 实例分析
3.1. 数据集
为了检验所提方法的有效筛选性能,将检验该方法在两个数据集(二分类数据集和多分类数据)上的筛选性能,二分类数据集采用Golub [9] 等人所得到的白血病数据集(ALLAML数据集),数据集包含72个样本和7129个特征,其中响应变量cancer包含两类,分别为急性髓系白血病(AML)共25例和急性淋巴母细胞白血病(ALL)共47例,将其转换成数值型变量,即0-1变量,其中标签0表示未患急性淋巴母细胞白血病(ALL),标签1表示患急性髓系白血病(AML)。多分类数据集采用UCI的肺癌数据集(lung数据集),该数据集包括203个样本和3312个特征,具有5个连续值标签,即为5分类的数据集。这两个数据集都可从https://jundongl.github.io/scikit-feature/datasets.html下载获取,两个数据集的参数汇总如表1所示:

Table 1. Summary table of dataset parameters
表1. 数据集参数汇总表
3.2. 实验设置
将两个数据集分别应用于本文所提出的筛选方法,其中保留的特征数量参考Fan和Lv [10] 等人所建议的特征个数,即
,其中
表示取a的整数部分,n为样本量,然后将降维后的数据集分别用支持向量机 [11] (Support Vector Machine, SVM)、随机森林 [12] (Random Forests, RF)和最近邻 [13] (K-Nearest Neighbors, KNN)建立分类模型,来评估特征选择方法的性能。在上述方法中,最近邻将K设置为3,支持向量机使用线性核,随机森林采用五折交叉验证且树的数量为500棵。
实验分为三部分,第一部分是数据处理部分,为了消除误差,确保计算的有效性,将数据进行了标准化处理,然后将80%的数据作为训练集,20%的数据作为测试集。第二部分为模型建立部分,分别建立SVM、RF和KNN模型,每个模型分别训练10次,特征个数从2增加到D。第三部分为模型评估部分,在本实验中,我们使用10次的平均分类精度和平均F1来评估特征子集上的分类效果。分类精度是指正确分类的样本数量占样本总数的比例。其中F1的定义如下:
(15)
其中P为精确率,R为召回率,其计算公式分别为:
(16)
(17)
上述两式中TP、FP、TN和FN分别为真正例、假正例、真反例和假反例。分类精度并不适用于所有情况,特别是在不平衡的数据集中,在某些情况下,F1值等可能更加全面地评估模型的性能。F1可以用来较好的度量二元分类问题,当类别数大于2时,可以使用宏平均F1将多类分类问题的F1值视为个二元分类的平均F1值。宏平均F1定义为:
(18)
其中
和
分别为宏观精度率和宏观召回率,计算公式分别为:
(19)
(20)
其中
为第i个类别的精度,
为第i个类别的召回率,k表示类别的数量。
3.3. 实验结果与讨论
3.3.1. 分类精度
为了证明本文提出的KMI算法的筛选性能,本节将文中提出的KMI算法与Haindl [8] 等人提出基于互相关性的方法进行了比较。分别使用RF、SVM和KNN分类器对两种特征选择算法筛选的特征进行训练,并利用分类精度来衡量不同特征选择算法的分类性能。
表2记录了KMI算法与基于互相关性算法两种特征选择算法在ALLAML数据集和lung数据集上的平均分类精度。在每个方法中用粗体标记出最大的平均分类精度。由表2可知,在ALLAML数据集集中,采用KMI方法进行变量筛选后,RF分类模型的平均分类精度达到了76.0%,而在其余两中方法上,由KMI方法筛选后比用互相关性方法筛选后建立的分类模型效果略低,且在三种分类模型中,SVM分类器的效果最好,分别达到了79.3%和78.0%。在lung数据集中,分类模型RF和KNN在使用KMI方法筛选后的分类效果都比使用互相关性的方法筛选后的效果要好,因此,与基于互相关性方法相比,KMI算法更能提高分类模型的性能。

Table 2. Classification accuracy of three classification algorithms on two data sets
表2. 三种分类算法在两个数据集上的分类精度
为了观察特征子集大小对分类精度的影响,比较两种不同特征选择算法在不同保留特征数下的分类精度,绘制了在两个数据集上三个分类方法保留特征数的分类精度图,其横轴表示特征子集中保留的特征数量,纵轴表示分类精度,在图中,使用不同的颜色来区分不同的特征选择方法。
图1绘制了三种分类方法在ALLAML数据集上的平均分类准确率图,图2显示了三种分类方法在lung数据集上的平均分类准确率图,由两图可知,模型的分类精度随保留变量的数量增大而逐渐提升,在lung数据集中,当保留变量个数在25个前,模型的分类精度增加较快,当保留变量个数达到25个后模型的分类效果呈缓慢的状态增加,且当保留变量数为25个时在三个分类模型中的分类准确率已经接近90%,且从整体上看,基于KMI方法的分类效果要优于基于互相关性方法的效果。而在ALLAML数据集中,当保留变量数高于15个时,基于KMI方法的分类效果总体略高于基于互相关性的方法。

Figure 1. Average classification accuracy of three classification methods on the ALLAML dataset
图1. 三种分类方法在ALLAML数据集上的平均分类准确率
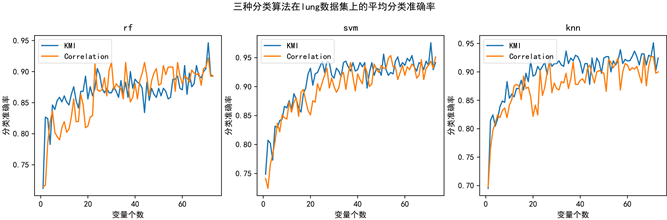
Figure 2.Average classification accuracy of three classification methods on the lung dataset
图2. 三种分类方法在lung数据集上的平均分类准确率
3.3.2. F1值
F1值是精确率和召回率的调和平均值,适用于类不平衡问题的性能评价。为了评价本文提出的KMI算法的筛选性能,我们使用F1值来评价在二分类数据集中的分类器的结果,使用宏F1值来评价在多分类数据集中的分类器的结果,表3记录了KMI算法与基于互相关性的方法两种特征选择算法在ALLAML数据集上的平均F1值和lung数据集上的宏平均F1值。由表三数据可知,在ALLAML数据集上,RF和SVM两个分类模型在KMI算法上的性能更好,在KNN上略低于基于互相关性的方法,在lung数据集上,三个模型在基于KMI上的F1值都比基于互相关性的方法要好。更进一步说明了KMI算法更能提高分类模型的性能。

Table 3. F1 values of the three classification algorithms in the two datasets
表3. 三种分类算法在两个数据集F1值
同时也绘制了在不同保留特征数量下三种模型的平均F1值和宏平均F1值,以比较两种不同特征选择算法在保留特征数条件下F1值的变化,如图3和图4所示,图3显示了三种分类方法在ALLAML数据集上的平均F1值,图4绘制了三种分类方法在lung数据集上的宏平均F1值。由图可知,其变化规律同三个分类模型的分类精度变化趋势类似。
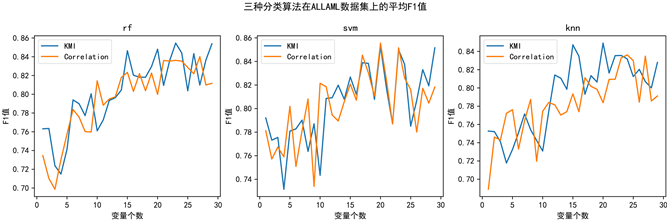
Figure 3. Average F1 value of three classification methods on the ALLAML dataset
图3. 三种分类方法在ALLAML数据集上的平均F1值
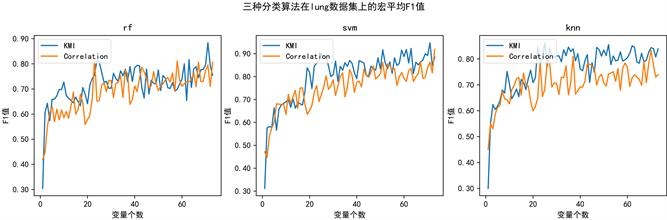
Figure 4. Macro_F1 values of the three classification methods on the lung dataset
图4. 三种分类方法在lung数据集上的宏平均F1值
4. 总结
本文主要研究了基于核密度互信息的无监督特征选择算法。研究的目标是去除特征之间冗余度较高的特征子集,以实现降维。作为一种过滤方法,它产生独立于要使用的分类器的特征。同时该方法引入互信息作为特征之间的相关性度量,可以很好的度量特征间的线性和非线性关系,采用核密度估计的方式估计特征的密度函数,可以对互信息更为准确的计算。文章通过两个高维数据集验证了所提方法的筛选性能,并将其与现有的基于互相关性方法作对比,最后将筛选后的数据应用于三个常用的分类模型,用分类精度和平均F1值对分类模型进行评价,实验结果表明,在两个数据集上,两种筛选方法在三个分类模型中的分类精度表现类似,而基于核密度互信息的方法筛选后应用三种分类算法的平均F1值比基于互相关性的方法表现更好,表明了本文所提方法具有更好的筛选性能。