1. 引言
结直肠癌(Colorectal cancer, CRC)是最常见的癌症之一,据相关统计,在2022年,中国有592,232例新发病例和309,114例死亡 [1] 。息肉作为最明显的CRC前体之一,可以通过筛查发现并在其发展为癌症之前移除。目前,内镜筛查作为筛查结直肠相关病变的金标准,可以提供结直肠息肉的外观和位置信息,帮助医生早期诊断和治疗。然而,医生的主观性和经验,以及高昂的人工成本会导致临床效果不佳。因此,计算机辅助诊断有利于克服这些局限性,减轻医生负担,提高诊断效率,防止误诊漏诊。目前人工智能研究已经取得了很好的进展,但由于结肠息肉颜色形态不一、病灶组织与背景差异性难以区分等问题,导致息肉分割的学习模型很难去鉴别,在息肉的自动分割上仍具有挑战。
近年来,卷积神经网络(Convolutional Neural Network, CNN)已经成为医学图像分割的主流 [2] [3] [4] 。以CNN构建的息肉图像分割算法,能够很好的发挥其局部提取空间和通道特征的能力 [5] 。U-Net [6] 在编码器中通过卷积层提取特征,并在对称解码路径中恢复空间信息。U-Net的U型结构已成为医学图像分割的一种经典网络架构。进一步将U-Net++ [7] 和ResUNet++ [8] 引入到息肉分割中,以减少U型网络编码器和解码器之间的语义差距,并产生一些可接受的结果。这些CNN所采用的编码器-解码器架构可以有效地消除传统手工制作的基于特征方法的局限性,但是CNN提取特征的时候容易忽略全局上下文信息,无法学习远程依赖关系。息肉的形态不一且与边界周围内在联系紧密,导致卷积网络在特征提取时会忽视一些信息或产生不准确的预测。针对CNN的缺点,引入注意力机制可以有效抑制息肉背景噪声,提升分割的准确率。Transformer有很好的全局上下文建模能力,能够有效的学习特征的远程依赖性。但其对于局部低层次细节信息获取不充分,因此会导致分割结果粗糙。目前,在息肉分割任务中去使用CNN和Transformer结合的方法越来越流行了。ColonFormer [9] 包含一个Transformer编码器和一个CNN解码器,用于高效准确的息肉分割。SwinE-Net [10] 结合了基于CNN的EffecentNet和基于vit的Swin Transformer [11] ,通过应用多重扩张卷积、多特征聚合和专注的反卷积来分割息肉。这些CNN-Transformer结构通常利用CNN特征的详细高分辨率空间信息和Transformer的全局上下文来提高息肉分割性能。
基于上述讨论,本文提出了一种基于CNN和Swin Transformer的双分支特征提取的医学图像分割网络(Dual branch feature extraction network based on CNN and Swin Transformer, DST-Net)。与几种最先进的网络相比,DST-Net的主要创新内容如下:
1) DST-Net包含一个双分支特征提取路径编码器,其中一个是VGG编码器分支,另一个是AC (Atrous convolution,空洞卷积) [12] 编码器分支。该双分支编码器可以同时提取局部边界特征和上下文多尺度特征,提高了网络在息肉分割中的特征表达能力。
2) 在编码器和解码器中间的底部模块融入两个连续的Swin Transformer模块,充分利用Transformer的远程依赖关系进一步加强网络的全局特征提取能力。
3) 在第一个U型的跳跃连接引用通道注意力模块,能精确定位息肉区域并抑制息肉背景的干扰,提高分割的精度。
在2个标准的息肉分割基准数据集(CVC-Clinic DB, Kvasir)上进行了实验,并比较了DST-Net与其它息肉分割方法的有效性。大量的实验表明,该网络能对息肉进行精准分割。
2. 方法
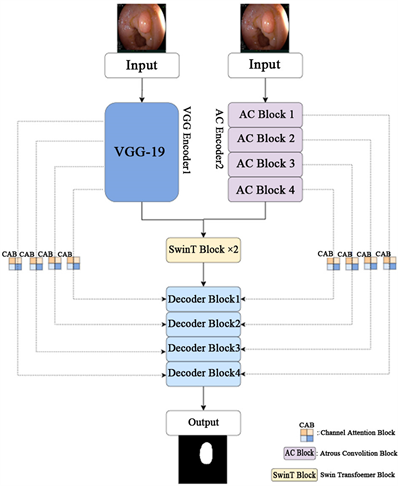
Figure 1. DST-Net network structure
图1. DST-Net网络结构
2.1. 网络架构框架
如图1所示,整个框架分为使用基于VGG和空洞卷积的双分支特征提取路径编码器、Swin Transformer底部模块和解码器。其中VGG编码器分支包含5个编码器块,使用了预训练的VGG-19作为骨干网络,经过5次下采样后,最后将原图缩小了32倍。AC编码器分支包含5个自定义的编码器块,每个编码器块都是一个相同的残差结构,包括1个空洞卷积模块加BN和激活函数,和一个2 × 2的池化层。Swin Transformer底部模块包含两个连续的Swin Transformer模块。解码器包含五个解码器子块,由一个双线性插值上采样层和两个3 × 3的卷积层加BN组成。上采样的特征通道数为256,128,64,32,16。在五次上采样中,通过跳跃连接将双分支编码器不同层输出的低级语义特征使用通道注意力模块重塑后与上一层解码器子模块得到的高级语义特征拼接。
2.2. 空洞卷积模块(AC Block)
如图2所示,空洞卷积模块对输入特征分别经过扩张率分别为1,6,12,18的3 × 3空洞卷积、BN和激活函数。然后对这四类特征进行拼接,再经过1 × 1的卷积、BN和激活函数处理。空洞卷积模块通过增强不同的感受野,捕捉图像的上下文信息,让提取到的特征具备上下文多尺度的特性。
其主要公式如下所示:
(1)
(2)
(3)
(4)
(5)
上列公式中,
代表输入特征,
代表
的普通卷积,BN代表Batch Normalization,
代表ReLu函数,
代表
4个空洞卷积分支的输出,
代表
的
空洞卷积,
代表堆叠,
代表哈达玛积,
代表最终输出。
2.3. Swin Transformer底部模块
如图3所示,Swin Transformer底部模块位于编码器和解码器中间,将双分支特征提取路径编码器的输出特征经过两个连续的Swin Transformer模块,利用Swin Transformer良好的全局上下文建模能力,能够有效的学习特征的远程依赖性,进一步增强了网络提取全局特征的能力。Swin Transformer 模块包括两个归一化层、一个窗口多头注意力模块和一个MLP层组成。Swin Transformer block是成对使用的,其中第一个block的多头注意力模块先使用一个Window MSA (W-MSA)结构,第二个block使用Shifted Window MSA (SW-MSA)结构。两个连续的Swin Transformer模块流程公式可表述为:
(6)
(7)
(8)
(9)
上列公式中,
表示来自L层的Swin Transformer模块输入,
代表L + 1层的Swin Transformer模块的输出,
和
分别代表L层和L + 1层的多头注意力的输出与输入的相加。
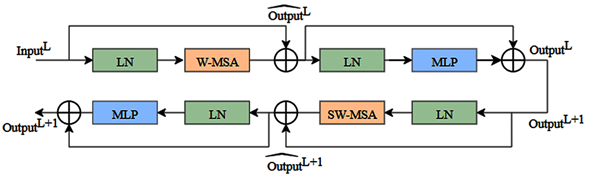
Figure 3. Two consecutive Swin Transformer modules
图3. 两个连续的Swin Transformer模块
2.4. 通道注意力模块 (CAB)
如图4所示,CAB对输入特征分别在通道域进行全局最大池化和全局平均池化。然后对这两类特征进行两个全连接层的处理,得到两个值域分别为0和1的一维向量。将两个向量连接起来并与原始输入特征相乘,最后再通过Dense层,能够抑制背景的干扰,增强感兴趣区域的特征响应。其主要公式如下所示。
(10)
(11)
(12)
上列公式中,
代表原始输入,
代表全局最大池化分支的输出,
代表全局平均池化分支的输出,
代表全局最大池化函数,
代表全局平均池化函数Dense代表全连接层,Concate代表堆叠,
代表哈达玛积,
代表最终输出。
3. 实验
3.1. 数据集和对比模型
基于两个具有挑战性的公开息肉数据集进行评估,即CVC-Clinic DB [13] ,Kvasir [14] 来验证提出模型的有效性。具体如下:
CVC-ClinicDB数据集通过31个结肠镜检查视频中收集了612张息肉图像。
Kvasir数据集通过内窥镜设备收集了1000张息肉图像。
为了评估网络的性能,将DST-Net与五个开源模型的分割结果进行对比,对比模型包括U-Net、ResU-Net [15] 、DoubleU-Net、PraNet [16] 、SwinU-Net。为了公平的比较,所有的网络在相同的训练、验证和测试集上进行评估。
3.2. 实验设置
本文的实验基于Ubuntu 18.04 LTS 64位操作系统中的python 3.7.0和Tensorflow + Keras版本2.5.0环境。在训练过程中,优化器使用Nadam,学习率 的初始值
= 1e-5,损失函数使用二进制交叉熵。Batch size设置为4,迭代次数设置为200。同时利用相应的回调函数,监控验证集损失,当损失50轮无法迭代时,停止迭代。实验借鉴了PraNet的训练设置,将CVC-ClinicDB和Kvasir的图像按照8:1:1随机分成训练集、验证集和测试集。所有方法都在带有NVIDIA GTX3080Ti (8GB)的服务器上进行。
3.3. 对比试验
本文使用ClinicDB和Kvasir-SEG数据集来评估所提出模型的学习能力。首先对训练数据集进行数据增强,通过水平翻转、垂直旋转、中心裁剪、弹性变换、高斯噪声和通道转置等方式对图像进行12次扩充。最终将训练数据扩充到15660张图片。从表1的结果可知,在ClinicDB数据集上,DST-Net网络在Dice、mIoU和Precision三项指标上均获得了最优的结果,分别达到了0.929、0.879、0.971。其中在Dice和mIoU的评分上比次优模型DoubleU-Net高3.0%和2.5%,比PraNet高3.1%和3.0%。在Recall指标上DST-Net网络取得0.840,只比最高的DoubleU-Net低0.3%,高于PraNet2.8%。在Kvasir数据集上,DST-Net网络在Dice、mIoU、Recall和Precision四项指标上均获得了最优的结果,分别达到了0.902、0.831、0.825和0.953。其中Dic和mIoU的评分上比次优模型PraNet高0.5%和0.01%,比DoubleU-Net高0.07%和1.4%。从对比实验结果可知,提出的模型优于现有的方法,证明了它通过统筹卷积和Transformer在局部和全局特征提取中各自的优势拥有了更好的学习能力和分割性能。图5给出在这两个数据集上DST-Net模型与比较模型的可视化结果。从图5中可知,DST-Net的分割结果和PraNet、DoubleU-Net和SwinU-Net对比是最接近真实标签的。DST-Net解决了SwinU-Net在局部边界特征提取能力上的不足,从图中可以明显看出DST-Net在边界细节上的处理更好。DST-Net解决了PraNet和DoubleU-Net在全局上下文特征提取能力上的不足,DST-Net对背景干扰的处理更好。DST-Net在面对内窥镜采集和结肠镜检查视频这两种不同的采集方式依然能保持稳定的分割能力。

Table 1. Comparative experimental results
表1. 对比实验结果

Figure 5. Comparison of experimental visualization
图5. 对比试验可视化
3.4. 消融实验
为了证明该网络的有效性,进一步对DST-Net进行了消融实验。消融实验通过逐渐去除与DST-Net网络相关的组件来进行。
验证通道注意力模块的有效性。在DST-Net中去除通道注意力模块形成DST-CAB网络。如表2所示,DST-CAB的Dice、mIoU、Recall和Precision四项指标在两个数据集上分别达到了0.921、0.867、0.837、0.964和0.896、0.817、0.820、0.949。其中Dice得分分别低于DST-Net约0.8%和0.6%,mIoU约1.2%和1.4%,通过对比验证了通道注意力模块的有效性。
验证空洞卷积模块的有效性。在DST-CA中去除空洞卷积模块形成DST-CAB-ACB网络。如表2所示,DST-CAB-ACB的Dice、mIoU、Recall和Precision四项指标在两个数据集上分别达到了0.913、0.857、0.815、0.958和0.887、0.804、0.814、0.931。其中Dice得分分别低于DST-CA约0.8%和0.9%,mIoU约1.0%和1.3%,通过对比验证了空洞卷积模块的有效性。
验证Swin Transformer 底部模块的有效性。在DST-CAB-ACB中去除Swin Transformer 底部模块形成DST-CAB-ACB-ST网络。如表2所示,DST-CAB-ACB-ST的Dice、mIoU、Recall和Precision四项指标在两个数据集上分别达到了0.906、0.831、0.816、0.953和0.881、0.802、0.801、0.930。其中Dice得分分别低于DST-CA-PIFM约0.7%和0.6%,mIoU约2.6%和0.2%,通过对比验证了Swin Transformer 底部模块的有效性。
此外,通过图6消融实验可视化可以观察到DST-Net的预测结果是最接近真实标签的。DST-CAB网络和DST-CAB-ACB网络的预测结果在边界的处理上稍差一点。DST-CAB-ACB-ST网络的预测结果是图中最差的。
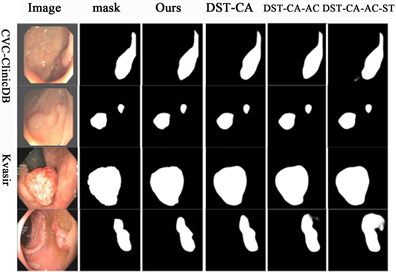
Figure 6. Ablation of experimental visualization
图6. 消融试验可视化
4. 总结
本文提出了一种基于CNN和Swin Transformer的双分支特征提取的医学息肉分割网络(DST-Net),通过统筹卷积神经网络和Transformer在提取局部特征和全局特征各自的优势,能够有效提高息肉分割的精度。我们设计了结合VGG和空洞卷积(AC)的双分支编码器,在VGG通过卷积运算提取局部细节特征的基础上,利用空洞卷积更大的感受野去补充特征的上下文多尺度信息,以此增强网络对息肉特征的表达能力。我们还设计了Swin Transformer 底部模块,通过Transformer的远程依赖关系进一步增强网络的全局语义特征特征提取能力。我们在跳过连接使用了通道注意力模块(CAB),能精确定位息肉区域并抑制息肉背景的干扰,提高分割的精度。本文提出的DST-Net在在CVC-Clinic DB和Kvasir两个公开的息肉数据集上都达到了最先进的性能,充分展示了强大的学习能力和出色的泛化能力。对预测结果的定性和定性分析表明,DST-Net可以促进计算机辅助诊断和治疗的发展。在未来的工作中,将考虑并推动轻量化模型进行更多的病灶分割。
基金项目
国家自然科学基金(61801288)。
NOTES
*通讯作者。