1. 引言
肾脏疾病及其并发症是人类的医学难题,严重威胁着人类的健康。小儿肾积水又是肾脏疾病中的常见病。而利尿肾图又是评价小儿肾脏功能的重要工具 [1] 。利尿肾图在评价分肾功能的同时,还可以协助临床鉴别机械性尿路梗阻和非机械性尿路扩张。目前已有多种影像学检查方法已用于儿童肾积水的评估,包括超声、静脉肾盂造影、CT尿路成像等 [2] 。而对于肾积水评估的关键在于核医学科医生对于肾实质的人工标注。这是一个耗时又费力,且不可重复的过程,所以人工标注的准确性在很大程序上也会取决于医生的经验和理解。
在过去的几十年里,许多学者对深度学习图像分割算法进行了研究 [3] 。深度学习由于其强大的非线性特征提取能力,在图像分类、目标检测、语义分割等方面取得了巨大成功。其中,卷积神经网络(CNN)就是最经典的算法,改进的CNN可以在医学图像数据集训练样本不足的情况下,完成精确的自动分割任务 [4] [5] 。例如,以全卷积网络(Full Convolutional Network, FCN) [6] 或Unet [7] 为代表的一系列模型被提出并广泛应用于医学,利尿肾图也不例外。Ronneberger等 [7] 借鉴了FCN的结构,提出了对称编解码结构的Unet,通过网络间的跳过连接,在生物医学图像分割中表现出优越性。
尽管CNN具有出色的表征能力,但是卷积运算中,由于U形结构感受野过小,导致在建模过程中,捕捉远距离关系方面受限。基于CNN的方法通常性能较弱,特别是对于处理纹理和形状方面差异较大的目标。为了克服这种局限性,人们提出了基于CNN的自注意力机制 [8] 。另一方面,Transformer在计算机视觉领域也得到了广泛研究,它完全不使用卷积算子,而仅仅依赖于注意力机制 [9] 。Vision Transformer (ViT) [10] 通过直接将具有全局自关注的Transformer应用于全尺寸图像,在ImageNet数据集上实现了最先进的分类。受ViT的启发,TransUNet [11] 进一步结合了Transformer和U-Net的优势,用于医学图像分割。然而,TransUNet的性能仍然受到医学图像数量的限制。而且,传统的Transformer还有一个典型的问题,就是它的计算复杂度。为了降低复杂度,Swin-Transformer [12] 使用本地窗口的自注意力来代替全局自注意力并划分移位的窗口,在各窗口之间的前一层建立连接。
为了解决这些问题,我们将所提出的算法应用于一个实际性的临床问题:在上海交通大学附属新华医院提供的利尿肾图数据集中进行肾脏自动分割。我们的工作贡献可以概括如下:
1) 提出了一种基于Swin-Transformer的编码器–解码器结构,并且优化了U型结构,在编码器每个阶段插入几个连续的不同膨胀速率的膨胀卷积,以获得足够的多尺度上下文聚合,该网络比单一网络能提取更多的全局特征。此外,利用特征增强模块通过扩展接受野来细化提取的深层特征,实现了对利尿肾动态显像中肾实质的分割。
2) 在跳跃连接中加入交叉注意力模块,它通过捕获多尺度编码器之间的通道和空间依赖关系来解决编码器和解码器之间的语义差距。
3) 设计了一个深度注意模块来融合提取的高级特征,该模块融合编码器的最后一个特征提取层和来自特征融合模块的特征,强制网络关注感兴趣区域。
2. 相关工作
2.1. 传统机器学习方法
传统方法在深度学习发展之前,许多传统的基于机器学习的方法已经应用于医学图像处理。Halkar等人 [13] 提出了一种单阈值方法,可以根据利尿肾造影自动绘制肾脏轮廓。但是肾造影的对比度相对较低,很难选择合适的阈值来有效区分前景和背景。Tomaru等人 [14] 提出了一种改进的双阈值半自动分割方法,但它仍需要手动操作指定两个肾脏的中心位置。Inoue等人 [15] 提出了另一种半自动分割方法,该方法将预设的矩形框手动移动到不同的ROI,利用每个部位不同的放射性峰值时间来减少非肾区域的干扰。这降低了对操作员的要求,但仍占用了大部分时间。Tian等人 [16] 提出了一种新的半自动分割方法,减少了人工参与。然而,当肾功能严重受损时,很难定位肾脏区域。这些半自动方法或多或少需要人类的参与,并且在一定程度上是主观的。同时,这些方法完全依赖于图像,并且算法的鲁棒性较差 [17] 。
2.2. 深度学习方法
Xie等人提出了SERUnet [18] ,这是一种用于肾脏和肿瘤分割的级联SE-ResNeXTUnet。首先,进行粗分割,在CT图像中找到肾脏的关键切片。然后将得到的patch作为输入,对分割进行细化。Wu等 [19] 使用级联的全卷积DenseNet对超声图像进行肾脏自动分割。通过预处理可以避免噪声和冗余信息的干扰。Chen等人 [20] 提出了DeepLabv3+,通过增加一个简单而有效的解码器模块来扩展DeepLabV3,以提高分割性能。Hsiao等人 [21] 提出了一种用于肾脏分割的编码器–解码器架构。实现了超参数优化过程,它允许计算总肾体积来估计ADPKD的肾功能。Azad等人 [22] 对DeepLabv3+模型进行建模,并通过使用Swin-Transformer作为架构的基本组件来建立TransDeepLab。该模型在各种医学图像分割任务中展示了有效建立远程依赖关系的潜力。
受先前工作的启发,提出了一种基于Swin-Transformer和注意力机制结合的编码器-解码器框架。它继承了Swin-Transformer的优点,有效提取全局和局部信息,对肾实质进行有效的分割。并且所提模型对架构进行了优化,在跳跃连接中引入了深度注意力模块,以有效进行信息交互。该方法通过对原始输入图像中的肾实质ROI区域进行分割,我们的模型成功地生成了肾ROI区域,实现了有效的分割。
3. 网络框架
3.1. 网络主体结构
基于利尿剂肾动态成像数据集,我们提出了一种基于Swin-Transformer的肾图自动分割网络。我们提出的网络结构是基于U型编码器–解码器网络构建的,其总体框架如图1所示。与常规U型结构网络不同,我们的网络结合改进了编码器、解码器和跳转连接,优化了U型结构。给定医学图像
的输入,其中,
表示图像的空间分辨率。图像先被输入Swin-Transformer模块,用于捕捉全局信息并构建分层特征表示,而后我们在每个阶段插入几个连续的不同扩展速率的扩展卷积,以获得足够的多尺度上下文聚合。在最后一层下采样中引入特征增强模块和深度注意模块,可以弥补空间表征并产生多尺度表征,并且通过深度监督的方式来获得具有判别性的高级特征。最后,通过Swin-Transformer解码器可以获得
的分割图。
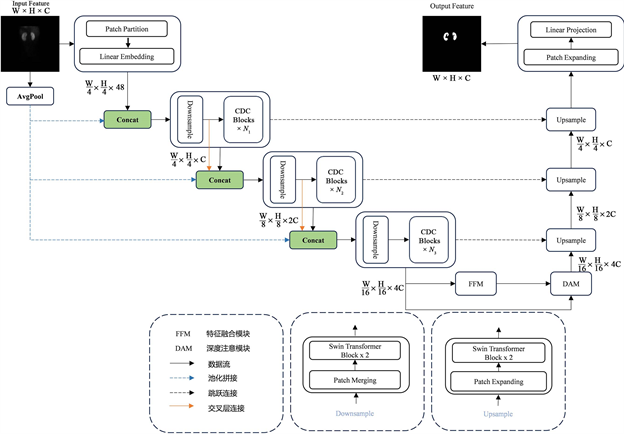
Figure 1. General framework of the proposed model
图1. 所提出模型的总体框架
3.2. Swin-Transformer模块
与传统的多头自聚焦(MSA)区块不同,Swin-Transformer区块 [12] 是基于移位窗口构建的。图2给出了两个连续的Swin-Transformer块。每个Swin-Transformer块由一个LayerNorm (LN)层、一个多头自聚焦模块、一个残差连接和一个具有GELU非线性的2层MLP组成。分别使用了基于窗口的多头自关注(W-MSA)模块和基于移位窗口的多头自关注(SW-MSA)模块。基于这种窗口划分机制,连续旋转变压器块可表示为:
(1)
(2)
(3)
(4)
其中
和
分别表示第
个块的(S)W-MSA模块和MLP模块的输出。根据先前的研究 [23] [24] ,自我注意力的计算公式如下:
(5)
其中
表示查询矩阵、密钥矩阵和值矩阵。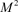 表示一个窗口内的面片数量。
是查询或键的大小。
表示相对位置偏移,B中的值取自偏移矩
。
表示一个窗口内的面片数量。
是查询或键的大小。
表示相对位置偏移,B中的值取自偏移矩
。
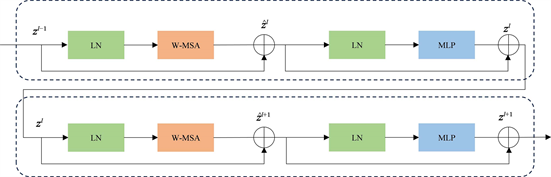
Figure 2. Swin Transformer block
图2. Swin Transformer模块
3.3. 连续膨胀卷积模块
如图3所示,本文提出的连续膨胀卷积模块(CDC)利用扩张卷积来提取多尺度局部特征。与仅在网络的最后一层使用并行不同,我们在网络的最后一层插入了多个连续的扩张卷积模块。不同的是,我们在每个阶段都插入了几个连续的稀释卷积,以实现充分的多尺度上下文聚合。给定一个二维信号
,二维稀释卷积的输出
可以定义为二维扩张卷积的输出可定义为:
(6)
其中,
为长度为
的滤波器,
为用于卷积输入
的膨胀率。在标准非展开卷积中
。通过使用扩展卷积,网络可以保持输出特征映射的大小固定,同时获得更大的感受野。考虑一个尺寸为
的输入特征
,CDC模块输出
为:
(7)
其中,
表示逐点卷积操作,然后是GELU [25] 激活。BN为批归一化层,
为
深度膨胀卷积,扩张速率为
。
3.4. 特征增强模块
如图4所示,在医学图像分割领域,特征表示的准确性直接影响到网络的分割和识别效果。为此,我们引入特征增强模块,旨在通过吸收补充信息来增强提取的特征,从而增强网络的判别能力。特征增强模块在不增加参数数量的情况下,使用了一个具有不同窗口大小(窗口大小 = 4,6,8,12)的Swin空间金字塔池化模块,以捕捉多尺度表示。在我们的设计中,较小的窗口尺寸旨在捕捉局部信息,而较大的窗口尺寸则用于提取全局信息。然后,生成的多尺度表征被输入到跨语境注意力模块,以非线性技术融合并捕捉通用表征。
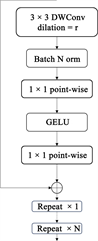
Figure 3. Continuous expansion convolution module
图3. 连续膨胀卷积模块
在Swin空间金字塔池化之后,由通道注意机制生成通道注意图(Channel Attention Map, CAM)。该CAM基于通道间的关系,使网络能够识别和优先考虑显著的特征通道,从而在医学图像中聚焦相关结构,同时减轻无关的背景噪声。连接的特征集通过
卷积和批处理归一化层进一步细化。通过
卷积处理与原始输入特征的加性融合来补充这一点,确保原始特征完整性与增强特征集的保留和整合。最后,特征集输入到空间注意机制(Spatial Attention Module, SAM),可自适应地重新校准空间权重分布。这种机制在突出重要的空间区域和减少非必要背景元素的影响方面至关重要。通过进一步改进了特征表示,提高了网络在医学图像分割任务中的性能和准确性。
3.5. 深度注意模块
如图5所示为深度注意模块的结构。该模块以原始模型中特征提取的最后一层和特征增强模块的特征为输入。这些特征是在点向卷积后添加的,其中通道减少率设置为4。经过ReLU、点向卷积和Sigmoid,我们得到了与原始输入特征大小相同的注意图。最后,将提取的注意力与来自特征增强模块的特征相乘,得到最终的注意力图。
对于利尿肾图,存在较多的干扰,目标与背景之间的对比度很低。因此,为了融合多尺度的特征并迫使网络专注于突出的目标区域,受 [8] 的启发,我们使用了一种新的策略,称为深度注意模块。深度注意模块将下采样的最后一层输出的特征和特征增强模块输出的特征为输入,输出的融合注意图
,我们将下采样的最后一层和特征增强模块输出的特征分别定义为
和
。为了减少网络模型的参数,我们选择了点向卷积来提取特征,用PConv表示。因此,深度注意模块可以表示为:
(8)
(9)
其中
表示获得的注意图。
表示ReLU激活函数,
表示Sigmoid激活函数。
表示最终的注意特征图,然后是全局自适应平均池化层和完全连接层,输出特定疾病的预测概率。因此,我们可以利用深度关注模块,通过深度监督的方式来获得具有判别性的高级特征。
4. 实验
4.1. 实验设置
我们提出的模型使用我们精心收集的肾脏动态成像数据集进行了评估。训练和测试的仿真在配备24 GB内存和英伟达RTX 3090的强大GPU上使用PyTorch执行。在仿真训练过程中,我们配置了特定的模型参数:输入图像大小为384 × 384,仿真训练过程跨度为150个epoch,批量大小为4,以提高学习效率。为了优化网络梯度和参数,我们采用了Adam优化算法,初始学习率为0.0001。
4.2. 数据集
本研究使用的数据集包括从上海交通大学医学院附属新华医院获得的利尿肾图。数据集是在患者的临床诊断过程中使用飞利浦医疗公司的SPECT设备采集的。新华医院批准使用这些数据,其中包括980个儿科患者的动态图像序列。原始数据的分辨率为128 × 128像素,以DICOM格式保存。每次诊断持续20分钟,共生成136幅原始图像。在第一分钟内,共采集了60幅原始图像,每幅图像以每秒一帧的频率拍摄;在随后的19分钟内,共采集了76幅原始图像,每幅图像以每15秒一帧的频率拍摄。根据核医学医生准确诊断肾功能的要求,我们选择了前2分钟的原始图像进行肾脏分割,特别是前64帧。选定的原始图像经过合成和增强预处理后,形成了分辨率为384 × 384像素的980幅图像数据集。图6显示了原始肾脏标签,黑色代表肾脏外部,白色代表肾实质。数据集被随机分成三个子集:784张图像分配到训练集,98张图像分配到验证集,98张图像分配到测试集。
所有原始图像的标注过程都是在一名经验丰富的核医学医生的指导下进行的。对利尿剂肾图中实质区域的识别经过了多次研究,并与核医学医生进行了讨论。医生进行了多次标记尝试并进行了彻底审查,反复修改,以确定肾脏标记的精确准则。最终,另一名核医学医生确认了所有图像标注,以确保在利尿剂肾图中准确标注肾实质。
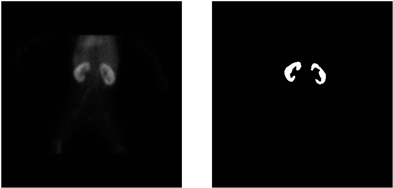
Figure 6. Diuretic renogram (raw data on the left, labels on the right)
图6. 利尿肾图(左为原始数据,右为标签)
4.3. 评价指标
分割结果使用四个指标进行评估:DSC、IOU、精确度和召回率。骰子相似系数(DSC)是一个区域级指标,用于衡量预测结果与地面实况之间的重叠率。它对掩码的内部填充特别敏感。图像分割可以作为单个像素的二元分类任务来处理。通过定量分析真阳性(TP)、真阴性(TN)、假阳性(FP)和假阴性(FN),我们可以有效地评估分割的性能。这些指标定义如下:
(10)
(11)
(12)
(13)
4.4. 实验结果
在本节中,我们通过与最先进的利尿肾图分割方法进行对比分析,评估所提出的模型的功效。所有模型都是在与模型相同的参数和仿真环境条件下进行评估的。表1中的性能评估使用四个指标来评估效果,展示了比较结果。在所有评估指标上,我们的方法始终优于其他方法,显示出卓越的效果。
表1全面比较了用于利尿肾图分割的不同分割方法。仿真实验结果清楚地表明,所提出的方法在各种评价指标上始终优于其他模型,展示了其卓越的功效。具体来说,该方法的最佳Dice和IoU分数分别为91.53%和83.29%,比Unet有显著提高,Dice和IoU分别提高了2.7%和1.95%。此外,本方法的Dice和IoU分别比TransUnet提高了0.92%和0.95%,Recall也提高了1.4%。与Swin-Unet相比,本方法的Dice和IoU分别提高了1.19%和1.1%。此外,在所有模型中,本方法的仿真得分指标均为最高,这些结果凸显了本方法与其他模型相比的优越性。

Table 1. Quantitative results on the diuretic renogram dataset
表1. 利尿肾图数据集上的定量结果
图7直观展示了各种方法的分割结果,提供了相应结果的定性可视化。前景中的白色区域表示需要分离的独特肾实质。我们选择了四个具有代表性的案例来研究不同网络的性能差异。在案例1中,虽然在检测过程中肾脏以外的部分器官可能会出现在肾脏动态可视化中,但由于肾功能更加明显,白色区域得以突出显示,因此最终预测结果更好。然而,在肾功能受损的案例2、3、4中,利尿剂肾图中肾实质的亮度明显降低。这给一些网络准确区分所有肾脏区域带来了挑战,导致预测不完整,并容易受到邻近器官的干扰。因此,这导致了不理想的分割结果。与其他方法相比,本文所提出方法的分割性能更好,这主要体现在来自非肾脏区域的干扰较少,网络更专注于对肾实质的学习。对于肾脏内差异不明显的区域识别准确率更高。总体而言,这些可视化结果验证了我们提出的方法的有效性。

Figure 7. Visualisation of quantitative results from different methods
图7. 不同方法的定量结果可视化
5. 结论
本文介绍了一种旨在自动进行肾脏分割的创新算法。我们的分割模型结合了Swin-Transformer、深度注意模块和特征增强模块的优势。通过利用Swin-Transformer自我关注机制的先进远程上下文信息建模功能,显著提高了不同医学图像中语义分割的准确性。特征增强模块在连接空间表征和通过空间金字塔池生成多尺度表征方面发挥了关键作用。此外,连续扩张卷积能通过获取多尺度上下文聚合来扩大感受野。在我们的仿真实验中,所提出模型在自动分割利尿肾图中的表现优于许多现有的医学图像分割模型。总之,我们的模型通过提取深层语义特征来自动识别和分割肾实质,在准确性上超越了现有模型。但是,复杂的网络势必会增加更多的训练参数,训练和推理的速度也会增加,这将在下一步的工作中进行改进。
基金项目
国家自然科学青年基金(61703277)。