1. 引言
胎盘是维持胎儿在宫腔内正常发育的器官,是沟通母亲与胎儿的桥梁,在孕期中起到了气体交换、营养交换、废物代谢等作用。同时胎盘对于细菌和较大的病原体也有一定的防御作用。因此,胎盘的健康与否严重影响着胎儿的生命健康以及孕妇安全。目前临床上有一些由于胎盘异常导致的疾病,例如胎盘植入,前置性胎盘,双胞胎输血综合征,宫内生长受限等。所以直观的展现出胎盘的结构有助于医生诊断效率的提高。
由于MRI图像的特性,腹部器官的灰度值都很多近似的地方,传统的分割方法难以有效的分割出目标区域,使用深度学习进行胎盘分割可以有效地解决这些问题,例如Gu Ran等人 [1] 提出了一种基于综合关注的卷积神经网络CA-Net,以实现更准确和可解释的医学图像分割用于胎盘的MRI分割。Wang等人 [2] 提出了一个改进的U-Net框架:RU-Net。在原有的U-Net收缩路径和扩展路径上加入了ResNet的直接映射结构来对胎盘分割以助力侵袭性胎盘增生和粘稠性胎盘的相关诊断。Bella等人 [3] 针对胎儿MRI扫描中胎盘的定量体积评估,提出了一种基于深度学习的自适应胎盘自动分割方法。
在二维情况下,虽然可以通过每个切面的状况对胎盘状况进行判断,但为了能够更好的满足临床的实际需求,例如测量胎盘体积等,需要对胎盘进行三维的可视化。曹艳文 [4] 等人利用Mimics软件对孕晚期的胎盘核磁共振成像数据进行数字化三维重建。Barrena等人 [5] 在其分割胎盘分割结果上使用了Marching cubes创建了胎盘及其周围血管的3D模型,用于识别胎儿疾病(如胎盘或前置血管)中与胎盘及其血管相关的严重异常。
上述的研究内容是分别对胎盘分割和重建进行了研究,为了能够方便用户能够直观的看到分割与重建的相关结果,使相关的研究内容能够更好用于实际,本文利用PYQT技术将分割与重建的算法进行了集成并加入交互功能,搭建了一个胎盘分割与三维重建系统。
2. 图像预处理以及胎盘分割
2.1. MRI图像预处理
MRI数据的一般存储格式为DICOM [6] ,而一般情况下,图像处理算法的输入为JPG、BMP等图片格式。故将MRI数据转换为512 × 512的.jpg格式的RGB图像。在RGB胎盘图像中,胎盘与周围区域对比度很低,胎盘像素与周围组织像素位于非常接近的灰度范围内,这不利于分割任务的进行。故考虑使用直方图均衡化(AHE)来提高图像对比度,使得胎盘与周围组织结构的边界更加清晰。
直方图均衡化主要是基于概率密度函数(probability density function, PDF)的均扩展来重新映射输入图像中每个像素的灰度级。首先,它从原始直方图构造一个累积直方图。其次,将此累积直方图归一化为输出图像的强度值。然后,将归一化的累积直方图作为到原始图像的传递映射函数。其转换公式主要由映射区间和映射函数即累积分布函数(cumulative distribution function, CDF)构成 [7] 。
上述所说的概率密度函数和累积分布函数可以用式(1)、(2)表达:
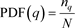 (1)
(1)
(2)
其中q表示直方图上的某一灰度级,nq表示q灰度级上统计像素的个数,表示输入图像的总像素量,start表示某区间的最小灰度级。
根据式(2)可得出直方图均衡化的转换公式为:
(3)
其中end-start表示映射区间。在传统的直方图均衡化转换公式中end为255,start为0。
但是直方图均衡化(AHE)会导致以下问题:
1) 直方图均衡是全局的,对图像局部区域存在过亮或者过暗时,效果不是很好;
2) 直方图均衡会增强背景噪声。
考虑到以上两点原因,本文考虑利用CLAHE(限制对比度自适应直方图均衡化)算法 [8] 提高了图像的对比度。该算法通过对图像的局部区域进行直方图均衡化,这样使算法能够更多的关注局部信息,防止全局信息的影响,同时通过限制对比度的方法,减少了背景噪声的干扰。图1展示了使用普通直方图均衡化算法以及CLAHE算法得出的不同结果,可以看出,CLAHE算法增强的效果更优。

Figure 1. Image enhancement processing result chart
图1. 图像增强处理结果图
2.2. 胎盘分割
2.2.1. 分割模型
本文的目的使要对胎盘进行三维可视化,而可视化的基础是对胎盘区域进行准确的分割,在医疗图像处理领域,U-Net网络起到了非常重要的作用。该网络在2015年由Ronneberger等人 [9] 提出,该网络在全卷积神经网络(FCN)的基础上,采用了编码器–解码器的结构。在编码器部分,每一个下采样编码模块都是有两个3 × 3的卷积层和一个2 × 2的最大池化层组成。解码器部分,每个上采样解码模块先于对应的解码模块输出的特征进行拼接,然后在经过两个3 × 3的卷积层构成。同时,在U-Net网络中还加入了跳跃连接。具体的网络模型如图2所示。
由于胎盘的位置大小会存在着较大的变化,原始的U-Net网络对于这种变化的适应性并不强,因此本文在原始U-Net网络的基础上改进了跳跃链接,通过特征链接模块(FLM)将相邻两个编码器模块的特征输出进行整合用于跳跃链接,使得传输的信息更加的充分,使网络能够更好的利用上下文的语义信息。同时还在在编码器与解码器中间加入了金字塔池化模块,通过金字塔池化,使网络对不同大小的胎盘特征都有足够的感知能力。具体网络如图3所示。
2.2.2. 实验设置
本文提出的胎盘分割网络通过使用pytorch框架进行网络的结构的搭建,在实验过程中,本文的相关参数设置如下:总训练轮次epochs设置为200,初始学习率设置为0.0001,训练批次大小Batch Size设置为16,使用Adam优化器进行优化。在损失函数的选择上,本文使用了交叉熵损失函数与Dice损失函数混合计算。经过相关的实验,提高网络的性能,我们将整体损失函数定义如下:
(4)
其中
为两个损失之间的比例参数。经过实验,本文最终将
设置为0.6。
为了验证本文方法的有效性,量化实验指标,本文使用了(Dice),并集(MIOU),准确率(ACC)以及豪斯多夫距离(Hd)作为评价指标,计算公式如下所示:
(5)
(6)
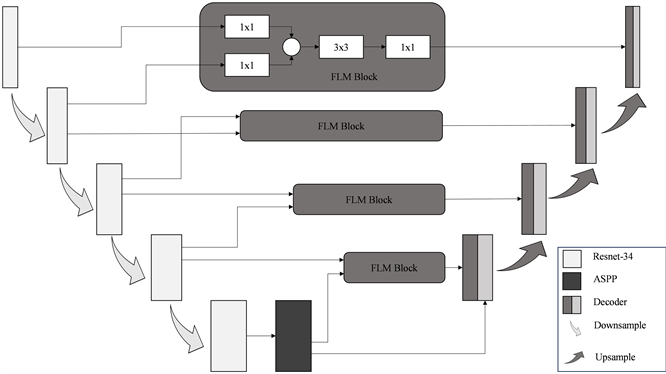
Figure 3. The network model in this paper
图3. 本文网络模型
(7)
其中,TP是预测目标区域与真实目标区域重叠的体素数量,FP是过分分割出目标区域的体素数量,FN是未检测到的目标区域体素数量,TN是非目标区域与测正确的体素数量。
(8)
其中,
分别为预测胎盘边界和真实胎盘边界,
表示边界之间的距离。
2.2.3. 实验结果
本文在一个包含400张包含胎盘的MRI数据集中进行了训练,测试结果如图所示,由图4可知,我们提出的方法可以对胎盘进行有效的分割,且与其他方法比较下也有更好的分割能力。同时本文使用了上文所述的指标对实验结果进行了量化,量化结果如表1所示,由表1可知本文的方法获得了0.9089的Dice值,5.0032的Hausdorff值,0.8305的Iou值和0.9940的ACC值,优于其他实验方法。
3. 胎盘三维重建
3.1. 三维重建方法
本文使用VTK工具包,利用分割完成的数据分别使用了面绘制中的移动立方体算法和体绘制中的光线投射算法对胎盘进行了三维重建。
在三维重建中,体绘制算法是通过将三维的数据中的每一个体素设置透明度后投影到二维中进行物体的可视化,这种算法的优点就是可以更好地展现重建体内部的细节,例如在医学上存在肿瘤侵入的位置使用体绘制的重建方法就可以很直观的看到侵入的程度。但是由于需要针对整个三维的数据进行处理,所以体绘制重建方法的实时性不强。

Figure 4. Comparison of segmentation results between the model in this paper and other models
图4. 本文模型与其他模型分割结果对比

Table 1. Comparison table of segmentation results between the model in this paper and other models
表1. 本文模型本文模型与其他模型分割结果对比表
体绘制的算法主要有以下几种:光线投影算法(Ray Casting)、错切–变形算法(Shear-wrap)、频域体绘制算法(Frequency Domain)和抛雪球算法(Splatting)。其中光线投影法较为常用,由于光线投射法的重建精度较高,且容易移植到GPU来加速重建速度,所以下面将对光线投影法进行详细的描述并使用该算法进行胎盘三维重建。
光线投影法的原理如图5所示,从视点所发出的光线会穿过整个物体的三维数据场,在穿过的物体内部均匀的进行数据的采样,针对每个采样点,采用插值技术以计算其体素值。通过应用颜色传输函数和不透明度传输函数,获取相应的颜色值和不透明度信息。随后,借助光线吸收模型,对颜色值进行累加,直至光线穿过三维体数据。这样,就可以得到当前平面像素的渲染颜色,最终生成显示图像。

Figure 5. Schematic diagram of the light projection method
图5. 光线投射法原理图
不同于体绘制可以很好反应重建物体内部的细节,面绘制的优点在于不需要把整个体数据用于计算,只需要获取重建体的等值面,利用等值面就可以重建出物体的三维形状,在重建的效率上优于体绘制算法。在面绘制算法中最常用的算法是移动立方体算法。移动立方体算法的基本思想是通过将由多个切面组成的数据看成三维数据,通过多个切面的提取的等值面,进而构建三维体的表面网格,最终构建整个三维模型。
在移动立方体算法中,定义由八个顺序排列的体素构成的方格叫做体元,根据体元中的顶点在体元中的位置划分。如图6所示,体元的状态可以被分为以下15种基本类型。而这15种基本类型一共可以衍生出256种类型状态,通过将这256种状态制作成查找表,然后对所有的体元进行遍历操作后,与查找表内的状态进行对应组合,即可构建三维模型的网格数据。
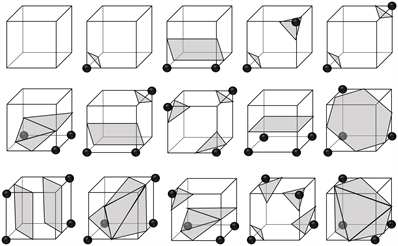
Figure 6. 15 basic positional divisions
图6. 15种基本位置划分
3.2. 三维重建实验及结果
本文通过使用VTK工具包完成对胎盘的重建程序的编写,在体绘制过程中主要使用了vtk GPU Volume Ray Cast Mapper函数来实现基于GPU加速的光线投射体绘制算法;同时使用vtk Piecewise Function函数来设置不透明度函数。在面绘制算法中,主要使用了vtk Marching Cubes函数来实现移动立方体算法。为了更好地展示重建结果,所使用的几种方法均进行了高斯平滑操作。
在分别使用3d-slicer,体绘制,面绘制对同一序列的胎盘核磁共振图像进行重建实验后,得到如下结果:
由图7可知,本文所使用的方法在胎盘三维重建的效果上与现有成熟软件所呈现的效果类似,甚至更好。而且通过结合深度学习技术对胎盘的自动分割,重建胎盘任务整体的效率得到了有效的提高。在胎盘的体绘制与面绘制的结果呈现中,虽然面绘制的效率会优于体绘制,但是重建的效果会有所欠缺,且考虑到后续的研究需要等,故本文最终选择面绘制作为胎盘三维重建的主要方法。
4. 胎盘分割与三维重建系统搭建
为了直观的显示胎盘分割与重建的结果,本文利用PYQT进行相关界面程序的开发,搭建了胎盘MRI分割与三维重建系统,如图8所示,系统整体分为两个部分:参数调节,结果展示。在参数调节部

Figure 8. Placenta MRI segmentation and 3D reconstruction system
图8. 胎盘MRI分割与三维重建系统
分主要有数据加载及预处理、图像分割以及三维重建三个部分组成。结果展示部分将会显示原始数据,预处理结果,语义分割结果以及三维重建结果。
如图9所示,该系统结合本文所使用的相关算法模块,在本文的实验中,所用到的原始数据均由原始DICOM格式的数据集导出的512 × 512大小的JPG格式的图片,图片依旧按照原始序列中的顺序进行排列,在导入原始数据后,首先将利用第二章所述的CLAHE算法进行图像增强处理,再利用训练完成的神经网络对图像进行分割,将分割完成的图片存储到指定的文件夹中,在选择插值方式以及重建算法后,系统会读取文件夹中分割完毕的图像用于胎盘的三维重建,上述的每一部分处理结果将会实时的展示在相应的图像显示区域。
5. 总结
在本文中,我们成功地应用深度学习技术,并结合VTK和PYQT等工具,开发了一套胎盘分割与三维重建系统。通过深度学习算法的应用,本文实现了对胎盘的高效自动分割,提升了核磁共振图像中胎盘区域分割的准确性和效率。同时,采用VTK和PYQT等工具赋予本文的系统直观的三维重建展示界面,为医生提供清晰全面的胎盘结构信息,同时提供了一定的交互能力便于展示不同算法得到的结果。在后续的研究中会更加注重功能的完善,使该系统不仅仅针对与胎盘这一个部位,包括对多个部位的分割,使其适用于更广泛的医学图像处理任务。
基金项目
上海市科委科技创新行动计划(21S31904200, 22S31903700);上海理工大学医工交叉项目(24USST-GD05)。