1. 引言
从1990年代起,循环神经网络(Recurrent Neural Network, RNN) [1] 在处理整数序列中展现出强大的分析和处理功能。通过大规格集成电路等相关计算工具的发展,目前已经有了长足的进步。近年来,针对不同应用,出现了各种基于RNN 的变体结构,并被应用在各个特殊的应用领域中 [2],循环神经网络已经成为在深度学习领域中一类非常重要的模型。
在现实生活中满足序列性质的数据是普遍存在的,这类数据也助推了RNN在实际应用中获得不断地拓展,例如:时间序列处理,股票行情预测 [3],金融数据预测 [4],雷达临近预报 [5];语音序列识别 [6] 等。对RNN的应用研究偏重于的是实际应用方面。本文将基因序列分类作为RNN 应用的代表,探究RNN分类器的稳定性问题,利用变值理论 [7] [8] [9] [10] 体系框架,达到展示RNN分类器所生成的分类序列的稳定性特征,从可视化的角度,展现其移位后不变的特性。
通过可视化方法将RNN稳定性抽象概念具体形象化,展示RNN分类器分类结果数据集中的数据分 布情况以达到对RNN 分类器稳定性探索与研究的目的。
2. 体系结构
本文将预分类的基因序列进行分段、移位。针对预分类后的序列操作,得到在不同移位长度值条件下的基因序列数据集合,将不同数据集分别作为RNN分类器的输入,得到对应的检测数据集。对不同检测数据集中的序列进行替换,得到最终可视化的序列数据集。然后通过统计计算模块操作提供可视化的模块展示。
文中使用的体系结构主要分为以下几个模块:预分类序列操作模块、RNN分类模块、可视化序列操作模块、统计计算模块、可视化模块。
该体系结构下,数据处理的流程如图1。
3. 核心处理模块
在文中所使用的框架下不同的处理模块会有不同的操作。
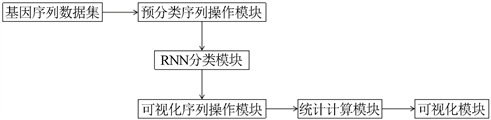
Figure 1. Flow Chart of Data processing
图1. 数据处理流程图
3.1. 预分类序列操作模块
该模块主要包括序列分段操作和序列移位操作,参阅“图2”。

Figure 2. Operation diagram of pre-classification sequence
图2. 预分类序列操作
首先是序列分段操作,将整个基因序列数据作为字符串,然后按照分段长度(dl)进行分段处理,将长度小于分段长度的序列舍去,使得基因序列字符串转变成为由固定分段长度的序列组成的数据集。
其次是序列移位操作,将序列数据集中的单条序列字符串分别向左平移,平移单位为移位长度值(ml)个字符,最后不同移位长度的移位操作可获得不同的序列数据集。而移位操作中的长度值范围为:1分段长度值。
例如,序列数据字符串为:GCTGGTCCGCAGCAACACGACCAGGTTGACGTACCGAT,其序列总长度为38,若将分段长度(dl)固定为9,则此时可得到数目为4段的序列数据集:GCTGGTCCG,CAGCAACAC,GACCAGGTT,GACGTACCG。而移位操作中若是移动长度(ml)为2,则将序列数据集中的每条数据序列向左平移2个序列字符,此时以上数据集的移位操作结果就为:TGGTCCGGC,GCAACACCA,CCAGGTTGA,CGTACCGGA。
3.2. RNN分类模块
该模块下,将测试数据集中的数据序列进行移动操作后作为RNN分类器的输入,进行分类操作后,得到相应的结果数据集作为输出,如“图3”。

Figure 3. Flow Chart of Classifier classification
图3. 分类器分类流程图
3.3. 可视化序列操作模块
可视化序列操作主要是两次序列替换操作。
任意一条DNA序列根据四种碱基的不同性质,可以被唯一描述为3种独立的嘌呤(R)和嘧啶(Y)的分布、氨基(M)和羧基(K)的分布、强氢键(S)和弱氢键(W)的分布,本文将三种分布作为替换关系,如下:
1) RY:嘌呤(R) = A、G;嘧啶(Y) = C、T;
2) MK:氨基(M) = A、C;羧基(K) = G、T;
3) SW:强氢键(S) = G、C;弱氢键(W) = A、T;
根据以上对应的替换关系,一条基因序列可映射成三条不同的序列,此为序列的第一次的替换操作,其主要针对的是序列中的元素。
例如:序列GTCCACTGGCATGGT可替换成三条独立的序列:1) RYYYRYYRRYRYRRY;2) KKMMMMKKKMMKKKK;3) SWSSWSWSSSWWSSW。
任意一条序列经过了第一次替换操作后其中的位置关系有四种,如下:
1) RY:‘RR’,‘RY’,‘YR’,‘YY’;
2) MK:‘KK’,‘KM’,‘MK’,‘MM’;
3) SW:‘SS’,‘SW’,‘WS’,‘WW’;
而该序列操作中的第二次替换是在所有位置关系选出特定的位置关系字符串作为替换时的判断依据,将整条数据序列中与之相等的子字符串替换为“1”,否则就替换为“0”,最终将整条序列替换成只包含“0”和“1”的序列。
综上可知,任意一条经过第一次替换的数据序列按照不同的位置替换关系一共可替换成4条“01”序列,作为统计计算模块的输入。
例如,若经过第一次替换后的序列:SWSSWSWSSSWWSSW,则第二次替换结果为:① ‘SW’:10010100010001;② ‘SS’:00100001100010;③ ‘WS’:01001010000100;④ ‘WW’:00000000001000。
3.4. 统计计算模块
统计计算模块主要是统计替换后的“01”序列中“1”字符的总数量,然后计算其测度,用“1”的统计总数除以序列的长度。
同一条序列因为不同的序列替换关系会有相对应不同的统计值集合。
3.5. 可视化模块
该模块中主要包括序列差异、分类结果统计、分类结果数据集可视化操作,以下主要介绍针对不同的可视化操作的数据操作。
3.5.1. 序列差异
序列移位操作前后的差异性可通过编辑距离 [11] 计算两序列相似度 [12] 来量化。其中编辑距离是指两个字串之间,由一个转成另一个所需的最少编辑操作次数。编辑操作包括将一个字符替换成另一个字符,插入一个字符,删除一个字符。一般来说,编辑距离越小,两个串的相似度越大。
编辑距离用数学语言描述有:
其中有:
指的是a字符串中前i个字符和b字符串中前j个字符之间的编辑距离。
其中计算相似度的公式如下:
以及有:
其中m和n分别为两个字符串的长度,而ldista,b表示字符串a和字符串b之间的编辑距离。
计算可得序列进行不同长度的移位操作后序列与不进行移位操作序列之间的相似度集合,该集合就作为序列差异可视化的输入数据。
3.5.2. 分类结果统计
将进行不同长度值移动操作后所得的测试数据集经过RNN分类器分类,然后对结果数据集进行统计操作,即将结果数据集中数据按照种类为单位进行数据统计。
结果可得到一系列不同长度值移动操作下分类结果的种类数量的统计值,将其作为分类结果统计可视化的数据源。
3.5.3. 分类结果数据集统计
此模块中的可视化数据来源于统计计算模块中的数据集,因为可以选择不同的元素替换关系以及不同的位置替换关系,所以可得不同的数据集。其后选择不同的数据值组合作为可视化2维图示的坐标值也就可以得到相应不同的可视化结果,从而获得不同的观察分析角度。
4. 数据简介
文中所涉及的数据为四类细菌的基因序列:① 肠沙门氏菌(Salmonella enterica);② 脓肿分枝杆菌(Mycobacteroides abscessus);③ 桑特氏三角菌(Terriglobus saanensis);④ 苏云金芽孢杆菌(Bacillus thuringiensis)。
所有数据全部来源于美国生物基因数据库NCBI网站。
本文中将分段长度值固定为100,移位长度值范围为1~99。
5. 结果展示与分析
5.1. 结果展示
本文结果主要是结果可视化模块中的内容。
5.1.1. 分类结果数据集统计
将不进行移动操作的数据序列与进行移动操作后的数据序列之间的相似度按照以上公式计算出来,然后计算整个数据集的差异性平均值,最后将其所得平均值可视化,结果如“图4”。
图中其横坐标为序列移动操作中的移动长度值,其为连续性变化数值,范围就为1~99,纵坐标为序列移动前后两序列字符串的相似度计算值。
观察可视化图示结果发现,移动长度数值范围在41~61时,两序列字符串差异性最大,同时对应的相似度也最低。
5.1.2. 分类结果统计可视化
将分类结果统计结果可视化后,可得如“图5”。
统计结果是按照分类操作中序列数据集的种类为单位可视化,本文中所涉及的数据种类数量为4。

Figure 4. Flow Chart of Classifier classification
图4. 序列差异值图示
横坐标为不同的连续序列移动操作的移动长度值,值所取范围是0~99,纵坐标为其中某一类数据所占结果数据集的比例。
从结果图示中,可知在不同的移动长度数值的移动操作条件下,所对应的不同结果数据集中四种不同类别的数据集数量变化并不大。
5.1.3. 分类结果数据集可视化
分类结果数据集统计结果可视化后可以得到一批不同的图示结果,在其中挑选出有特定序列差异值的数据集可视化图示进行展示如图7至图11,对应的移动操作的移动长度分别为0,1,21,41,61,81。
图示结果为二维频次直方图,其中横纵两坐标轴的值分别是数据集经过两次替换操作后的相应统计值,而值的选取可以是多种不同的组合,由于第一次替换关系总共有三种,而每一种替换关系里位置替换关系总共有四种,所以可以获得大量的可视化组合结果图示。图示中颜色的分布信息表示数据分布情况,颜色由‘红黄青蓝’组成,其中蓝色表示数据含量最少的投影,红色表示数据含量最多的投影。
本文中以下可视化结果展示的是:第一次替换关系是‘SW’,第二次位置替换关系是‘SW’和‘WW’,具体结果如下图系列。
其中单张图示中包括(a)、(b)、(c)、(d)四个部分,每个部分分别代表不同的分类结果,从上至下,从左至右顺序依次是:① 肠沙门氏菌(Salmonella enterica);② 脓肿分枝杆菌(Mycobacteroides abscessus);③ 桑特氏三角菌(Terriglobus saanensis);④ 苏云金芽孢杆菌(Bacillus thuringiensis)。
不进行移动操作时,按‘SW’与‘WW’位置关系统计值可得如图“图6”:
进行移位操作,且移位操作中移动长度(mlen)为1,21,41,61,81时,按‘SW’与‘WW’位置关系统计值可如下:
1) 移动长度(mlen)为1可得如图“图7”;
2) 移动长度(mlen)为21可得如图“图8”;
3) 移动长度(mlen)为41可得如图“图9”;
4) 移动长度(mlen)为61可得如图“图10”;
5) 移动长度(mlen)为81可得如图“图11”;
观察图示可发现,分类结果数据集的变化并不大,虽然数据分布情况仍有变动,但是数据集数据整体分布情况变化不大。
综上,将图6作为对比项,较之图7至图11可视结果中的差异数据统计占比如“表1”:

Table 1. System resulting data of standard experiment
表1. 标准试验系统结果数据
5.2. 结果分析
综上所有的结果图示,虽然在移动长度为41~61的范围内进行移动操作后的序列与不进行移动操作的序列两者之间的相似度最低即差异性最大,但在该区间范围内分类结果数据集的每个种类分类的结果统计量变化不大,且单一种类的结果数据集的数据分类情况整体稳定,具有固有的分布特征。虽然本文处只展示了‘SW’和‘WW’一种组合的图示结果,但是其他组合结果图示在同样的操作流程下也是可得的,并且所有图示所反映结果信息的也是一致的。
RNN分类器在序列元素位置变化的情况下,分类操作时仍然是稳定的,序列数据中元素的位置改变在一定范围内对RNN分类器的影响是十分有限的。
6. 总结
本文利用变值测量模型对RNN分类器的稳定性进行了初步的分析与探索,其处理的流程主要包括:序列操作模块、RNN分类器分类模块、统计计算模块、结果可视化模块。其中多种组合的图示结果提供了较为全面的观察与分析角度,从数据分布特征对RNN分类器的分类稳定性研究提供了一种可行的处理流程和方法,为观察RNN分类器结果的稳定性提供了系列可视的比较结果。
基于目前的研究结果,整个框架仍有可拓展的部分,虽然可视化结果中展示出了RNN分类器的稳定,但是其中仍有细微的差异,可将差异作为独立的可视化单元进行进一步的分析与探索。
致谢
感谢云南大学软件学院,感谢云南省软件工程重点实验室提供良好的工作环境。感谢云南省科技计划项目(KC1810123)对该项目提供的资金支持。
NOTES
*通讯作者。