1. 引言
乳腺癌是女性癌症中最常见的疾病之一,其发病率在癌症发病率中位居世界第二。根据美国癌症协会(ACS)的统计,乳腺癌可分为5个阶段,早期乳腺癌5年生存率可达100%,晚期乳腺癌5年生存率仅为22%。可见,乳腺疾病的早期诊断对于乳腺肿瘤的治疗至关重要。传统的病理诊断可以获得诊断的病变组织诊断结果的同时却出现了形态学改变的问题,难以实现早期诊断。拉曼光谱具有无损、直接测量而无需进行预处理等优点,因而越来越多的研究致力于将该技术应用于疾病的检测和诊断中,拉曼光谱在乳腺组织检测方面成为新的研究热点。Michael等 [1] 于2005年将特征提取方法主成分分析(PCA)应用到乳腺疾病的鉴别当中,开辟了乳腺组织拉曼光谱数据处理的新思路。2013年胡成旭等人应用PCA [2] 和高斯过程的机器学习 [3] 识别乳腺疾病类型。但是特征提取方法 [4] [5] 将原始数据变换到另外一个空间,其重点在于实现了对一个数据集的全局结构的降维,仍缺乏对拉曼光谱数据细致的分析;特征选择方法 [6] 则在不改变原始数据结构的基础上,选择出最优的特征子集,既可使我们得到原始特征的组合,同时机器学习模型的性能也得到了提高。
本研究采用共聚焦拉曼光谱仪对新鲜乳腺组织进行检测,对所得拉曼光谱数据进行预处理,然后使用ReliefF方法,分析拉曼光谱的每一个特征,得到最优的特征子集,最后构建SVM模型,并引入集成学习的方法提高实验的准确度。结果表明基于机器学习的特征选择方法相比传统的人工选择方法在准确性、敏感性和特异性方面具有更优越的预测性能。
2. 相关工作
与乳腺癌领域相关的拉曼光谱仪(如大型综合分析)显示:放疗可以降低乳腺癌的死亡率。随机对照试验图谱证实:他莫昔芬(tamoxifen)的10年辅助治疗在减少乳腺癌复发和相关疾病的死亡方面优于5年标准治疗。近年来,应用人工智能技术治疗乳腺癌取得了较大进展 [7]。2016年6月,在国际生物医学影像研讨会上,来自贝斯以色列女执事医疗中心(BethIsrael Deaconess Medical Center, BIDMC)和哈佛医学院的研究小组开发研究出一种基于深度学习的人工智能技术,将病理学家的分析与人工智能自动计算诊断方法相结合后,对乳腺癌前哨淋巴结转移的诊断准确率提高到了99.5%。
3. 实验部分
3.1. 标本采集
我们采集了吉林大学第一医院乳腺外科的临床病理组织及其周围的乳腺疾病组织,并对组织进行一定的修剪以便于拉曼光谱检测。实验过程中首先用冻存切片机(leica-cm1950,德国)将冻存的样品进行切片,将临床采集到的乳腺患者的新鲜病灶组织平均切成2部分,其中一份染色后用于病理检测,另一份用于拉曼光谱检测,分别得到病理诊断结果和拉曼光谱数据。标本处理的过程如图1所示。实验共获得454条拉曼光谱数据,对应的病理检测结果显示有234条良性组织数据和220条恶性组织数据,每条数据由3128个强度值构成。然后对每条拉曼光谱数据进行预处理,完成数据基线校正,数据平滑和数据归一化,建立新鲜乳腺病灶组织的拉曼光谱数据库。并同时采用SPXY方法将454条拉曼光谱数据划分为训练集和测试集,考虑到尽量减少目标变量的数据分布不均的问题,利用SPXY选取其中394条数据做训练集,60条数据做测试集。文中引入特征选择方法ReliefF对拉曼光谱数据中的3128个光谱强度进行特征权重计算以减少数据的冗余和噪声。实验共迭代10次计算权重的平均值,并根据权重值对3128个光谱强度进行排序,最终保留了权重较大的34维光谱强度。以34维光谱强度为特征,应用支持向量机方法构建乳腺病灶识别模型。在SVM理论的基础上运用另外一种机器学习方法——集成学习,不仅可以获得较高的泛化能力,而且可以获得很好的分类性能。

Figure 1. The process of sample detection
图1. 标本检测具体流程
3.2. 仪器
本实验使用共聚焦拉曼光谱仪来采集光谱数据,根据对显微镜物镜的观察和标记,扫描同一块组织的多个点(扫描波长范围为600~2000 cm−1)后得到10~15个扫描点的拉曼光谱数据。扫描每个点的积分时长为60 s,最终得到454条光谱数据,对应的病理检测结果为良性组织234条,恶性组织220条。
3.3. 光谱预处理
由于拉曼光谱数据存在维数大、异常值大、噪声大等缺陷,直接利用拉曼光谱数据诊断乳腺良恶性肿瘤很难取得有价值的结果 [7],可信度有所欠缺。所以在构建模型之前需要对原始的光谱数据进行预处理,以防直接利用这些数据建立分类模型可能出现过拟合的问题。图2展示了预处理过程中的数据基线校正,数据平滑和数据归一化。
 (a)
(a)  (b)
(b)  (c)
(c)
Figure 2. (a) Raw roman spectra; (b) Raman spectra with baseline processing; (c) Raman spectra after smooth processing
图2. (a) 原始拉曼光谱图;(b) 基线处理后拉曼光谱图;(c) 平滑处理后拉曼光谱图
图2显示了经过基线校正和光滑处理后的光谱图。图2(a)给出了共聚焦拉曼光谱仪采集到的原始光谱数据图,之后采用NGSLabSpec软件上的Baseline方法完成基线校正,以解除由于仪器、背景、扫描次数等因素导致的基线漂移现象。图2(b)就是对原始数据进行基线校正后的光谱图。为了消除图2(b)中的噪声,本文引入邻近点比较法完成数据平滑。邻近点比较法将每个样本的每个数据点的值和邻近的数据点的值进行比较,验证是否存在干扰信号。当一个数据点与临近数据点的数值相差太大,超过给定的阈值时,便将该点的拉曼光谱强度值用邻近点的平均值代替。图2(c)是数据平滑处理后的光谱图,图中显示出平滑处理消除了测试变量以随机误差和偏移的方式出现的噪声数据。癌症的发生与发展涉及基因变异、表观遗传改变、基因表达异常以及信号通路紊乱等诸多层次的复杂调控机制。归一化的目的是让不同维度之间的特征在数值上有一定可比性,以提高数据分析的精度和模型优化的速度。归一化处理需要利用特征选择算法进行降维处理,选出每组数据的最优特征数,有Min-max,Z-score和Sigmoid函数等方法。本文用样本的每一个峰位值除以样本数据中最高的峰值完成样本的归一化,提高了模型的收敛速度。
4. 建模部分
4.1. 子数据集划分
划分训练集和测试集是建模的重要步骤,训练集用来训练识别模型,测试集用来测试模型的稳定性。本文采用SPXY样本集划分方法 [8] 将整个数据划分为两个子集,通过计算数据的欧几里德距离,经过逐步选择形成一个训练集;另一个集合是由其他数据组成的测试集,它考虑了目标变量,极大地提高了模型的预测性能。本文利用SPXY方法最终将454条拉曼光谱数据中的394条数据做训练集,60条数据做测试集。
4.2. ReliefF特征选择算法
特征选择是建模前非常关键的工作,应用不同的特征建模会得到不同精度的识别模型。Kira和Rendell提出的ReliefF [9] 法是比较典型的特征选择算法。ReliefF根据各个特征和类别的相关性赋予特征不同的权重,权重大于规定阈值的特征组合形成最优的特征子集。通常,权重值是根据相似样本和不同样本之间的距离计算的。如果特征与相似样本之间的距离较短,且不同样本之间的距离特别长,则赋予特征更大的权重。特征权重算法具体过程如下:随机从训练集中选择一个样本,先从同类的样本中寻找该样本的最近邻样本,再从不同类的样本中寻找该样本的最近邻样本,计算选择样本与两个最近邻样本的距离,然后根据以下规则更新每个特征的权重:如果选择样本与同类的最近邻样本之间的距离小于与不同类最近邻样本之间的距离,则增加该特征的权重,说明该特征对区分同类和不同类的最近邻样本是有益的;反之,则降低该特征的权重,说明该特征对区分同类和不同类的最近邻样本起负面作用。重复以上过程,最后得到每个特征的权重。具体公式如下:
(1)
(2)
其中diff(A, R1, R2)表示样本R1与R2在特征上的差异,Mj(c)表示不同类别C中的第j个最近邻样本。结果表明:当权值较大时,越有利于对当前数据进行分类;而当权值较小时,对当前数据的分类能力则相对较弱。
4.3. SVM算法
支持向量机最初是在Vapnik的结构风险最小化(SRM)原理中实现的,具有较低的泛化误差,且不存在过拟合问题。Xu等人使用基于支持向量机的特征选择方法实现了应用基因特征预测乳腺癌的生存率 [10]。该方法采用特征选择算法处理高维的特征集合,利用径向基函数解决小样本、非线性和高维度的问题 [11]。如图3所示,它的主要思想是建立一个分类超平面作为决策曲面,使得正例和反例之间的隔离边缘间距最大,其神经网络的构成包括三层,其中每一层都有着完全不同的作用。输入层由一些感知单元组成,它们将网络与外界环境连接起来;第二层是网络中仅有的一个隐层,用于从输入空间到隐层空间之间进行非线性变换,在大多数情况下,隐层空间有较高的维数;第三层的输出层是线性的,它为作用于输入层的激活模式提供响应。本研究的主要目标是区分恶性乳腺组织与良性乳腺组织,属于二分类问题 [12]。
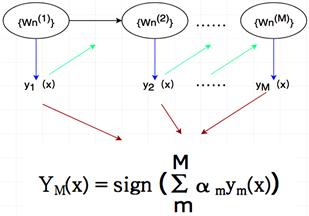
Figure 3. Kernal function’s machine architecture
图3. RBF径向基函数的机器结构
4.4. 集成学习方法
集成学习理论和算法的研究一直是机器学习领域的一个热点,在人脸识别、地震信号分类、精确图像分析等许多领域都有着广泛的应用,在医学领域中模型的构建方面也逐渐成为一个重要的实践。
Schapire于1990年首次提出boosting算法,1993年Drucker和Schapire首次将神经网络作为基本学习器,并将boosting应用于实践中。Wolpert于1992年提出了stacking的一般结构,并将其应用于实际数据集,用来减小模型的推广误差。1996年,Breiman提出了类似boosting的bagging技术,强调基学习器在集成学习稳定性方面对预测精确度的较大影响 [13]。实战证明集成学习通过多次迭代运算确实可以达到将弱分类器训练为强分类器的效果。
本文利用迭代算法AdaBoost结合SVM构造强分类器的方法,在权值设置不同的情况下,弱分类器的分类结果也优于随机猜测。根据前一次迭代的分类错误率ε,AdaBoost可以构造强分类器更新训练集中每个样本的权重值D,新的权重D是与ε相关的函数。对于分类错误率ε较小的样本赋予较小的权值D,而分类错误率ε较大的样本赋予较大的权值D [14],通过设置权值使整个样本的分类错误率降到所期望的范围内。
除了提高预测精度、提高学习模型的稳定性、减少小样本的出现、避免过拟合外,集成学习的优势还在于其基学习器输出的多样性、对参数选择的改进及对整体泛化性能的提高方面。
5. 实验结果分析
5.1. 特征选择结果
在波长为600~2000 cm−1的扫描范围,拉曼光谱的每一个激发波长点上都会有不同的光谱强度。由于组织化学成分的不同,当给定相同的激发波长时,特征峰和强度表示出多样性。
表1所示列出了不同峰位对应化学组成如下。


Table 1. Peak assignments (cm−1) of the Raman spectra of breast tissue
表1. 乳腺组织拉曼光谱的特征峰归属表(cm−1)
本研究应用ReliefF算法计算特征值的权重,将所有光谱强度从1到3128按顺序编号,并将每个光谱强度视为一维特征。在算法运行之前先设置一个阈值,通过迭代10次来计算特征的权重平均值并选出平均值大于阈值的特征,应用筛选出的特征构建模型。图4给出了用ReliefF算法计算乳腺组织数据特征权重的散点图。图5的气泡图描述了从2300到2600的特征权重值正是上表中1741 cm−1和1745 cm−1波长处脂质的特征峰。图中横坐标表示属性编号所对应值的波长(cm−1),纵坐标表示属性编号所对应的特征权重值(%)。由图可知,所选阶段的特征属性权重值随着波长的增加也呈线性增长的趋势,且变化较稳定集中,对应于图4中红线所标出的范围。

Figure 4. Characteristic weights of breast cancer calculated by the ReliefF algorithm
图4. 用ReliefF算法计算乳腺癌特征权重
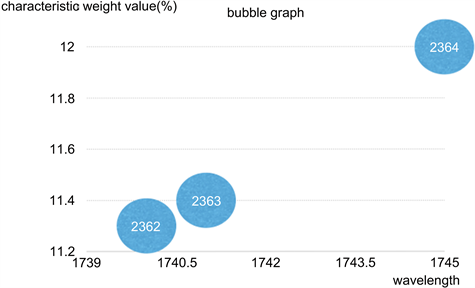
Figure 5. Feature weight of the attribute
图5. 属性的特征权重
5.2. 支持向量机的结果
通过图6可以观察到基于支持向量机模型的分类结果。垂直坐标代表乳腺病变的分类结果,(ο)代表疾病类型,(*)代表预测值,只有当两者一致时,预测才是正确的。图中显示有3个良性组织数据被预测为恶性组织,只有一个恶性组织数据被预测为良性组织。因此,本实验整体预测的准确性为93%,灵敏度为96%,即96%的乳腺癌患者可以被检测到,从而有效地降低了漏诊率,特异度为91.5%,验证了支持向量机分类具有良好的泛化性能,所预测的精度基本满足医学要求。

Figure 6. Classification results of SVM
图6. 支持向量机分类结果
5.3. 集成学习的结果
为了增强分类效果,本研究利用AdaBoost构造强分类器,经过AdaBoost的迭代运算,总体分类精度有所增加。表2对比了原始SVM处理后和经过AdaBoost组成强分类器分类后的数据结果。与简单支持向量机的两种分类结果相比,对角线上的元素进一步增加:总体分类准确率提高到95.1%,且灵敏度为96%,特异性为94.3%,假阳性率较低,也没有出现过拟合现象,从而再次证明了该方法可以有效提高分类的效果和精度。

Table 2. The results of support vector machine and integrated learning method
表2. 支持向量机与集成学习方法处理后的结果对比
6. 结论
本文介绍了利用共焦拉曼光谱技术对新鲜病变乳腺组织进行检测的过程,建立了乳腺良恶性组织的数据库。经过数据预处理,利用ReliefF算法提取光谱的主要特征从而简化原始数据。将数据分为训练集和测试集也有助于建立支持向量机的分类模型,采用AdaBoost集成学习的方法,通过改变权值,在不丢弃特征的情况下达到较好的预测效果,既提高了数据的准确性,也提高了分类性能。将计算机应用技术中机器学习算法应用于模型的建立,得到的预测精度在一定意义上说明了本文的研究方法可用于良恶性组织的鉴别同时也为乳腺癌的诊断提供了参考。希望本文所研究内容能够对乳腺癌的治疗有所帮助,若有可能但愿在临床上可以得到广泛的应用。
基金项目
国家自然科学基金面上项目(81773171)。
NOTES
*通讯作者。