1. 引言
2020年12月,联合国粮农组织发布了全球首篇关于全球土壤生物多样性的研究报告,报告指出土壤退化是与全球气候变暖、地表环境破坏同等重要的生态环境问题 [1]。我国是世界上土地退化沙化危害最严重的国家之一 [2]。全国有荒漠化土地261.16万平方公里,占国土面积的27.2%;沙化土地172.12万平方公里,占国土面积的17.9% [3]。内蒙古自治区为我国荒漠化第二大省区,包含五大沙漠、四大沙地,全区1/2的土地遭受荒漠化侵蚀,其中科尔沁沙地的面积最大 [4],已成为我国北方半干旱农牧交错带荒漠化最严重的地区 [5]。科尔沁沙地曾是水草丰美的大草原,然而由于近代人类对水资源环境的破坏再加上气候干旱,逐渐演变成我国面积最大的沙地 [6]。60年代初期,美国学者曾使用衬膜技术进行过蔬菜的试验性种植 [7]。1962年,日本学者内岛善兵卫等于日本岛根县沙地建立衬膜水稻试验田 [8],研究表明对沙地进行衬膜工程改造以达到水稻种植的目的是可行的,但由于当时的技术原因,衬膜改造工程量大,成本过高,故该技术没有得到推广。同年松岛省三也进行了小面积的沙地衬膜水稻种植并展开相关研究,取得了一定的成果 [9]。
本课题组于2014年起在科尔沁沙地试验地应用衬膜技术进行水稻种植,2016年种植面积达到5000亩。经过多年的实践与研究发现,与荒漠化沙地土壤相比,经过衬膜沙地水稻技术处理修复的沙地水稻种植田的土壤的有机质、氮、磷、钾的土壤含量大幅度提高 [10],土壤肥力得到恢复,土壤的修复效果明显。土壤质量是一个综合概念,包含了多方面的内容 [11]。目前,土壤质量评价方法尚未有统一的标准 [12]。以往的指标多是从土壤肥力方面进行研究,本研究选取了微生物多样性这一评价指标。土壤微生物参与土壤生态系统的能量流动和养分循环 [13],影响生物的地球化学循环过程,其多样性和群落组成在决定土壤生态系统的功能和可持续性中起着重要作用 [14]。本试验从微生物多样性角度,对采用衬膜水稻种植技术修复的荒漠化土壤修复效果进行评价,建立基本的数据基础,为衬膜水稻技术的推广提供重要依据,从而促进荒漠化地区土壤生态评价系统的完善。
2. 材料和方法
2.1. 试验地概况
该试验地位于科尔沁沙地南部,行政区划隶属于内蒙古自治区通辽市奈曼旗,该地位于东经120˚19'40''~121˚31'44'',北纬42˚14'40''~43˚32'30'',海拔约360 m,气候属大陆性温带半干旱气候,降雨主要集中6~8月,年平均降水量335.9 mm,年平均气温6.8℃,年最大蒸发量1104.6 mm,最小蒸发量871.5 mm。
2.2. 样品采集
2020年9月进行采样。为了保证野外采样的科学性,在地形地貌等土壤环境条件基本一致的内蒙古自治区通辽市奈曼旗科尔沁沙地内选取了进行衬膜水稻种植的样地和邻近荒漠化沙地样地。以五点法进行采样,将水稻连根拔出,取水稻根际土,土样以四分法缩分至1 kg装袋保存,记录采样时间和采样深度,带回实验室,剔除石子根茎等杂质,置于冷冻保存。
2.3. 研究方法
试验设计:本研究通过定期对衬膜水稻试验田进行采样,对衬膜水稻种植的样地和邻近未经修复的荒漠化沙地的微生物多样性进行比较分析。
土壤测定:高通量测序技术是探讨微生物菌群的不可或缺的技术 [15] [16] [17],高通量测序技术的发展,使我们能在短时间内测定大量物种的核酸序列,对某个菌种,我们可以测定上百株细菌的基因组序列,这就为我们进行基于全基因组信息的分类标准建立奠定了良好基础 [18]。本试验的细菌多样性采用16SrDNA高通量测序技术进行测序。实验步骤如下:
1. 基因DNA的提取
完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。
2. PCR扩增
按指定测序区域,合成带有barcode的特异引物,或合成带有错位碱基的融合引物。
为保证后续数据分析的准确性及可靠性,需满足两个条件:
1) 尽可能使用低循环数扩增;2) 保证每个样本扩增的循环数一致。随机选取具有代表性的样本进行预实验,确保在最低循环数中使绝大多数样本能够扩增出浓度合适的产物。
PCR产物使用1%琼脂糖凝胶电泳检测扩增目的条带大小,并用Agencourt AMPure XP核酸纯化试剂盒纯化。
3. Miseq文库构建
1) 连接“Y”字形接头;
2) 使用磁珠筛选去除接头自连片段;
3) 利用PCR扩增进行文库模板的富集;
4) 氢氧化钠变性,产生单链DNA片段。
4. Miseq上机测序
1) DNA片段的一端与引物碱基互补,固定在芯片上;
2) 另一端随机与附近的另外一个引物互补,也被固定住,形成“桥(bridge)”;
3) PCR扩增,产生DNA簇;
4) DNA扩增子线性化成为单链;
5) 加入改造过的DNA聚合酶和带有4种荧光标记的dNTP,每次循环只合成一个碱基;
6) 用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类;
7) 将“荧光基团”和“终止基团”化学切割,恢复3'端粘性,继续聚合第二个核苷酸;
8) 统计每轮收集到的荧光信号结果,获知模板DNA片段的序列。
3. 结果与分析
对荒漠化沙地土壤和衬膜水稻土壤两种不同土壤中的6个土壤样品进行测定。未经修复过的荒漠化沙地土壤编号为S1、S2、S3,经衬膜水稻种植技术修复的水稻土壤编号为W1、W2、W3。
3.1. OTUs聚类分析
通过对序列进行归类操作可以了解一个样品测序结果中的菌种、菌属等数目信息。将序列按照彼此的相似性分归为许多小组,一个小组就是一个OTU。根据不同的相似度水平,对所有序列进行OTU划分,通常对97%相似水平下的OTU进行生物信息统计分析。得到的OTU数目如表1。

Table 1. Statistics of OTU number of single samplet
表1. 单个样本的OTU数目统计
为了比较直观的表现两组样本的OTU数目组及其样本或分组之间的重叠情况,绘制了可用于统计多个样本中所共有和独有的OTU数目的Venn [19] 图,如图1。根据OTU数目韦恩图可以看出,两种土壤之间OTU数目差异很大,荒漠化沙地土壤的OTU数目远远大于衬膜水稻土壤,两种土壤相同的OTU数目为1877。荒漠化沙地土壤样本间的差异性大于衬膜水稻土壤。

Figure 1. The number of OTU Venn diagram
图1. OTU数目韦恩图
3.2. Alpha多样性分析
3.2.1. 稀释性曲线
如图2,样品稀释曲线 [20] 趋近平缓。曲线趋向平坦时,说明测序数据量合理,更多的数据量只会产生少量新的OTU。因此,稀释性曲线表明样品目前的测序量能够覆盖样本中的绝大部分物种,测序深度足够,测序数据量合理。
3.2.2. Shannon-Wiener曲线
如图3,Shannon-Wiener曲线趋向平坦,说明测序数据量足够大,可以反映样本中绝大多数的微生物信息。此外,由Shannon-Wiener曲线图可看出荒漠化沙地土壤样品的Shannon指数大于衬膜水稻土壤。

Figure 3. Shannon-Wiener graph
图3. Shannon-Wiener曲线图
3.2.3. Rank-Abundance曲线
Rank-abundance [21] 曲线是分析多样性的一种方式。Rank-abundance曲线可用来解释多样性的两个方面,即物种丰度和物种均匀度 [22]。在水平方向,物种的丰度越高,曲线在横轴上的范围越大。荒漠化沙地土壤的曲线跨越宽度大于衬膜水稻土壤,表明荒漠化沙地土壤的物种的丰度大于衬膜水稻土壤。曲线的形状(平滑程度)反映了样本中物种的均度,曲线越平缓,物种分布越均匀。与衬膜水稻土壤相比,荒漠化沙地土壤的曲线更平缓,表明荒漠化沙地土壤的物种的均度大于衬膜水稻土壤(见图4)。

Figure 4. Rank-abundance graph
图4. Rank-abundance曲线图
3.2.4. Alpha多样性指数
如图5 alpha多样性指数box图,具体数据见表2 alpha多样性指数统计表。

Table 2. Alpha diversity index statistical table
表2. Alpha多样性指数统计表

Figure 5. Alpha diversity index box map
图5. Alpha多样性指数box图
Chao 1:即菌种丰富度指数,用以估计群落中的OTU数目。衬膜水稻土壤样品的chao 1指数大于荒漠化沙地土壤,表明衬膜水稻土壤菌种的丰富度大于荒漠化沙地土壤,衬膜水稻土壤细菌群落的丰富度高。
goods_coverage和observed_species指数表征各样本文库的覆盖率,其数值越高,则样本中序列被测出的概率越高,而没有被测出的概率越低。该指数反映本次测序结果代表了样本中微生物的真实情况。
PD_whole_tree [23]:谱系多样性,是兼顾考虑了物种丰度以及进化距离的多样性指数。数值越大,群落多样性越高。荒漠化沙地土壤样品的PD_whole_tree指数大于衬膜水稻土壤样品,因此,表明荒漠化沙地土壤群落多样性高于衬膜水稻土壤。
Shannon:用来估算样品中微生物多样性指数之一。Shannon值越大,说明群落多样性越高。荒漠化沙地土壤样品的Shannon值大于衬膜水稻土壤样品,表明荒漠化沙地土壤群落多样性高于衬膜水稻土壤。
Simpson [24]:用来估算样品中微生物多样性指数之一,结果中交付为1-D值。
Simpson指数兼顾丰富度和均匀度。D值在0~1之间,0表示无限多样,1表示没有多样性。所以1-D值越大,说明群落多样性越高。荒漠化沙地土壤样品的Simpson值大于衬膜水稻土壤样品,表明荒漠化沙地土壤群落多样性高于衬膜水稻土壤。
3.3. 物种组成分析
使用统计学的分析方法,观测两种土壤样本在不同分类水平上的群落结构。将多个样本的群落结构分析放在一起对比时,还可以观测其变化情况。本研究用柱状图可视化观察不同样本(或者分组)的物种组成情况 [25]。从门、纲、目、科、属等五个水平上作柱状图。
3.3.1. 两种土壤中微生物菌门组成
图6展示了两种土壤中门水平细菌群落结构,图中不同颜色色块代表不同门细菌种类,高度代表某一个门相对丰度的大小。结果表明,在门水平上各处理优势种群结构相似,但两种土壤细菌丰度存在差异。衬膜水稻土壤中变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)和放线菌门(Actinobacteria)是优势菌群,相对丰度分别为34%、13%和6.7%。荒漠化沙地土壤中变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和酸杆菌门(Acidobateria)是优势菌群,相对丰度分别为30%、23%和13%。采用衬膜水稻技术修复土壤和邻近荒漠化沙地土壤两种土壤所含变形菌门丰度相差不大。荒漠化土壤的放线菌门是衬膜水稻的三倍多,出现这种现象应该是由于放线菌一般为好气性,而荒漠化土壤都是沙土,透气性强。荒漠化土壤中所出现的酸杆菌门是新近被分出的一门细菌,对其知之甚少。在衬膜水稻土壤中的另一优势菌群为绿弯菌门,为兼性厌氧生物在有氧无氧环境都能生存。

Figure 6. Abundance of soil bacterial division level
图6. 土壤微生物菌门丰度图
3.3.2. 两种土壤中微生物菌纲组成
图7展示了两种土壤中纲水平细菌群落结构。结果表明,在纲水平上两种土壤优势种群结构存在很大差异,由于含量较少在图中无法显示,衬膜水稻土壤的菌纲种类略多于衬膜水稻土壤,细菌丰度也存在差异。衬膜水稻土壤中β变形菌纲(Betaproteobacteria)、放线菌纲(Actinobacteria)和δ-变形菌纲(Deltaproteobacteria)是优势菌群,相对丰度分别为13%、10.7%和9.8%。荒漠化沙地土壤中β-变形菌纲(Betaproteobacteria)、α-变形菌纲(Alphaproteobacteria)和放线菌纲(Actinobacteria )是优势菌群,相对丰度分别为10.7%、10.5%和9.7%。衬膜水稻土壤和荒漠化沙地土壤中的优势菌群都集中在放线菌门和变形菌门,与对两种土壤进行的菌门分析呈现一致性。
3.3.3. 两种土壤中微生物菌目组成
图8展示了两种土壤中目水平细菌群落结构。结果表明,在目水平上两种土壤优势种群结构存在很大差异,由于含量较少在图中无法显示,衬膜水稻土壤的菌目种类略多于衬膜水稻土壤,细菌丰度也存在差异。衬膜水稻土壤中厌氧绳菌目(Anaerolineales)、微球菌目(Micrococcales)和伯克氏菌目(Burkholderiales)是优势菌群,相对丰度分别为8.9%、8.5%和6.7%。荒漠化沙地土壤中伯克氏菌目(Burkholderiales)、Blastocatellales菌目和鞘脂杆菌目(Sphingobacteriales)是优势菌群,相对丰度分别为7.2%、5.2%和4.9%。
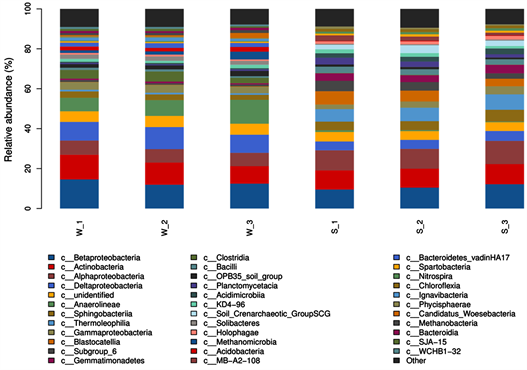
Figure 7. Abundance of soil bacterial class level
图7. 土壤微生物菌纲丰度图

Figure 8. Abundance of soil bacterial order level
图8. 土壤微生物菌目丰度图
3.3.4. 两种土壤中微生物菌科组成
图9展示了两种土壤中科水平细菌群落结构。结果表明,在科水平上两种土壤优势种群结构存在很大差异,由于含量较少在图中无法显示,衬膜水稻土壤的菌科种类略多于衬膜水稻土壤,细菌丰度也存在差异。衬膜水稻土壤中无花果科(Anaerolineaceae)、微球菌科(Micromonosporaceae)和丛毛单胞菌科(Comamonadaceae)是优势菌群,相对丰度分别为8.9%、7.2%和4.0%。荒漠化沙地土壤中Blastocatellaceae_Subgroup_4、芽单胞菌科(Gemmatimonadaceae)和噬几丁质菌科(Chitinophagaceae)是优势菌群,相对丰度分别为5.3%、3.8%和3.8%。
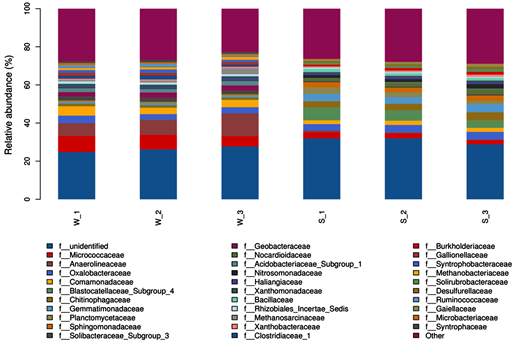
Figure 9. Abundance of soil bacterial family level
图9. 土壤微生物菌科丰度图
3.3.5. 两种土壤中微生物菌属组成
图10展示了两种土壤中属水平细菌群落结构。结果表明,在属水平上两种土壤优势种群结构存在很大差异,由于含量较少在图中无法显示,衬膜水稻土壤的菌属种类略多于衬膜水稻土壤,细菌丰度也存在差异。衬膜水稻土壤中假节杆菌属(Pseudarthrobacter)、Anaerolinea和马赛菌属(Massilia)是优势菌群,相对丰度分别为7.2%、3.4%和2.6%。荒漠化沙地土壤中RB41、假节杆菌属(Pseudarthrobacter)和马赛菌属(Massilia)是优势菌群,相对丰度分别为4.4%、2.5%和2.4%。
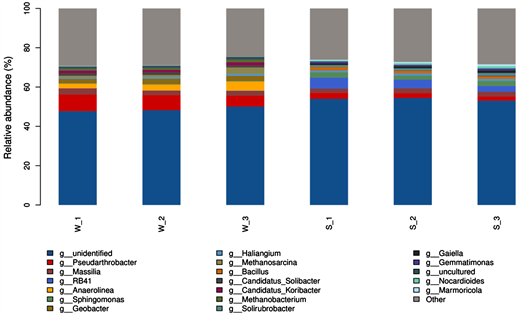
Figure 10. Abundance of soil bacterial genus level
图10. 土壤微生物菌属丰度图
3.4. Beta多样性分析
3.4.1. 基于Unifrac的Heatmap
为了更好的显示多个样本之间的距离关系,将未加权与加权的unifrac [26] [27] [28] 样本距离矩阵用热力图表现出来,如图11所示。衬膜水稻土壤样本之间距离比较小,说明衬膜水稻土壤样本之间的相似性比较大,样本具有代表性。荒漠化沙地土壤样本之间距离略大于沙漠水稻土壤样本,但仍距离比较小,样本比较具有代表性。很明显衬膜水稻土壤样品与荒漠化沙地土壤样品之间距离多远,两种土壤差异性很大。
3.4.2. PCA分析与PCoA分析
PCA分析(Principal Component Analysis),即主成分分析,是一种对数据进行简化分析的技术,这种方法可以有效的找出数据中最“主要”的元素和结构,去除噪音和冗余,将原有的复杂数据降维,揭示隐藏在复杂数据背后的简单结构 [29]。如图12所示,群落结构组成相似度高的样品倾向于聚集在一起,群落结构组成差异很大的样品则会远远分开。衬膜水稻土壤样品间距离非常接近,表示群落组成结构相似。荒漠化沙地土壤样品间距离则偏大,由于沙漠沙地属于自然形成,范围比较大,衬膜水稻样地为人工生态系统,出现的这种情况较为正常。衬膜水稻土壤样品与荒漠化沙地土壤样品距离较大,群落结构组成差异性较大。
PCoA分析,即主坐标分析(principal co-ordinates analysis),也是一种非约束性的数据降维分析方法,与PCA分析类似;主要区别在于,PCA基于欧氏距离,PCoA基于除欧氏距离以外的其它距离。如图13所示。PCoA分析结果与PCA分析结果一致。

Figure 13. PCoA analysis of bray-curtis
图13. 基于bray-curtis的PCoA分析
3.4.3. NMDS分析
非度量多维尺度法(NMDS)是一种将多维空间的研究对象(样本或变量)简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法 [30]。其特点是根据样本中包含的物种信息,以点的形式反映在多维空间上,通过点与点间的距离体现不同样本间的差异程度 [31]。衬膜水稻土壤样品间距离非常接近,表示样本间差异性较小。荒漠化沙地土壤样品间距离则偏大,表示样本间差异性较大。由于荒漠化沙地属于自然形成,范围比较大,衬膜水稻样地为人工生态系统,出现的这种情况较为正常。衬膜水稻土壤样品与荒漠化沙地土壤样品距离较大,物种信息差异性较大(见图14)。
4. 结论
对修复前后两种土壤的细菌群落多样性分析发现,虽然荒漠化沙地土壤的微生物多样性高于修复后的衬膜水稻土壤,但是原有的菌群经土壤修复后会发生变化,衬膜水稻修复技术会改变荒漠化沙地土壤的群落结构,会增加荒漠化沙地土壤优势菌种群落丰度,增加土壤菌落丰度。总的来说,从土壤微生物多样性方面,采用衬膜沙地水稻种植技术修复的荒漠化土壤修复效果显著,值得推广和更加深入的研究。
土壤微生物多样性研究技术难度大、复杂度高,包括物种多样性、遗传多样性、结构多样性及功能多样性等许多方面 [32]。荒漠化沙地作为自然生态系统稳定性强于人工建立的衬膜水稻生态系统是意料当中的,如何减少对沙地生态系统稳定性的影响,增加修复后的土壤微生物多样性也是今后我们研究的重点。寻求合理的修复技术和方案提供数据基础和可行性支持,建立和完善荒漠化土壤的评价体系迫在眉睫。相信在不断地探索和实践中,荒漠化土壤的修复将取得更为巨大的成果,荒漠化地区的人们将迎来更为美好的生活环境,经济发展将迈出历史性的一大步。
NOTES
*通讯作者。