1. 引言
生物信息学作为一门前沿交叉学科,它不仅推动了分子生物学的发展,而且它使得数学与生物学在这个框架中实现了新的结合。随着生物信息学的崛起,遗传序列的系统发育分析变得重要起来,它成为了研究所有类型生物之间进化的必要条件,与此同时了解群体之间的自然关系也非常重要,例如目、科以及属等 [1]。系统发育分析的方法通常依赖于多序列比对,由于生物序列的数量急剧增加,传统的序列比对方法变得不可行,大量基于遗传序列的数字表征的非比对方法被提出,用来补偿传统的基于比对方法的低效性 [2]。
目前在生物序列中的比对中,主要的研究对象是DNA序列与蛋白质序列。在基因组中,遗传信息存储在DNA序列中,同时在DNA序列中,突变在分子水平以或多或少随机的方式发生,从而选择决定了进化的方向 [3]。突变的方式可能是点突变、插入、缺失或者重排,有些非比对方法对于数据缺失会更敏感,可能会造成错误的匹配,因此突变的存在不允许被忽视,本文将突变考虑到DNA序列的两两比对中。
无论是对DNA序列还是蛋白质序列研究,都提出过用K-词的数量与分布来度量相似性,但对于如何选择使用K-词的长度没有一个标准,在文献 [4] 中闻佳等人提出复合K-词联合来唯一地量化每个蛋白质序列,同理这种想法在DNA序列研究中也适用,也就是利用前缀集来唯一刻画每个DNA序列。
1973年Peter Weiner提出了前缀树模型及其算法 [5]。每一个前缀都是唯一出现在序列中的K-词,以单拷贝序列的形式存在于序列中,是整个基因组中独有的,代表了生物序列中独特的特征,因此具有很大的研究价值。有文献利用信息熵,基于共同前缀的位置差提出新的DNA序列相似性度量方法 [6],也有文献利用欧氏距离,针对前缀集提出基于共同前缀的位置差法(CPPA算法) [7],本文在CPPA算法基础上加入错配法则,也就是考虑突变存在,创建了新的模型。通过对70条哺乳动物线粒体DNA序列进行实验,在完全匹配与错配法则的双重查找后得到成对序列最佳匹配的所有位置,再进行位置差计算,最后构建系统发育树,得到优于上述两个文献的结果。
2. 材料
本文应用到的实验数据集来自NCBI (National Center for Biotechnology Information)的Genebank数据库,数据集与文献 [6] 中所用相同,均为70条哺乳动物的线粒体完整基因,经过处理以后的70条DNA序列中,不含有非A、C、G、T的字符。
数据集中序列由原兽亚纲类(Prototheria)、有袋类(Marsupialia)、有胎盘类(Placentalia)三类构成,其中原兽亚纲类序列只来自1个目,有袋类序列来自2个目,有胎盘类序列来自非洲兽总目(Afrotheria)中的3个目、贫齿目(Xenarthra)中的2个目、灵长总目(Euarchontoglires)中的3个目和劳亚兽总目(Laurasiatheria)中的6个目,总共由17个目构成,其中又包含了41个科,因此聚类相对困难。
3. 方法
3.1. 环形前缀标识的定义
对于一条生物序列S,其位置i处长度为m的前缀标识需满足以下两个条件:
1) 从生物序列的第i个位置开始,到第
个位置结束的字符串在序列中是唯一出现的,并且是唯一出现的字符串中长度最小的。
2) 此字符串去掉最后一个字符后,在序列中至少出现两次 [8]。
3.2. 环形前缀集的定义
令
是一条长度为N的核苷酸序列,其中
,
。将序列S的前n个碱基拼接在序列的末端,构成长度为
的新核苷酸序列
,对于新序列S1的第i个位置,都存在唯一的前缀
,由
,
,构成的集合
称为序列S的环形前缀集。
3.3. 完全匹配与错配法则
由于存在基因突变的可能,本文在CPPA算法的前缀树模型基础上进行改进,主要在获取两条序列共同前缀的过程中遵循完全匹配与错配两种法则,其中完全匹配则认为只有每个位置的字符都相同的前缀才是匹配的,例如序列S1前缀集中的ACTT与序列S2前缀集中的ACTT是匹配的,也就是CPPA算法中的匹配方法。
本文加入的允许错配则认为序列S1前缀集中的ACTT与序列S2前缀集中的ACTTA,ACTTC,ACTTG,ACTTT均是可以进行匹配的,最后根据它们的位置信息,在ACTTA,ACTTC,ACTTG,ACTTT中选取与S1中ACTT的最佳匹配。错配原则是在完全匹配原则进行之后,在剩余前缀集中进行的。
3.4. 模型介绍
第一步:利用环形前缀树法构建n条序列的环形前缀集
,
,接下来成对处理序列Si与Sj,其中
。
第二步:根据完全匹配原则,获取Si与Sj序列的共同前缀集
,同时记录共同前缀的个数n,并且记录共前缀集eij在序列Si与序列Sj中的位置(与ACCP法中位置的处理方式相同,本模型中获取的位置信息也需标准化),分别记为
,
。对应的位置差为:
。(1)
第三步:获取Si与Sj序列的剩余前缀集
与
,并且记录剩余前缀的位置集
,
,其中
,
。
第四步:将剩余前缀集
与
中的前缀均去掉最后一个字符,获得新的字符串集合
与
,利用错配原则,获取交集
,以及交集
,同时记录交集的总个数m,在记录两个交集中的元素在Si与Sj的标准化位置时,会出现交集中的某个元素在Si与Sj出现的位置不是一对一,而是一对多的情况,这样会出现多个位置差,只需选择最佳的位置差,处理如下:
假设交集中的元素a在Si中出现的标准化位置为L1,在Sj中出现的标准化位置为
,
,
,那么对于元素a的位置差为:
(2)
将第二步与第四步所获得的位置差合并成一维向量:
(3)
第五步:大多数非比对方法都是仅基于计数方法的,即仅考虑公共邻接单词的频率,而无需对其在序列中的位置进行任何校正 [9],本文不仅考虑计数方法,而且还要考虑其在序列中的位置。计数方法被频繁使用是因为计数方法比匹配长度方法在较高的差异情况下更准确 [10],因此在考虑错配时加入的参数一为交集的总个数
,同时基于共同前缀的位置差来计算进化距离,序列间的进化距离越小,序列就越相近,因此加入参数二为序列对的最小长度
,最终定义相似性距离为:
(4)
4. 模型结果与分析结论
4.1. 模型结果
通过用MATLAB软件编写程序实现了本文的模型,运用邻接法构建了进化树,并且用iTOL网站进行画树,得到了更直观的关于70条哺乳动物DNA序列的系统发育树(见图1)。为了体现本实验方法的有效性,同时也用MATLAB软件实现了ACCP法模型,构建了关于70条哺乳动物DNA序列的系统发育树(见图2),还与基于共同前缀的位置差法(见图3)以及傅里叶分析方法 [11] (见图4)的系统发育树的结果都进行比对分析。
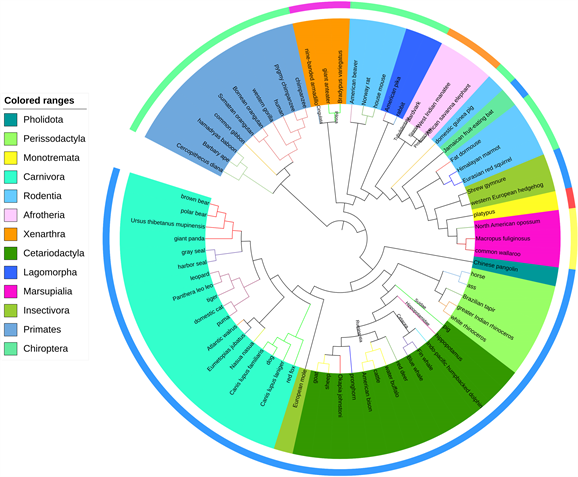
Figure 1. Based on this experimental method, a phylogenetic tree of 70 mammalian mitochondrial DNA sequences was established
图1. 基于本实验方法建立70个哺乳动物线粒体DNA序列的系统发育树

Figure 2. Establishment of a phylogenetic tree of 70 mammalian mitochondrial DNA sequences based on the ACCP method
图2. 基于ACCP方法建立70个哺乳动物线粒体DNA序列的系统发育树

Figure 3. A phylogenetic tree of 70 mammalian mitochondrial DNA sequences by the position difference method based on the common prefix [6]
图3. 基于共同前缀的位置差法建立70个哺乳动物线粒体DNA序列的系统发育树 [6]

Figure 4. A phylogenetic tree of 70 mammalian mitochondrial genomes by The DFT distance of DNA sequences with the 2D numerical mapping [11]
图4. 采用傅里叶分析方法对70条哺乳动物线粒体DNA基因组生成的系统发育树 [11]
4.2. 结果分析
在超目的角度进行分析,图1与图3的进化树中单孔目(Monotremata)、有袋目(Marsupials)、非洲兽总目(Afrotheria)以及贫齿目(Xenarthra)均正确聚类,而图2中的非洲兽总目(Afrotheria)和贫齿目(Xenarthra)发生了错误聚类,均被分成两部分,由此可看出本文的非比对方法已经优于ACCP法 [7]。在图4进化树中,非洲兽总目、贫齿目、劳亚兽总目和灵长总目均没有将序列聚类到各自的类别,聚类结果最差。
在目与科的角度分析,图3中啮齿目(Rodentia)被分成4部分,并且所涉及的4个亚目也未能正确聚类,贫齿目(Xenarthra)中的皮毛目(Pilosa)下的两个目的动物未聚到一起,而图1中的啮齿目(Rodentia)分成3部分,并且亚目下所有的科均聚类正确,同时贫齿目(Xenarthra)中的皮毛目(Pilosa)与有带下目(Cingulata)均聚类正确。图3中灵长目(Primates)的人科(Hominidae)中分进来一个长臂猿科(Hylobatidae)动物,而图1中灵长目(Primates)的所有科均聚类正确,由此可看出本文的非比对方法也优于基于共同前缀的位置差法 [6]。
可以直观看出,无论是在超目,还是目与科的层面分析,本实验结果都是优于傅里叶分析方法,本实验方法不足的是未能解决劳亚兽总目(Laurasiatheria)与灵长总目(Euarchontoglires)的聚类错误问题。
4.3. 结论
通过结果分析可以清楚地知道,把共同前缀标识符作为研究对象,用位置差作为度量因素是有效的方法,将突变考虑进去以后的进化分析模型则是更优的,得到的进化树也符合生物进化关系。但是本实验方法仍然存在不足,比如耗时略长,并且没有考虑到共同前缀标识符的长度以及出现的概率,对于距离的度量仍可以考虑其他因素,因此还有很大的改进空间。
致谢
感谢两位师姐对我的帮助,不仅给我提供了许多参考资料,而且对于软件的应用问题给我耐心讲解,在此由衷感谢。
参考文献