1. 引言
肾细胞癌作为泌尿系统肿瘤中最常见的三大恶性肿瘤之一,其发病率和死亡率一直位于全球男性恶性肿瘤发病率的前几位。根据世界卫生组织国际癌症研究机构(International Agency for Research on Cancer)最新发布的《2020年全球最新癌症负担数据》显示:2020年全球男性新发癌症1007万例,其中肾癌新发病人数27万例,成为了全球男性第九常见癌症 [1] 。肾细胞癌是一种在病理和分子组织上高度异质的疾病 [2] ,包含多种肾细胞癌亚型,每个亚型都有不同的组织学特征、遗传改变、临床表现和治疗反应 [3] 。由于癌症具有较强的隐蔽性,很多病人发现病情时已经是中晚期,这对于癌症的治疗来说非常困难。随着癌症早期筛查诊断技术的完善和推广,癌症患者能够在早期发现病情并及时给予干预和治疗,普遍延长五年生存时间,整体提高生存率。因此,本研究主要针对肾细胞癌早期病例进行分析,以亚型分类的方式了解肾细胞癌亚型的形成机制,在临床上对肾细胞癌的分类诊断治疗具有重要意义。
肾细胞癌的主要亚型包括肾透明细胞癌(Kidney Renal Clear Cell Carcinoma, KIRC)、肾乳头状细胞癌(Kidney Renal Papillary Cell Carcinoma, KIRP)和肾嫌色性细胞癌(Kidney Chromophobe, KICH),分别占肾细胞癌总病例的65%、20%和5% [4] 。除了这三大类外,还有透明细胞乳头状癌、黏液性管状癌和梭形细胞癌等几种更为罕见的癌症 [5] 。对于肾细胞癌亚型的研究前人做了很多分析:Ricketts等 [6] 研究了三个亚型的甲基化模式发现,KIRP相较其他亚型具有较高的甲基化水平。对染色体拷贝数谱进行分析发现KIRC表现出染色体3p的显著丢失和5q的增加,KICH主要表现出1、2、6、10、13及17号染色体显著丢失的模式,且KIRC的突变率高于KICH的三倍 [7] [8] 。Davis等 [9] 发现线粒体代谢途径在KICH中过度激活,而在KIRC中受到抑制。有相关研究 [6] [10] 对TP53和PTEN的突变进行分析,发现它们的突变降低了KIRC和KIRP的生存率。这些研究通过组学之间的对比,发现了不同亚型独特的组学表现形式及分子特征。
肾细胞癌的亚型分类,Han等 [11] 利用基于图像的深度学习框架,使用计算机断层扫描获得的图像来区分肾细胞癌的三种主要亚型,得到了85%的正确率,88%的特异性。Chaudry等 [12] 从完整图像中提取基于纹理、形态和小波的特征,用于RCC亚型分类,通过选择感兴趣区域,分类准确性达到了92%。但伴随着肾细胞癌亚型组学数据的积累和丰富,相比于使用图像数据对肾细胞癌分类研究,建立基于分子特征的分类模型成为可能。此外,利用组学数据研究分类问题能够发现癌症发生的独特机制,进而对癌症的治疗提供帮助。例如Gui等 [13] 基于基因表达谱、体细胞突变和DNA甲基化确定了KIRC的预后良好的基因并找到了出现敏感反应的药物;Deng等 [14] 基于基因表达数据确定了与KIRP的免疫相关的潜在治疗靶点。
在这项研究中,利用DNA甲基化和mRNA表达数据对肾细胞癌亚型早期样本的特征基因进行筛选,寻找到显著分类特征基因,建立分类模型进行分类,并使用独立检验集进行验证;同时对每种亚型的特征基因做GO (Gene Ontology)分析,发现了每种亚型在通路上的不同之处,这为临床上研究肾细胞癌亚型形成机制以及早期诊断治疗提供了理论基础。
2. 材料与方法
2.1. 数据下载与预处理
2.1.1. 数据下载
癌症基因组图谱 [15] (The Cancer Genome Atlas)是世界上最大的基因类工程项目,它包括肾细胞癌在内的30多种癌症类型。本研究的肾细胞癌数据取自该项目(https://portal.gdc.cancer.gov/),主要包括了KIRC、KIRP和KICH的“unstranded”类型的mRNA表达数据和DNA甲基化数据(表1),删除其中临床分期为II-IV期的样本。甲基化阵列平台是illumina Human Methylation450 BeadChip (GPL13534) [16] ,甲基化探针注释从ENCODE project (http://genome.ucsc.edu/ENCODE/downloads.html)下载 [17] 。

Table 1. mRNA data (DNA methylation) statistics of renal cell carcinoma subtypes
表1. 肾细胞癌亚型的mRNA数据(DNA甲基化数据)统计
利用基因表达数据库(Gene Expression Omnibus, GEO, http://www.ncbi.nlm.nih.gov/geo)检索符合条件的肾细胞癌样本集,形成独立检验集。筛选标准:包含“KIRC”、“KIRP”或“KICH”,并包含研究中筛选的所有分类特征基因。本研究通过检索GEO数据库,得到了GSE76351作为独立检验集,该检验集的注释平台是GPL11532,包含了12例肾透明细胞癌。
2.1.2. 数据预处理
本研究使用的是mRNA表达数据和DNA甲基化数据。考虑到影响基因表达的DNA甲基化主要发生在启动子区域 [18] ,选择转录起始位点上游200 bp (TSS200)的基因组区域的探针,并将该区域所有探针的β值的均值作为该基因的甲基化水平。
对任一肾细胞癌亚型样本数据集,由于基因数据差异较大并且维度较高,为了减少后续工作的复杂度,删除在所有样本中表达为“0”的基因,对剩余基因进行归一化处理。归一化的具体操作如公式(1):
(1)
其中Xi是各样本在某一基因的表达值,Xmin是某一基因在各样本中表达量的最小值,Xmax是某一基因在各样本中表达量的最大值。
2.2. 肾细胞癌不同亚型分类特征基因的筛选
1) 肾细胞癌亚型差异基因筛选。对特定肾细胞癌亚型的癌症样本和正常样本:计算基因表达数据(和甲基化数据)与样本分类标签之间的Spearman [19] [20] 相关系数,筛选绝对值大于0.5的基因,并使用单因素方差分析 [21] [22] 筛选出样本中的差异显著性基因。在基因表达数据中,符合|log2FC| > 1的基因作为差异表达基因 [23] (Differentially Expressed Gene, DEG);考虑到甲基化数据对基因表达的影响,将符合|log2FC| > 0.5的基因作为差异甲基化基因(Differentially Methylation Gene, DMG)。
2) 肾细胞癌亚型特征基因筛选。保留基因表达和DNA甲基化共有的癌症样本。针对特定肾细胞癌亚型的癌症样本,由于甲基化数据和基因表达数据之间负相关关系,筛选差异甲基化基因和差异表达基因之间负相关的基因,即高甲基化下调基因和低甲基化上调基因;其次,由于DNA甲基化与癌症发生密切相关 [24] ,因此保留超异常甲基化基因(|log2FC| > 2)及其对应的表达基因。合并上述两部分得到的基因构成特定肾细胞癌亚型特征基因。
3) 分类特征基因筛选。针对肾细胞癌三种亚型的全部癌症样本及两个组学的亚型特征基因数据,利用方差分析筛选保留三种癌症亚型间的显著差异(P < 0.05,FDR < 0.05)特征,并使用弹性网络 [25] [26] 对其进行分类贡献值的排名,综合排名及其对分类结果的影响确定分类特征基因。
2.3. 分类器的构建及评估
本研究使用支持向量机 [27] 模型(Support Vector Machine, SVM),但面对三分类问题,需要在外层嵌套“One-vs-Rest”(OvR)或“One-vs-One”(OvO)的结构构建三分类器 [28] 。OvR是在训练过程中取样本中N个类别某一类作为单独一类,将其余N − 1类作为另一类,但此方法可能会出现负样本的数量远远大于正样本,从而导致样本量失衡,使训练速度缓慢;OvO是在训练过程中每两个类别之间构建一个分类器,共需要构建N(N − 1)/2个分类器,但是此方法避免了正负样本不平衡的问题。因此本文在构建模型时本研究采用了OvO策略。
SVM是一种解最优分类面问题的方法。通过调用python中sklearn库完成基于支持向量机函数的分类模型的构建。本研究通过分析和测试,最终确定的SVM的主要超参数如下:
1) 核函数:目前SVM的核函数主要有线性核函数、多项式核函数、Sigmoid函数和高斯核函数(radial basis function),针对研究中样本数据线性不可分问题,本研究使用高斯核函数。
2) 惩罚系数C和核函数的系数gamma:惩罚系数是平衡向量的复杂度和误分类率两者间关系的参数;gamma是RBF核函数中表示单个样本对整个分类平面影响的参数。本研究使用网格调参的方法最终确定“C = 30”,“gamma = 3”。
3) 交叉验证cv:为了得到稳健可靠的模型,并对模型的泛化误差评估,本研究在构建模型时引入了五折交叉验证(cv = 5)。
本文通过混淆矩阵直观展示分类模型的结果,并利用准确性(Accuracy, ACC)、敏感性(Sensitivity, SEN)、精确性(Precision, PRE)和F1-score (F1)对分类结果进行评估。计算公式如下:
(2)
(3)
(4)
(5)
其中:TP表示正确分类的正样本的数量,TN表示正确分类负样本的数量,FP表示负样本被判定成正样本的数量,FN表示正样本被判定为负样本的数量。
为了更好的衡量三分类的情况,本文引入了kappa系数。它是一种基于混淆矩阵衡量分类精度的指标。通常情况下kappa取值在0~1之间,可分为五组一致性级别:0.81~1几乎完全一致,0.61~0.80高度一致性,0.41~0.60中等一致性,0.21~0.40一般一致性以及0~0.20极低一致性。计算公式如下:
(6)
其中:以三分类为例,样本总个数为n,假设每一类真实样本个数分别为a1,a2,a3;预测出来的每一类样本个数分别为b1,b2,b3。po表示总体分类的正确率;
。
2.4. 功能富集分析
分别对方法2.2中得到的DEGs和特定肾细胞癌亚型特征基因进行功能富集分析,以探索与肾细胞癌亚型相关的潜在生物学过程。GO分析包含了生物过程(Biological Processes, BP)、细胞成分(Cellular Components, CC)和分子功能(Molecular Functions, MF),本研究主要聚焦于生物过程方面。此外,使用在线工具STRING (https://string-db.org/)对肾透明细胞癌的富集结果进行了蛋白质–蛋白质相互作用网络分析;通过Cytoscape软件中“cytoHubba”筛选得到10个hub基因,分析hub基因与KIRC的免疫细胞(主要包括B细胞、CD8 + T细胞、CD4 + T细胞、巨噬细胞、中性粒细胞和树突状细胞)浸润水平的相关性并对hub基因进行了简要分析。
2.5. 本研究的流程图
本研究以RCC三种主要亚型为研究对象,结合转录组和DNA甲基化数据,实现了对这三种亚型的精准分类,具体筛选流程如图1所示。
3. 结果与讨论
3.1. 亚型特征基因筛选
将mRNA表达数据和DNA甲基化数据归一化,利用2.2给出的差异基因筛选方法分别得到了三种亚型的DEGs和DMGs,如图2所示,纵坐标轴的阈值选择的是2 (对应q value = 0.01);其中红色表示肿瘤中上调(高甲基化)基因,蓝色表示下调(低甲基化)基因。相比于KICH,KIRC和KIRP的DEGs具有更高的显著性差异;三种亚型的基因表达的差异显著性普遍高于DNA甲基化。
利用2.2给出的肾细胞癌亚型特征基因筛选方法,获得肾细胞癌亚型特征基因。将同一亚型的DEGs和DMGs匹配,匹配结果见图3。对KIRC亚型,获得了271个在DNA甲基化和基因表达数据之间具有负相关关系的匹配基因,此外筛选得到了31个超异常甲基化基因及其对应的表达基因,得到基因表达和甲基化两组学总计302 * 2个(基因表达和DNA甲基化)亚型特征基因。同理,从KIRP获得了92 * 2个亚型特征基因,包括82个匹配基因及10超异常甲基化基因;从KICH中获得了113 * 2个亚型特征基因,包括98个匹配基因及15个超异常甲基化基因。

Figure 1. Flowchart of renal cell carcinoma subtype classification
图1. 肾细胞癌亚型分类流程图
3.2. 分类特征基因筛选
利用2.2给出的分类特征基因筛选方法,获得分类特征基因。利用方差分析筛选得到了852个显著差异特征基因,利用弹性网络计算显著差异特征基因对分类的贡献,取贡献值排名前10的特征基因为候选分类特征基因。如表2所示,可以看出TOP10的特征基因中只有一个DNA甲基化基因(“M_TMEM88”),其余均是mRNA表达基因。说明相比于甲基化特征基因,mRNA表达特征基因对三个亚型分类的贡献度更大。
3.3. 分类模型构建及分类特征基因确定
基于得到的候选分类特征基因,通过构造SVM分类模型,实现了对肾细胞癌三种亚型的有监督分类。为了以最少分类特征获得较优分类结果,本研究对不同数目的候选分类特征基因分别进行建模及分类结果比较,结果见表3和图4。结果显示:当分类特征数选择6个时,准确性、敏感性和精确性分别为96.6%、94.7%和93.4%,F1-score值和Kappa系数达到了0.94,整体指标均达到了最高。因此,选择前6个基因(PRPS2, FGF7, TM4SF19, PXDNL, COQ3, SLC25A5)作为最终的分类特征基因。
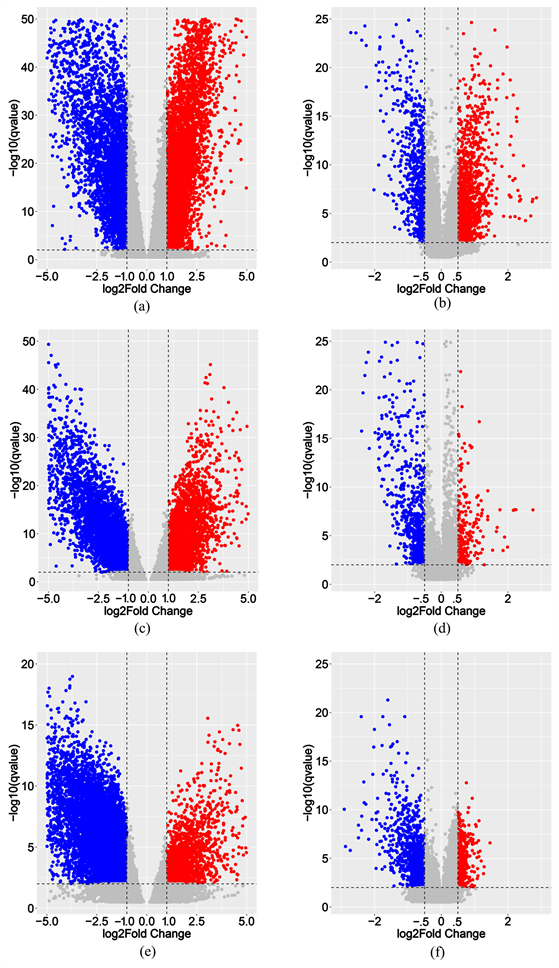 (a) and (b):KIRC的DEGs和DMGs;(c) and (d):KIRP的DEGs和DMGs;(e) and (f):KICH的DEGs和DMGs。蓝色表示下调(低甲基化)基因,红色表示上调(高甲基化)基因。
(a) and (b):KIRC的DEGs和DMGs;(c) and (d):KIRP的DEGs和DMGs;(e) and (f):KICH的DEGs和DMGs。蓝色表示下调(低甲基化)基因,红色表示上调(高甲基化)基因。
Figure 2. Differentially expressed genes and differentially methylated genes of three subtypes
图2. 三种亚型的差异表达基因和差异甲基化基因
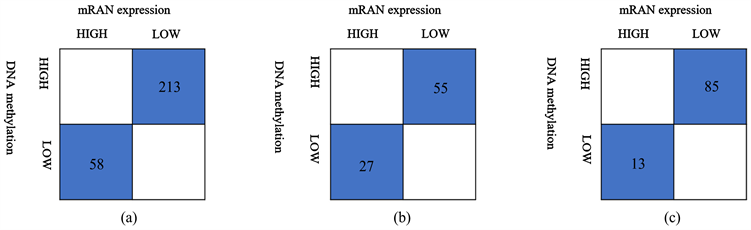
Figure 3. The matching map of gene expression and methylation of Renal Cell Carcinoma. (a) KIRC, (b) KIRP, (c) KICH
图3. 肾细胞癌基因表达与甲基化的匹配图。(a) KIRC,(b) KIRP,(c) KICH
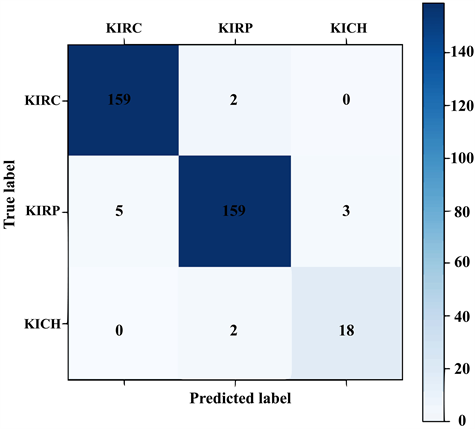
Figure 4. Classification prediction matrix of renal cell carcinoma
图4. 肾细胞癌分类混淆矩阵

Table 2. Ranking of contribution values of gene
表2. 基因贡献值排名
*“M_TMEM88”中的“M_”是DNA甲基化基因的前缀。

Table 3. Comparison of evaluation indexes of TOP gene classification
表3. TOP基因分类评价指标的比较
在构建肾细胞癌亚型分类模型时,使用五折交叉验证证明了分类模型的可靠稳定性;为进一步验证亚型分类模型的普适性,对独立检验集GSE76351的样本进行识别,模型分类正确率达到93.1%:即筛选的独立检验集中的12个肾透明细胞癌预测正确了11个,结果表明分类模型具有一定的普适性,分类特征基因选择比较合理。
分类特征基因在不同亚型中表达量分布见图5所示。从图中可以看出:COQ3和SLC25A5基因在三种亚型中表达的差异较大,FGF7、PXDNL和TM4SF19基因在KICH中的表达量较其他两种亚型高,TM4SF19在KIRC的表达量低于其他亚型,同时发现PXDML和FGF7在KIRP表达量最低。其中PRPS2、COQ3和SLC25A5是KIRC的特征基因,且在KIRC中都是下调;FGF7、TM4SF19和PXDNL是KIRP的特征基因,除了PXDNL是上调,其余两个都是下调基因。FGF7是成纤维细胞生长因子(Fibroblast Growth Factor, FGFs)家族成员,广泛参与细胞的增殖、分化、迁移、胚胎发育、血管生成以及损伤修复等生物过程 [29] 。PRPS2是一种嘌呤生物合成途径中的关键限速酶 [30] ,促进了MYC转化细胞中核苷酸生物合成的增加。PRPS2通过PRPS2内的一个专门的顺式调节元件耦合蛋白质和核苷酸的生物合成5'UTR,并且它使Myc的翻译调节能够直接增加核苷酸生物合成,与癌细胞蛋白质合成率的增加成比例 [31] 。
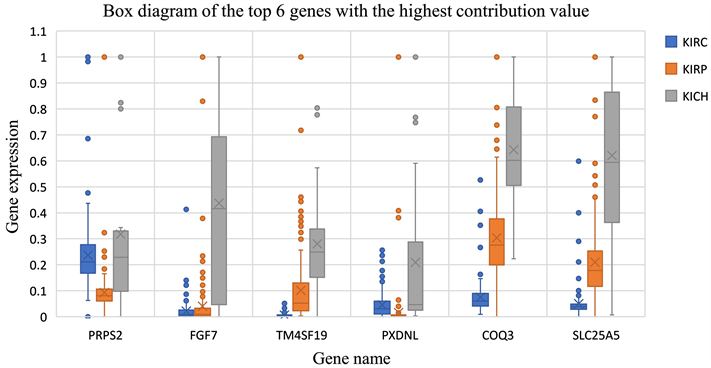
Figure 5. Box diagram of the top 6 genes with the highest contribution value
图5. 贡献值最高的前6个基因的箱式图
3.4. 肾细胞癌亚型基因特征的富集分析
首先,对特定亚型特征基因进行了GO分析,表4和表5分别是KIRC和KIRP的富集的部分结果。KIRC富集到的通路主要与钠离子的运输、钠离子运输的正调控、细胞连接组件以及免疫细胞的增殖的相关通路有关;KIRP主要与突触的组装相关通路有关。对肾细胞癌亚型DEGs进行的GO分析显示:KIRC主要与免疫系统的负调节、单核细胞的增殖与分化、白细胞增殖和黏附、淋巴细胞的增殖与分化等相关的免疫系统的相关通路有关(表S1);KIRP主要与肾脏和肾脏系统的发育、钙离子的运输和泌尿生殖系统的发育等相关通路有关(表S2);KICH主要与肾脏和肾脏系统的发育、肾单位的发展以及纤毛组织等相关通路有关(表S3)。

Table 4. Functional enrichment of KIRC preliminary signature genes
表4. KIRC初步特征基因的功能富集

Table 5. Enrichment analysis of KIRP primary signature genes
表5. KIRP初步特征基因的功能富集
对比肾细胞癌亚型特征基因和差异表达基因的富集分析,发现了KIRC在两种不同基因集的富集中都发现其与免疫系统的负调节,淋巴细胞的增殖和分化以及免疫细胞的增殖等相关通路有关(表4和表S1)。这与Xu等 [32] [33] 在其研究中发现免疫反应是透明细胞肾细胞癌致癌作用和治疗效果的重要特征相一致。此外,一些研究 [2] [34] 也已证实KIRC比KIRP和KICH增加了免疫细胞浸润基因表达特征,并且有报告 [35] 基于基因表达和回归分析等方法找到了KIRC的潜在免疫生物标志物,这些发现进一步支持了免疫反应在KIRC中的致癌作用。
免疫反应是肾透明细胞癌的致癌和治疗的重要特征。通过对肾透明细胞癌的特征基因进行蛋白质互作网络的分析(图6),得到了10个hub基因(CDH1、ERBB2、ITGAX、ESRP1、GRHL2、GZMB、CCL5、OCLN、RAB25和PRSS8),其中ITGAX、CCL5和GZMB是表达量上调基因,其余是下调基因;对这几个基因进行了免疫细胞浸润性分析,发现ITGAX和CCL5表达量的升高与这六种免疫细胞浸润均显著相关(P < 0.05) (图7),GZMB的上调与CD4 + T细胞、CD8 + T细胞和中性粒细胞的浸润性显著相关(图S1),ERBB2、GRHL2和PRSS8的下调与CD4 + T细胞的浸润性显著相关(图S1)。
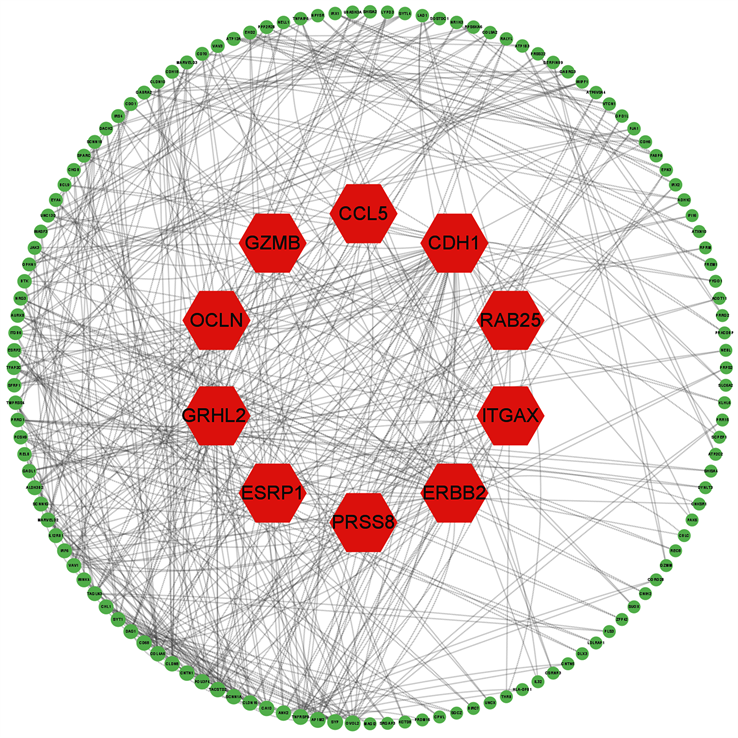 (红色基因是hub基因,外环基因按照程度中心性从大(深色)到小(浅色)的顺序排列)
(红色基因是hub基因,外环基因按照程度中心性从大(深色)到小(浅色)的顺序排列)
Figure 6. PPI network map of KIRC signature genes (Genes in red are hub genes, Outer ring genes are arranged in order of degree centrality from large (dark color) to small(light color))
图6. KIRC特征基因的PPI网络图
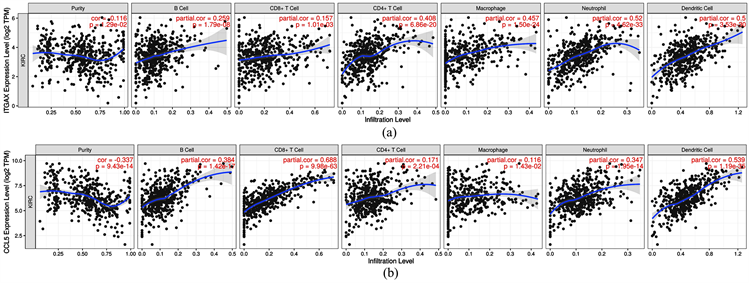
Figure 7. Immune infiltration of ITGAX and CCL5
图7. ITGAX和CCL5的免疫浸润
本研究通过构建PPI网络得到的hub基因中CCL5基因与KIRC的发生和发展显著相关。CCL5是趋化因子CC家族的一员,CC亚组的趋化因子在调节肿瘤微环境中起重要作用 [36] 。其主要表达于T细胞、巨噬细胞和部分肿瘤细胞 [37] 。CCL5通过募集T细胞和巨噬细胞等间接促进肿瘤的增殖 [36] 。本研究发现CCL5基因的上调与CD8 + T细胞和树突状细胞的免疫浸润强相关,白等 [38] 的研究也证实了CCL5的上调在KIRC中与CD4 + T细胞、CD8 + T细胞、Tregs细胞和静息树突状细胞等有显著关系。有研究报告证实了CCL5基因可以作为KIRC患者的潜在治疗靶点 [38] [39] 。
4. 结论
本研究旨在从转录组数据和DNA甲基化数据两个组学层面对早期的KIRC、KIRP和KICH进行特征基因筛选及有监督的分类,寻找肾细胞癌亚型之间的不同分子特征。利用支持向量机、弹性网络和交叉验证等方法构建了三分类的分类器,筛选得到了贡献值最大的6个特征基因作为分类特征基因,准确性达到了96.6%,精确性和敏感性分别是93.4%、94.7%,F1-score值和Kappa系数都达到了0.94,说明了分类器的可靠性。同时,独立检验集正确率达到了93.1%。需要说明的是,通过检索GEO数据库,得到的独立检验集仅包含了KIRC一种亚型,因此对KIRP和KICH需要在未来满足条件时加以验证。
对各个亚型中的特征基因使用GO分析进行了通路富集分析,发现KIRC主要与免疫细胞的增殖等相关通路有关,与其不同的是,KIRP和KICH都和肾脏与肾脏系统的发育有关。此外,KIRP还与钙离子的运输和泌尿生殖系统的发育有关,而KICH则与纤毛组织相关通路有关。本研究也发现CCL5的上调与免疫之间存在显著相关性,这也为精准免疫治疗提供了理论基础。
致谢
感谢国家自然科学基金和重点研究开发项目的支持;此外,特别感谢TCGA数据库提供的原始数据。最后,特别感谢编辑和各位匿名审稿人对本文的支持和帮助。
基金项目
本研究由国家自然科学基金(61931013)和重点研究开发项目(2017YFC011104)资助。
附录

Table S1. Functional enrichment of KIRC differentially expressed genes
表S1. KIRC差异表达基因的功能富集

Table S2. Functional enrichment of KIRP differentially expressed genes
表S2. KIRP差异表达基因的功能富集

Table S3. Functional enrichment of KICH differentially expressed genes
表S3. KICH差异表达基因的功能富集
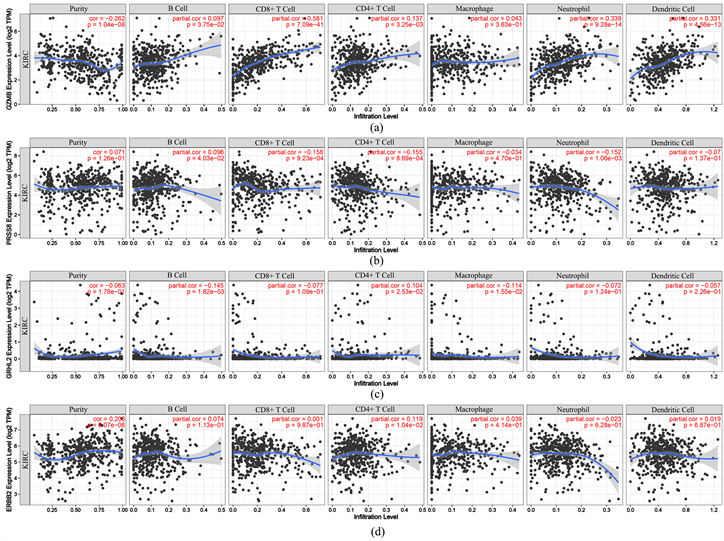 (a)~(d)依次是GZMB、PRSS8、GRHL2和ERBB2
(a)~(d)依次是GZMB、PRSS8、GRHL2和ERBB2
Figure S1. Immune infiltration of hub gene
图S1. hub基因的免疫浸润
NOTES
*通讯作者。