1. 引言
种群生态数学模型可以很好地刻画种群或种群间的变化规律,如预测人口变化趋势或传染病的传播和爆发。其中较为知名的有Logistic模型、Gompertz模型和Richards模型等。对于前两种模型,它们的生长曲线具有特定的拐点,只能准确描绘一种特定形状的S型曲线。因此对给定生长过程建模时,在生长方程的比较和筛选方面需要做大量工作。Richards在1931年研究流体 [1] 通过多孔介质中毛细管传导作用时建立了Richards方程,情况才得以改观。Richards认为Bertalanffy方程中的异速生长常数限定为2/3太严格,这一限制使得该模型不能应用到大部分生物生长上,将其设为一般化参数m,得到一个含有4个参数的非线性S型增长模型。这种模型对生长过程描述的准确性达到了前所未有的水平 [2]。
邢黎峰 [3] 在研究Richards方程时发现,当异速生长参数m变化时,不仅包括了Brody、Logistic等生长方程,而且包含了它们的中间过渡形式和更为广义的形式,并得出Richards模型比众多生长模型更为准确的结论。对种群生态数学模型的参数估计是模型得以应用的前提,因而对该类模型的参数估计受到许多学者的关注和研究。吴新元 [4] 指出常用于Logistic方程拟合的方法有三点法、麦考特法和枚举优选法,但这些方法在应用上都有其局限性;魏冠军 [5] 等将参数视为随机变量,利用Bayes理论进行参数估计,同遗传算法和最小二乘法进行对比,得出前者的估计效果优于后两者;徐文科 [6] 提出双向差分拟合方法和双向差分广义加权最小二乘法 [7],并对初始预报值进行了改进,通过与优选法、麦考特法等方法比较,结果表明精度有所提高;刘洋 [8] 利用双向差分广义加权最小二乘法对Gompertz增长曲线方程的参数估计进行研究,并在SARS的疫情实例中得到了较好的拟合效果;乔钰 [9] 利用线性最小二乘法和双向差分加权最小二乘法对Richards增长曲线的参数进行估计,并对具有相关关系的Richards方程组建立半相依回归模型 [10]。
综上所述,对于一系列生物生长方程,已有许多的数值方法和统计模型对参数进行估计,在实际应用中,选择合适的模型和参数估计方法就显得尤为重要。本文研究了Logistic和Richards两类种群生长模型,利用线性最小二乘法对参数进行估计,最后结合湖南省的新冠疫情实例说明增长曲线方程的适用性及参数估计的有效性。
2. Logistic生长模型模拟新冠疫情
2.1. Logistic方程的产生
种群动态研究一直是生态学研究的核心问题之一。Logistic方程指出了有限空间种群增长的基本规律:当一个物种迁入到一个新生态系统中后,其数量会发生变化。当物种的起始数量小于环境最大容纳量时,数量会增长;若此生态系统中食物、空间等资源有限(非理想环境),则种群数量呈S型增长。Logistic方程是研究种群动态的重要工具,其微分形式为:
(1)
其中N为种群个体总数,t为时间,r为种群增长潜力指数,
表示环境最大容纳量,
为
时刻的种群数量。利用分离变量法并代入初值条件
,解得:
(2)
Logistic方程的性质:
1) 由式(1)知,
是关于t的递增函数;
2) 对式(1)求导并结合式(2),得到拐点坐标
(3)
3) 存在两条渐近线,当
时,
;当
时,
。
2.2. 生物生长曲线的一般特征
生物体在一个完整的生长过程中其生长速度通常具有慢–快–慢的变化特征。它的累积生长量最初较小,随时间的延长逐渐增大,最终稳定在一个饱和值附近。这一过程若用曲线来表示,将是一条拉长的S型曲线(见图1)。
COVID-19累计病人数具有慢–快–慢的变化特征,且初值较小,随着时间的延长逐渐增大,最终稳定在一个饱和值上。Logistic和Richards方程能够很好地描述其初始生长阶段、快速增长阶段和稳定生长阶段的累计病人数的变化情况。而任意两个生长过程都不会完全相同,它们的差异受生物物种、生态环境、观测指标和时间序列等诸多因素的影响 [11]。
2.3. Logistic增长曲线的参数估计
由式(2),Logistic方程的一般形式为
,取对数得:
. (4)
对式(4)求导得:
. (5)
由式(4)、(5)可得:
. (6)
对式(6)取对数得:
.(7)
进一步令
,
,上式可表为
. (8)
再用向后差分
近似微分,设误差为
,得差分方程
(9)
令
,
,
,
,
将式(9)写成矩阵形式即
,进而得到
的最小二乘估计
. (10)
对于式(10),已求得B、K的估计值,只需将所有观测值代入求得A的一系列观测值,并剔除异常点,取均值即得A的估计值。
至此,Logistic方程的参数估计值全部得到。
对2020年湖南省的疫情历史数据进行复现拟合,利用上述方法得到一组初值
,进而利用LM非线性拟合方法得到参数,最后得到的Logistic方程为:
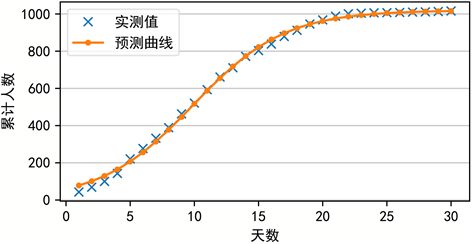
Figure 2. Fitting results of logistic equation of “S” growth curve
图2. “S”型生长曲线logistic方程拟合结果
Logistic方程的拟合结果与实测值的均方根误差(RMSE)为15.3300,由图2可以看出,Logistic方程对前期数据的拟合效果较差,通过查找文献,我们发现了一类适应度更加广阔的生长方程,即Richards方程。
3. Richards方程模型
3.1. Richards方程的产生
Richards认为Bertalanffy方程中的异速生长常数限定为2/3太严格,应将其设为一般化参数
,它的取值与生物物种和环境条件有关,并提出假设:
(11)
这是m次的Bernoulli微分方程,V是种群个体数,
、
和m为异速增长参数。令
,化简得非齐次微分方程:
. (12)
利用通解公式解得:
(13)
代入初值条件
得到特解 [10]:
(14)
式中,
,
,
。通常把式(14)叫做Richards增长曲线方程,
这里P、Q、H、m为四个待估参数,分别含有不同的生物学意义。P是研究对象的生物学上限,Q是生长初始值参数,H是生长速率参数,m是异速生长参数,其决定着曲线图像的走势以及拐点的位置。
3.2. Richards生长方程的性质与可塑性
在文献 [12] 中,Fengri Li研究了Richards方程各参数不同取值的情况,并依据解结构和生物学解释划分为8种类型。因为COVID-19疫情的变化特点,在本文我们只考虑P、
,
且
的情形,此时Logistic方程是
时的特例。
1) 对式(14)求导知,
是关于t的递增函数;
2) 对式(14)求导并令二阶导数为0,得到拐点,其坐标为
(15)
在该点生物增长率达到峰值
,称之为拐点;
3) 存在两条渐近线,
时,
;
时,
。
Richards方程具有很强的可塑性,当方程中的参数m连续取不同的值时,曲线形状会不断发生变化(见图3)。由(15)式可知,参数m的取值影响拐点的横、纵坐标大小,也即影响拐点的相对位置。当m取值较小时,“S”曲线的拐点位置偏低,该曲线的下半臂较短,上半臂更长;反之,当m取值较大值时,“S”曲线的拐点位置偏高,该曲线的下半臂较长,上半臂更短。特别地,通过变换Richards增长曲线方程中参数m的值,可以分别得到许多著名且应用广泛的方程。例如令参数m为0、2/3、2,分别得到Mitscherlich方程、Bertalanffy方程和Logistic方程。

Figure 3. Plasticity of Richards equation
图3. Richards方程的可塑性
3.3. Richards增长曲线的参数估计
同上,对式(14)取对数求导得:
.(16)
进一步令
,
,
,
上式可表为:
.(17)
再用向后差分
近似微分,设误差为
,得差分方程
(18)
令
,
,
,
,
将式(18)写成矩阵形式即
,假设
,
,进而得到
的最小二乘估计
. (19)
对于式(19),已求得
、H、C的估计值,将所有观测值代入求得P的一系列观测值,剔除异常点,取均值得至此,Richards方程的参数估计值全部得到。
利用上述方法得到一组初值
,进而利用LM非线性拟合方法得到参数,
最后得到的Richards方程
。
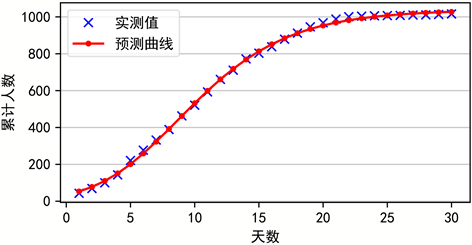
Figure 4. Fitting results of Richards equation
图4. Richards方程拟合结果
Richards方程的拟合结果与实测值的RMSE为10.6444,由图4可以看出,Richards方程较Logistic的结果大大改进了,但后期的值存在波动,所以我们利用残差倒数对两者的结果结合进行预测。
4. 组合预测方法
采用下式所示的误差倒数法对两个模型的结果进行加权组合,其中
为权值系数,
和
分别为Logistic和Richards的预测值。权值系数中
、
分别为两者的误差。
(20)
组合模型的RMSE为9.8681,从下图5可以看出对两类曲线方程的拟合效果都较好,使用残差倒数法对结果进行校正,该方法对误差小的模型会赋予较大的权值系数,从而使整个组合模型误差减小,达到提升整体预测精度的效果。
5. 结论
对于具有微分方程形式的生物数学模型,本文利用线性最小二乘法,从定量的角度对两类增长曲线方程的参数进行估计,并将其应用到湖南省的新冠疫情实例中。结果表明利用增长曲线模型预测传染病的传染增长规律具有一定的可行性,Richards方程具有更为广泛的适应性、良好的解析性和预测性。这种方法易于编程实现,不仅适合于Logistic和Richards曲线方程的拟合,而且适用于其他非线性曲线的拟合,因此在生物学中有广泛的应用。
基金项目
湖南省大学生创新创业训练项目(NO.202010536019)。
NOTES
*通讯作者。