1. 介绍
近年来,多药联用正成为治疗复杂疾病(如糖尿病和癌症)的一种有前途的策略。当两种或两种以上药物同时服用时,可能会引发意想不到的副作用、不良反应、甚至严重的毒性反应。多种药物在治疗过程中引发的药理作用称为药物–药物相互作用(drug-drug interactions, DDIs)。因此,重要的是确定DDIs并揭示其对多药安全的潜在机制。然而,在大量药物对的检测中发现DDIs既费时又昂贵,所以很多计算方法被提出用于预测DDIs。在过去的十年中,这种通过实验确定DDIs促进了计算方法的应用,特别是通过深度学习的计算方法以发现潜在的DDIs。各种基于深度学习的计算方法伴随着效率高、成本低的优点已经被证明是一种很有前途的方法,可以提供DDIs的初步筛选。在本篇文章中,我们着重介绍近年来基于图卷积网络的药物相互作用预测及其最新研究进展,这是其发挥生物医药学功能的一个重要作用机制。
2. 通用数据集
目前,药物相互作用的可靠的数据分布在多个来源上。已知的主流的药物相互作用数据库是DrugBank数据库,此外还有PharmGKB、KEGG Drug、OFFSIDES和BioSNAP-DDIs。DrugBank数据库是一个权威标准DDIs数据库,涵盖294,980个DDIs (阳性),跨2,286个批准药物。它结合了详细的药物相关信息,包括化学、药理、药物数据和全面的药物靶标信息。PharmGKB数据库包含12,664种药物条目,包括2,588种已批准的小分子药物、1,287种已批准的生物技术药物、130种营养药物和超过6,305种实验药物。PharmGKB数据库是一个存储基因组、分子和细胞表型数据的仓库。它还包含临床信息和遗传变异对药物反应的影响。PharmGKB包含基因、疾病、药物和通路相关数据,以及影响药物代谢的470个遗传变异的详细信息。KEGGDrug数据库包含代谢途径,链接到代谢产物和蛋白质/酶信息。截至2019年5月,KEGGDrug数据库拥有药品相关信息10,979条,DDIs关系501,689条。OFFSIDES数据库包含了基于PharmGKB的不良事件报告中挖掘出来的药物效应,该数据库报告了438,802种药物副作用。最后,BioSNAP-DDIs 数据库包含从药物标签和生物医学文献中提取的1,514种药物之间的一共48,514次相互作用。
3. 图卷积网络模型介绍
近年来,卷积网络快速发展 [1] [2],并因其建模的高效性引起大家重视。卷积网络给图像处理 [3],图像识别 [4],机器翻译 [5],语音识别 [6] 带来了很大的突破。传统卷积神经网络局限于对欧氏空间数据进行建模,而图卷积操作利用图表示对非欧氏空间数据进行处理,使卷积操作更加适用于药物数据结构。图卷积操作主要包括基于空域以及频域的图卷积类。空域图卷积将图卷积操作定义为图中相邻节点之间的特征信息的聚合,基于频域的图卷积则利用图信号处理(graph signal processing),引入滤波器实现频域图卷积的推导。无论是空域图卷积还是频域图卷积,本质上都是对图拓扑结构的建模利用,即空间特征的提取 [7]。
首先给出常见通用的符号定义,如表1所示。

Table 1. Common symbol definitions
表1. 常用的符号定义
一般地,
用于表示常用的无向带权图。A表示图的邻接矩阵(adjacency matrix),定义节点之间的相互连通关系。H为关联矩阵(incidence matrix),用于定义图中边与节点之间的连接,当
,
,其他情况下,
,对于图中的节点,其度矩阵Dv中元素定义为连接到该节点所有边的权重之和 [7]。即
相似地,De中元素定义为连接到该边的节点数量之和,即
GCN的基本思路:对于每个节点,我们从它的所有邻居节点处获取其特征信息,这当然也包括它本身的特征。将从邻居节点获得的特征信息和本身的特征信息融合,输入到average()函数中,得到一个平均值。同样的,我们再对其他节点进行类似的操作。最后,我们将这些计算得到的所有平均值输入到神经网络中。
4. 预测模型
在2022年,Wang等人提出一种多核图卷积网络(GCNMK)来预测潜在的DDIs [8]。该模型将DDIs图分为两大类,其中一类是“增加”相关的图GI,例如,药物A可能会增加药物B的活性。另外一类与“减少”有关的图GD,例如,当药物A与药物B结合时药物A的代谢会降低。然后将两类DDIs图GD和图GI分别与药物特征矩阵整合,分别输入两个GCN块。GCN层有两层,第L1层有两个并列的GCN块,第L2层有一个GCN块。第L2层的GCN块被用于去整合第一层的两个GCN块的输出,第L2层的GCN块输出的是最终DDIs特征矩阵,是药物的低维表示向量。在每个块中采用的是输入的线性组合的加法程序。最后将DDIs特征矩阵输入三个完全连通层来预测潜在的DDIs。在隐藏层的激活函数用的是RELU,输出层用的是Sigmoid,损失函数用的是cross-entropy和L2-regularization。L2-regularization用来解决过拟合问题,损失函数越小,预测的准确性越高。实验调整了参数去实现最优的性能,最后计算最优的参数为学习率lr = 0.002,L2-regularizatio系数λ = 0.0003。实验最后通过五倍交叉验证,证明了该模型确实可以有效进行药物和药物相互作用关系预测。具体流程如图1所示。
在2022年,Feng等人提出一种用于药物-药物相互作用多标签预测的深度图卷积网络框架(称为deepMDDIs)来预测DDIs及其类型 [9]。首先将多关系网络DDIs分解成m个子网络,用m个邻接矩阵表示子网络去作为编码器的输入。deepMDDIs利用相似正则化约束,用深度图卷积网络设计了一种编码器来捕获DDIs网络的拓扑特征。编码器通过捕捉药物的复杂拓扑性质,构造一个p层关系GCN (R-GCN),将多关系DDIs网络中的药物编码为嵌入向量,并且从第2个隐藏层添加一个残留策略到最后一个隐藏层。同时,采用药物相似度矩阵约束嵌入空间的相似药物,使其尽可能接近。使一个节点在网络中聚合其一阶邻居节点以及自身的特征,以重新更新自己特征。然后,将所有更新的特征累加起来,通过一个ReLU激活函数,生成最终的嵌入。整个DDIs网络由一个p层的R-GCN进行传播,以捕获其p阶邻居的信息。之后采用类张量解码器对单次交互和多次交互进行统一建模,以确定未标记的类型特异性药物对是否产生一种或多种药理作用。译码器是一种类张量分解矩阵运算,将嵌入的特征矩阵、特定类型的特征重要性矩阵和平均的特征关联矩阵相结合,重构出多元关系的DDIs网络。
通过实验结果表明,deepMDDIs可以有效区分DDIs网络中一个未标记的类型特异性药物对是否导致一种或多种药理作用,也可以预测DDIs网络中已知药物与网络外新药之间的潜在DDIs。通过与目前最先进的基于深度学习的方法在DDIs多分类任务中的比较,在单倍DDIs和多倍DDIs预测中均取得了令人鼓舞的效果。证明了该算法的优越性。具体流程如图2所示。
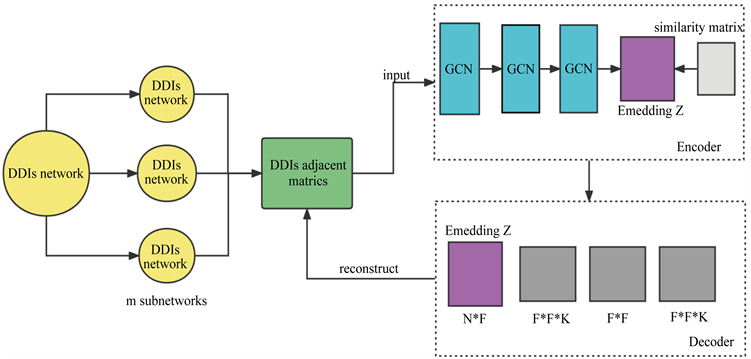
Figure 2. Flow chart of deepMDDIs model
图2. deepMDDIs模型流程图
在2019年,Karim等人提出了一种基于知识图形嵌入和和Conv-LSTM网络的药物–药物相互作用预测 [10]。使用来自DrugBank、KEGG Drug、PharmGKB和OFFSIDES的数据创建了一个包含2,898,937个药物–药物交互对的数据集,由于该背景数据的格式不能作为典型分类器的直接输入,因此应用了几种节点嵌入技术,使用RDF2Vec、SimpleIE、TransE、KGloVe、CrossE和PBG进行KG嵌入,为KG中的每个节点创建一个密集的向量表示,通过执行KG嵌入程序将信息从图编码成密集向量。KG嵌入包括三个步骤:表示实体和关系、定义评分函数以及学习实体和关系表示。之后将KG嵌入后的结果输入由CNN和LSTM层构造的Conv-LSTM网络中,CNN使用卷积滤波器来捕捉药物特征中的局部关系值,而LSTM网络可以从CNN提取的特征中携带所有关系。
网络中第一层是嵌入层,它将药物样本作为一个序列映射到一个实向量域。然后将大小为100 × 300的嵌入矩阵送入一维卷积层,该层有100个滤波器,核大小为4。然后将每个卷积层的输出传递给Dropout层进行正则化学习以避免过拟合。直观上可以被认为是迫使分类器不依赖于任何微不足道的单个药物特征。然后卷积层将输入特征空间卷积为100 × 100表示,由沿着嵌入维的池大小为4的1维最大池化层(MPL)进行下一步采样,产生形状为25 × 100的输出。其中,25个维度中的每一个都可以被认为是一个提取的特征。MPL通过在每个时间步长维度上取最大值来扁平化输出空间,从而产生一个1 × 100的载体。LSTM层将扁平的特征向量的维度视为时间步长,并且每个时间步长输出100个隐藏的单元。第二个LSTM层发出一个输出H,H被重塑成一个特征序列,并被送入完全连接的层,以预测下一步的DDIs。然后使用一个全局MPL,在经过另一个Dropout后,最具影响力的特征被送入一个全连接层,最后送入SoftMax层,该层生成类的概率分布。此外,我们在每个Conv、LSTM和稠密层中引入高斯噪声,以提高模型的泛化能力。在5倍交叉验证测试中,分别获得了高达0.94,0.92,0.80的AUPR,F1-score和MCC,显示在预测潜在的DDIs方面有很高的可信度。具体流程如图3所示。
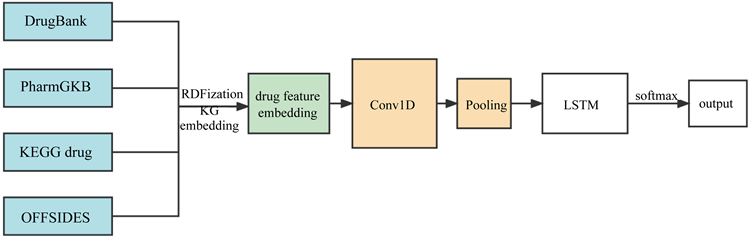
Figure 3. Flow chart of Conv-LSTM model
图3. Conv-LSTM模型流程图
在2019年,Zhong等人提出一个增大图形卷积神经网络模型预测DDIs [11]。该方法将图卷积神经网络与注意力池化网络相结合,提取药物对之间的结构关系并进行DDIs预测。具体地说,我们使用RDKit将药物SMILES字符串转换为图形拓扑。然后应用权重共享图卷积神经网络(GCNN)生成图表示。在这样的GCNN层之后,我们嵌入了一个注意力池化网络来计算两种药物如何相互作用。在两个输入对之间建立一个紧密联系的双向注意力机制,算法通过应用CNN或者LSTM去计算一个软结合G = tanh (QTWA),表示两者之间的交互相关分数。最后通过一个SoftMax规范化操作,应用行和列的最大池化,被分隔的两边整合了共同的信息,即图中的hA,hB。然后将hA和hB连接起来,输入完全连接的层。最后,一个简单的Sigmod函数确定最终的分类。最后实验通过五折交叉验证显示,ROC为0.988,F1-score为0.956,AUPR为0.986。此外,该模型还可以通过不同颜色的原子来揭示两种DDIs药物在结构上的相互作用。这可能有助于药物发现过程中的药物设计。具体流程如图4所示。
在2015年,Kc等人提出了一种名为高阶图卷积网络(HOGCN)来聚合来自高阶邻域的信息,去用于生物医学实体对之间的交互预测 [12]。具体来说,HOGCN收集不同距离上邻居的特征表示,并学习它们的线性混合,以获得有用的生物医学实体的代表。HOGCN使用交互网络结构G或特征X去学习每个生物医学实体的表示,以交互网络G为输入,重构交互网络中的边的高阶图卷积网络(HOGCN)。HOGCN主要有两个组成部分:编码器:一种高阶图卷积编码器,它在交互图G上运行,并通过在不同距离上聚集来自k阶邻域的特征来产生生物医学实体的表示。该层可以学习混合不同距离的邻居的特征表示,以进行交互预测。解码器:利用HOGC层产生的特征表示重建输入交互网络G中的边缘。采用双线性层融合生物医学实体vi和vj,并且学习边eij。边eij被输入到两层的全连接神经网络中去预测实体vi和vj相互作用的可能性。编码器–解码器的方法使HOGCN成为一个端到端可训练的交互模型。在药物–药物相互作用上表明,HOGCN具有更准确和标准的预测。在考虑不同距离下邻居特征表示的情况下,HOGCN在有噪声、稀疏的交互网络上表现良好。此外,通过基于此文献的案例研究,验证了一些新颖的交互预测。具体流程如图5所示。
5. 结果比较
表2为五种基于图卷积网络模型在药物相互作用关系预测的比较。

Table 2. Comparison of experimental results of five models
表2. 五种模型实验表现结果对比
6. 结论与展望
通过实验结果证明,基于图卷积网络预测药物之间的相互作用取得了有效的结果,证明了预测确实是很有必要的,这样能节省大量的时间和物质成本。但即便如此,模型也存在局限性。在GCNMK模型中构造DDIs图和生成药物集时,实验中的药物至少有一个DDIs。但这里将不含任何已知DDIs的药物移除。因此,模型不能在分离的药物中识别DDIs。未来的研究有几个方向,在GCNMK的DDIs图中,边属于同一类型。我们可以将其应用于任何异质网络,比如药物疾病网络。药物–疾病关联的描述包括两种类型:疗法和标记物/机制,这可能有助于采用GCN模型。未来的另一个方向是区分更多类型的DDIs。根据它们的功能,每种类型的DDIs都可以被用来构造一个图核,并且该模型有潜力来识别预测的DDIs的特定类型。在另外一个HOGCN模型中只考虑已知的交互来标记潜在的交互。生物医学信息的其他来源,如生物医学实体的各种物理化学和生物特性,可以提供关于交互作用的额外信息,可以计划研究将这些特征集成到模型中。由于HOGCN模型在不同的距离上聚集邻域信息,并可以标记新的交互作用,因此,以输入交互网络G的一个小子图的形式为预测提供可说明的解释将是有趣的。
在这篇综述中,我们全面回顾了近些年来使用图卷积网络来进行药物之间相互作用关系预测的文章,这些文章中的模型已经成功预测了药物之间的相互关联,并取得了优良结果。此外,在文章最后我们也总结了上面模型中存在的缺点,并为未来从事此研究方向的学者提出了一些建议,希望这些能帮助改进现有的方法来取得更优的结果。