1. 引言
2019年12月出现的新型冠状病毒(Severe Acute Respiratory Syndrome Coronavirus 2,SARS-CoV-2,简称新冠病毒)感染引发的新型冠状病毒肺炎(Coronavirus Disease 2019,COVID-19,简称新冠肺炎),已在全球范围内肆虐传播两年多,对全球公共卫生及人类生命健康造成巨大威胁 [1]。尽管全球各地已采取各种防疫措施,但每日新增确诊病例数及死亡病例数仍在快速增加。据世界卫生组织(World Health Organization,简称WHO)实时统计数据,截至北京时间2022年5月30日,全球累计新冠肺炎确诊病例252,467,084例,累计死亡病例6,285,171例 [2]。新冠病毒在大规模传播的同时不断发生变异进化,产生基本传播系数(Basic reproductive number,简称R0)或致病性更强的变异株,如Alpha变异株、Beta变异株、Gamma变异株、Delta变异株,对新冠防控和疫苗开发造成巨大影响 [3]。2021年11月9日于南非首次检测到的Omicron (B.1.1.529)变异株,在短短一周时间即成为南非豪登省主流新冠变异株,并于同月26日被WHO定性为最高级别的“关切变异株” [4]。目前全球有150多个国家及地区检测出Omicon变异株 [5],该变异株已成为全球优势变异株 [6]。
作为Omicron变异株发现前传染性最高的变异株,Delta变异株的R0值约为5~8 [7],而Omicron变异株的R0数值在全球范围内可能高达10 [8]。流行病学追踪显示,Omicron变异株比Delta变异株更易于传播,其传播速度亦高于Delta变异株 [9]。病毒基因测序显示,2022年2月底开始爆发,导致感染人数超过52万的上海市新冠疫情,主要是Omicron变异株传播造成 [10]。新冠病毒主要有四种结构蛋白,分别为表面刺突蛋白(S蛋白)、膜蛋白(M蛋白)、核衣壳蛋白(N蛋白)及包膜蛋白(E蛋白) [11]。其中S蛋白具有识别人体细胞表面血管紧张素转化酶II (简称ACE2蛋白)受体蛋白,并与之特异性结合,从而介导细胞膜融合的功能,能够影响冠状病毒感染宿主细胞、特异性识别特定宿主的能力 [12]。S蛋白与ACE2受体蛋白的亲和力决定了新冠病毒感染能力,因此S蛋白的氨基酸位点变异与新冠病毒传染性密切相关 [13]。Omicron变异株是当前R0数值最高的新冠病毒变异株,其S蛋白的变异位点及其特征研究具有重要意义。本文对新冠病毒Omicron变异株的S蛋白变异位点进行分析,与其他冠状病毒的S蛋白进行多序列比对及构建系统进化树,以便加深人们对Omicron变异株S蛋白功能的理解,并对后续研究、筛选靶向S蛋白的新型抗病毒药物提供理论依据。
2. 材料与方法
2.1. Omicron变异株的S蛋白序列获取
从NCBI (https://www.ncbi.nlm.nih.gov)下载原始新冠病毒Wuhan-Hu-1 (NC_045512.2) S蛋白氨基酸序列及核苷酸序列。以来源于印度的Omicron变异单株(EPI_ISL_10204983)为例,通过GISAID (https://www.gisaid.org/)数据库下载该测序株的基因组序列。通过线上网站EMBOSS Needle (https://www.ebi.ac.uk/Tools/psa/emboss_needle/)获取其S蛋白核苷酸序列,通过线上网站Expasy (https://web.expasy.org/translate/)将其转换为氨基酸序列。
2.2. Omicron变异株S蛋白变异位点预测
通过线上网站Nextclade (https://clades.nextstrain.org)对Omicron变异株S蛋白进行变异位点分析,并利用在线工具Phyre2对S蛋白三级结构进行模拟构建,使用PyMOL对结果进行可视化操作,标记主要突变位点的所在空间位置。
2.3. 不同新冠病毒变异株的多序列比对
从NGDC (https://ngdc.cncb.ac.cn/ncov/)及GISAID数据库中,下载WHO认可的5个关注VOC变种(Alpha, Beta, Gamma, Delta, Omicron)和7个需留意的VOI变种(Epsilon, Zeta, Eta, Theta, Lota, Kappa, Lambda)的全基因组序列,按照上述2.1方法获取新冠病毒变异株的S蛋白序列。将12种新冠病毒变异株和Wuhan-Hu-1、猫、狗、猴、马来菊头蝠等冠状病毒的S蛋白序列,利用MEGA11软件进行多序列比对。
2.4. 不同冠状病毒的系统进化树构建
从NCBI网站中获取水貂(Neovison vison)、孟加拉虎(Panthera tigris)、亚洲狮(Panthera leo persica)、白鼬(Mustela erminea)、马来亚虎(Panthera tigris jacksoni)、白尾鹿(Odocoileus virginianus)、骆驼(Camelus)、蝙蝠(Chiroptera)、猪(Sus)、牛(Bovini)、麻雀(Passer)、兔(Rabbit)等13种动物的新冠病毒S蛋白序列,并与前文的Alpha、Beta、Gamma、Delta、Omicron 5种新冠病毒变异株,以及猫(Felinae)、狗(Canis lupus familiaris)、猴(Primates)、马来菊头蝠(Rhinolophus malayanus)等冠状病毒和Wuhan-Hu-1共24种冠状病毒的S蛋白序列,利用MEGA11软件的邻接法构建系统进化树。
3. 结果与分析
3.1. Omicron变异株S蛋白变异位点分析
由线上网站Nextclade比对分析显示,所选Omicron变异株S蛋白与最早发现的Wuhan-Hu-1标准毒
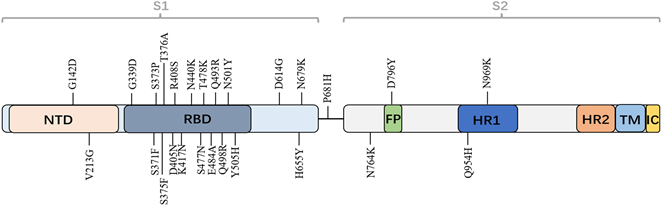
Figure 1. The S protein mutation site of the SARS-COV-2 Omicron variant
图1. 新冠病毒Omicron变异株的S蛋白变异位点
株S蛋白进行比对,共有26个位点发生突变,分别为G142D、V213G、G339D、S371F、S373P、S375F、T376A、D405N、R408、K417N、N440K、S477N、T478K、E484A、Q493R、Q498R、N501Y、Y505H、D614G、H655Y、N679K、P681H、N764K、D796Y、Q954H、N969K (见图1)。其中与人ACE2受体结合特异性和亲和力相关的RBD区域,共发现16个氨基酸突变位点,分别为G339D、S371F、S373P、S375F、T376A、D405N、R408S、K417N、N440K、S477N、T478K、E484A、Q493R、Q498R、N501Y、Y505H。
3.2. Omicron变异株S蛋白三级结构分析
利用在线工具Phyre2对Omicron变异株S蛋白的三级结构进行模型构建,在获得的结果中下载pbd文件,使用PyMOL进行可视化分析并标记出RBD区域的主要突变位点,相关结果见图2。图2(a)显示出S1和S2蛋白的三维结构,以及S1蛋白的RBD区域(灰色)和NTD区域(麦色)。图2(b)显示,在Omicron变异株的S蛋白RBD区域的16个氨基酸变异位点中,K417N、N440K、T478K、E484A、Q493R、Q498R、N501Y、Y505H共8个氨基酸位点与人受体蛋白ACE2结合域相互作用。因此上述位点变异可能增强新冠病毒与人ACE2的结合能力,进而提高病毒感染力。
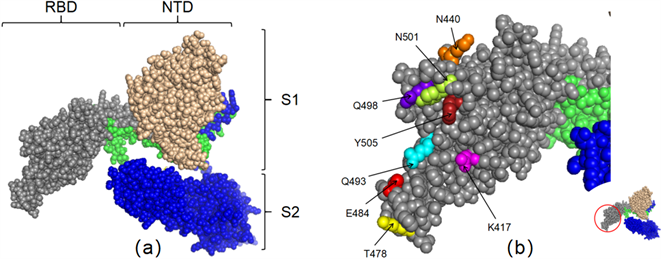
Figure 2. The tertiary structure of S protein and RBD mutation position with SARS-COV-2 Omicron variant; (a) The tertiary structure model of Omicron variant’s S protein; (b) RBD mutation position of SARS-COV-2 Omicron variant
图2. Omicron 变异株的S蛋白三级结构和RBD区域重要突变位点示意图;(a) Omicron变异株的S蛋白三级结构;(b) S蛋白RBD区域重要突变位点标记示意图
3.3. 不同新冠病毒变异株的多序列比对
对5个关注VOC变种,7个需留意VOI变种,以及4种动物冠状病毒的S蛋白进行多序列比对,部分比对集合图见图3。结果显示,17种冠状病毒S蛋白中,完全一致的氨基酸残基有1187个,占93.17%,表明S蛋白的氨基酸序列保守性较高。其中与Omicron变异株S蛋白序列一致性最高的是Zeta变异株,两者序列一致性高达98.11%;其次是猫、猴的新冠病毒S蛋白序列,序列一致性皆为98.04%;与新冠病毒Lambda变异株相似性最低,其序列一致性仅为97.01%。
3.4. 不同冠状病毒的S蛋白系统进化分析
对17种不同动物冠状病毒、5种新冠病毒变异株以及SARS病毒共24种冠状病毒的S蛋白进行多序列比对,并采用邻接法构建系统进化树,分析结果见图4。结果显示,骆驼、麻雀、猪、蝙蝠、牛、兔以及人类SARS等冠状病毒聚为一类,自展值为100,说明它们在进化上相距较近,可能来源于同一祖先。人类Omicron变异株(EPI_ISL_10204983)、Gamma变异株(OU066503.1)以及Beta变异株(MZ314998.1)

图3 不同冠状病毒的S蛋白多序列比对集合图
与上述冠状病毒遗传距离较近,进而可推测这些冠状病毒在进化上方向一致。
聚类分析显示,白鼬、亚洲狮、狮子、白尾鹿、马来菊头蝠的冠状病毒以及人类新冠病毒Delta变异株(Z706155.1)聚为一支,马来亚虎、虎、猫、猴的冠状病毒以及最早发现的新冠病毒Wuhan-Hu-1 (NC_045512.2)聚为一支,上述结果说明冠状病毒进化可能与宿主物种无关,冠状病毒S基因的RNA序列变异具有随机性和多样化,导致其进化与宿主物种及发生时间等没有相关性。
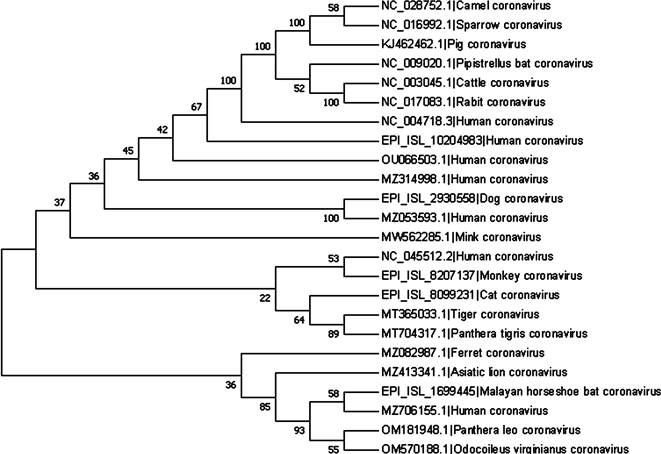
注:SARS冠状病毒(NC_004718.3);水貂Neovison vison (MW562285.1):马来亚虎Panthera tigris jacksoni (MT365033.1);亚洲狮Panthera leo persica (MZ413341.1);狮子Panthera leo (OM181948.1);白鼬Mustela ermine (MZ082987.1);虎Panthera tigris (MT704317.1);白尾鹿Odocoileus virginianus (OM570188.1);牛Bovini (NC 003045.1);骆驼Camelus (NC_028752.1);蝙蝠Pipistrellus (NC_009020.1);猪Sus (KJ462462.1);麻雀Passer (NC_016992.1);兔Rabbit (NC_017083.1);Alpha变异株(MZ053593.1);Beta变异株(MZ314998.1);Gamma变异株(OU066503.1);Delta变异株(Z706155.1);Omicron变异株(EPI_ISL_10204983);猫Felinae (EPI_ISL_8099231);狗Canis lupus familiaris (EPI_ISL_2930558);猴Primates (EPI_ISL_8207137);马来菊头蝠Rhinolophus malayanus (EPI_ISL_1699445);Wuhan-Hu-1 (NC_045512.2)
图4. 各类冠状病毒S蛋白的系统进化树
4. 讨论
新冠肺炎是当前国际社会关最受关注的公共卫生事件,SARS-CoV-2的快速传播危及全世界人民的生命健康及全球经济的发展 [14] [15]。2021年11月首次发现的Omicron变异株,不仅传染速度高于其它新冠病毒变异株,而且容易让康复后的感染者再次感染 [16]。S蛋白对于冠状病毒入侵宿主细胞至关重要 [17],其S1亚基的RBD结合域能与人ACE2结合,协助病毒穿过细胞膜进入宿主细胞,进而感染人类,因此RBD区域变异位点与新冠病毒传染性强弱密切相关 [18] [19] [20]。本研究显示,Omicron变异株与原始新冠病毒Wuhan-Hu-1相比,S蛋白中发现26个氨基酸突变位点,其中16个变异位点位于RBD区域。Shang等报道新冠病毒RBD区域内的417、449、487、489、493、500、501、502、505共9个变异位点与ACE2结合力直接相关 [21],本研究发现K417N、Q493R、N501Y、Y505H 4个相同的变异位点,这些变异位点可能增强新冠病毒与人ACE2的结合能力,进而提高病毒感染性 [22]。
Omicron变异株的4个关键变异位点中,N501Y变异位点广泛存在于Alpha、Beta、Gamma等高传染性变异株中 [23]。Frampton等人研究证明,N501位氨基酸是冠状病毒受体结合域的关键接触残基之一,直接参与RBD和ACE2的结合作用,而N501Y突变增强了RBD和ACE2的结合亲和力 [24]。刘洋等研究表明,N501Y变异位点能增加新冠病毒S蛋白对上呼吸道细胞受体的亲和力,在巴西、南非等国的趋同进化表明,N501Y是最主要的关注适应性突变位点,是上呼吸道感染和冠状病毒传播性增强的关键因素 [25]。Q493能与S蛋白中和抗体LY-CoV555相结合从而形成三个稳定力,当S蛋白发生Q493R突变时,会导致该位点与抗体无法形成相互作用力,表明Q493R突变体可能是LY-CoV555 逃逸突变体。K417N突变不直接与ACE2相互作用,但其有助于病毒更有效地与hACE2结合 [3]。
5. 结论
综上所述,本研究对Omicron变异株S蛋白变异位点进行分析,与其他新型冠状病毒变异株的S蛋白进行多序列比对及构建系统进化树,有助于深入了解Omicron变异株S蛋白的功能,为后续研究、筛选靶向S蛋白的新型抗病毒药物提供理论依据。
基金项目
广东省大学生创新训练计划项目(S202010580047)。