1. 引言
蛋白质是基因表达的产物,参与几乎所有生物活动,是生物体中不可缺少的物质 [1] 。其中,某些蛋白质对生物的生命维持、繁衍及生理调控有着至关重要的影响,并在疾病的形成和进展中扮演着核心角色,我们通常将这些蛋白质称为关键蛋白质 [2] 。因此,在疾病的预防和治疗中,准确而迅速地定位这些关键蛋白质靶点变得格外关键。
传统的识别关键蛋白质的方法主要依靠生物学实验,例如RNA干扰 [3] ,单基因敲除 [4] 、条件性基因剔除 [3] 等,这些方法虽然准确性较高,但实验过程复杂、成本昂贵、实验周期长,而且适用于有限的物种。近年来,高通量蛋白质技术的发展为积累了大量的蛋白质相互作用(PPI)数据提供了条件,如蛋白质相互作用数据库(Databaseof Interacting Protein, DIP),美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)等。这些数据库的建立为研究人员设计计算方法识别关键蛋白质奠定了数据基础。随着蛋白质数据的累积,PPI网络的构建为理解蛋白质复杂性、生物通路和细胞组织功能提供了新的视角 [5] [6] 。一系列基于PPI拓扑结构的中心性方法被提出,例如,Degree Centrality (DC [7] )方法采用度中心性,Subgraph Centrality (SC [8] )采用子图中心性,Eigenvector Centrality (EC [9] )采用特征向量中心性,Information Centrality (IC [10] )引入信息中心性,Neighborhood Centrality (NC [11] )引入邻居中心性,Local Average Connectivity (LAC [12] )引入局部特征中心性。基于PPI网络拓扑特性提出的一系列方法虽然有效地提高了关键蛋白质识别率,但是蛋白质相互作用数据本身具有很高的假阳性,使得基于拓扑结构的方法存在局限性。为了解决上述问题,更多的研究者提出将蛋白质相互作用网络的拓扑特征与生物特征相结合 [13] [14] [15] [16] [17] 。一种名为RWHN [18] 的方法通过整合加权蛋白质–蛋白质相互作用网络和域–域关联网络,构建了一个异构网络来识别必需蛋白质。Lei等人提出了RWEP [19] 算法,该算法首先利用基因表达信息和基因本体注释信息构建加权的蛋白质相互作用网络,然后采用随机游走算法来度量蛋白质的关键性。这些算法利用蛋白质内在的生物学特性优化网络结构,消除噪声对原始网络数据的影响,识别效果显著增强。
生物学网络分析已成为生物信息界和计算机界的热门话题。多头注意力机制作为一种强大的特征识别方法在生物网络分析中崭露头角。是自注意力机制的一种扩展,主要用于提高模型在处理不同位置间关系时的能力。同时,多头注意力机制允许模型自适应地关注网络中不同节点的信息,根据节点之间的相关性分配不同的注意力权重。这种机制使得模型能够更好地识别关键的节点和边,从而提高生物网络分析的准确性和解释性,更快地识别关键蛋白质。
与以往不同的是,本文提出一种基于多头注意力机制的关键蛋白质靶点识别方法(EPMHG)。该方法将基因表达谱和同源性特征与PPI网络拓扑特征融合,构建融合PPI网络,引入图注意力神经网络学习融合PPI网络中的节点表示,为了使得模型可以在多个表示空间中并行地捕获不同信息,增强模型的学习能力,多头注意力机制被采用,这可以有效的改进识别蛋白质的精度。与现存的基于拓扑特征和随机游走策略的方法相比,EPMHG方法在识别精度上更胜一筹。
2. 关键蛋白质识别架构
2.1. 基于多头注意力机制的关键蛋白质识别框架
本文提出一种基于多头注意力机制的关键蛋白质靶点识别方法(EPMHG)。PPI网络拓扑特征和基因表达谱、同源相似性相融合作为蛋白质的初始特征从而构建一个融合PPI网络。随后引入图注意力神经网络对融合PPI网络进行节点学习,为了更全面地捕获蛋白质之间的关联关系信息,我们采用多头注意力机制改善模型的学习能力,进而使得关键蛋白质识别精度得到改善。如图1所示,基于多头注意力机制的关键蛋白质靶点识别框架图。
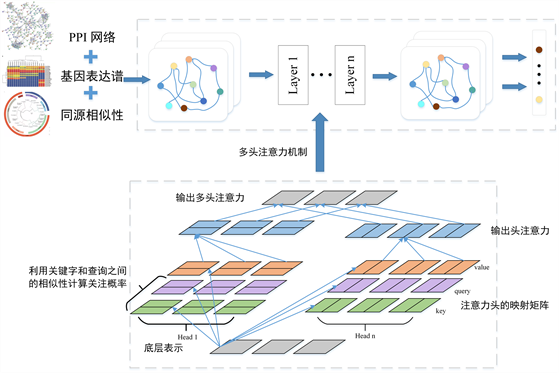
Figure 1. A framework for identifying essential proteins based on multi-head attention mechanisms
图1. 基于多头注意力机制的关键蛋白质识别框架图
2.2. 构建PPI网络
细胞中的蛋白质不是独立的,它们相互作用,构成PPI网络。PPI网络是一种重要的生物网络,满足复杂网络(如小世界、无标度和模块化)的拓扑特性。构建一个无向图
,其中,V为节点集,E为边集。在这个网络中,节点集由不同的蛋白质组成。当且仅当蛋白质相互作用时,在PPI网络中每对蛋白质之间都有一条边。
2.3. 特征公式化
2.3.1. 计算边聚集系数
据研究表明,关键蛋白质更频繁的与其他关键蛋白质相连。通过分析关键蛋白质之间相互作用的拓扑特性,他们发现了一个几乎完全连通的指数网络,这意味着蛋白质的关键性与其相邻蛋白质的关键性之间存在着很强的相关性。
因此,我们将边聚集系数概念引入蛋白质相互作用网络中预测关键蛋白质 [20] 。边聚集系数描述了一条边的两个连接节点与其周围其他节点之间的紧密程度,该系数不仅可以评价边缘在PPI网络中的重要性,又可以描述两个蛋白质之间的紧密程度。我们定义u和v两个节点边距集系数公式如下:
(1)
其中,
指的是相互作用的边
在PPI网络中形成的实际的三角形的数量,
表示的是相互作用的边
在PPI网络中最大可能会构成的三角形数量。
2.3.2. 基因表达相似性
基因表达是一个基因被表达为功能性基因产物的过程,这些产物通常是蛋白质。基因表达也被广泛用于鉴定关键蛋白质。对于一个给定的基因u,它在不同时间的基因表达可以通过一个载体来表达,。
为基因μ在i时刻的表达量。
PCC是用来衡量两个变量之间线性相关的简单相关系数 [21] ,其值在[−1, 1]之间,我们引入PCC来表征基因共表达的相似性,它在自然科学中被广泛应用。对于u和v基因,他们之间的计算如下:
(2)
式中
表示v基因在各个时刻的平均表达量,
表示v基因在各个时刻的表达量的标准方差。如果为正,则基因
正相关,如果值为负,则基因
呈负相关。
2.3.3. 计算同源相似性
直系同源物是指源自共同祖先的同源蛋白质,它们通常具有高度相似的氨基酸序列,并保留相同或非常相似的功能.许多研究使用同源信息来识别PPI网络的进化信,发现蛋白质进化速率,推断蛋白质保守性 [22] 。
给定一个PPI网络
,设S为参考物种集,用于获取
的同源信息,s表示S集的元素。对于蛋白质
,其同源分数
被定义为蛋白质
在参考物种集中具有同源物的数量,其中
。对同源相似性
进行归一化处理,公式为:
(3)
其中,
表示G中所有蛋白质的
最大值。如果蛋白质
在S物种集中的所有生物都存在同源关系,则同源得分为1。相反,如果蛋白质
没有和任何生物存在同源关系,同源得分为0。
3. EPMHG方法原理
3.1. 构建特征集合
EPMHG方法通过融合PPI网络拓扑特征与基因表达特征、同源相似性特征,突出蛋白质之间强关联关系。边聚集系数(ECC)强调蛋白质之间的紧密程度,皮尔逊系数(PCC)衡量两个蛋白质之间线性相关性,同源相似性(NOS)推断蛋白质的保守性。PPI网络被定义为一个无向图
,V表示节点集,其中包含N个蛋白质节点。ECC, PCC, NOS作为每个蛋白质节点的初始特征,因此,获得一个3维的蛋白质节点特征集合,可表示为
,
,
。
3.2. 计算注意力分数
注意力层测目的是输出新的节点特征集,
,
。在这个过程中特征向量的维度可能会发生改变,即
。为了保留足够的表达能力,将输入特征转化为高阶特征,至少需要一个可学习的线性变换。对于蛋白质节点
,它们的特征
应用线性变换
,从而F维转化为
维新特征
:
(4)
在将输入特征运用线性变换转换为高阶特征后,使用自注意力为每个节点分配注意力权重。其中,a表示一个共享注意力机制:
,用于计算注意力系数
,也就是蛋白质节点
对
的影响力系数。
注意力机制的计算考虑了每个蛋白质节点对目标节点的影响。对于目标节点
,只计算其邻域内的节点
对目标节点的相似度
。
为了更好的在不同节点之间分配权重,我们需要将目标节点与所有邻居计算出来的相关度进行统一的归一化处理,这里用softmax归一化:
(5)
关于a的选择,可以用向量的内积来定义一种无参形式的相关度计算
,也可以定义成一种带参的神经网络层,只要满足a:
,即输出一个标量值表示二者的相关度即可。在论文实验中,a是一个单层前馈神经网络,参数为权重向量
,使用负半轴斜率为0.2的LeakyReLU作为非线性激活函数:
(6)
其中‖表示拼接操作。完整的权重系数计算公式为:
(7)
得到归一化注意系数后,计算其duiyingtezheng1的线性组合,通过非线性激活函数后,每个接待的最后输出特征向量为:
(8)
3.3. 多头注意力机制
本文使用多头注意力机制来增强模型的学习能力,即对公式(8)调用K组相互独立的注意力机制,然后将输出结果拼接起来:
(9)
其中,‖表示拼接操作。
是第k组注意力机制计算出的权重系数,
是对应的输入线性变换矩阵,最终输出的节点特征向量
包含了
个特征。为了减少输出的特征向量的维度,也可以将拼接操作替换为平均操作。
(10)
4. 仿真实验
4.1. 实验数据
酵母蛋白质相互作用数据是目前使用最广泛的生物数据,实验以酵母蛋白质相互作用网络作为研究对象。本文选取来源于DIP (Databaseof Interacting Protein)数据库的蛋白质相互作用数据集来进行构建PPI网络。滤除数据集中重复的蛋白质,DIP数据集中包含5093个蛋白质,1167个关键蛋白质和24,743对相互作用边。本文在实验部分提出将EPMHG方法与现存的六种经典方法进行比较,分别是Degree Centrality (DC [7] , Subgraph Centrality (SC [8] ), Eigenvector Centrality (EC [9] ), Information Centrality (IC [10] , Neighborhood Centrality (NC [11] ), Local Average Connectivity (LAC [12] )。
4.2. 实验结果对比及分析
4.2.1. 识别数量
为了验证EPMHG算法在识别数量的表现,分别选择DIP数据集中蛋白质排名在Top 1%、Top 5%、Top 10%、Top 15%、Top 20%、Top 25%的蛋白质数量作为关键蛋白质,然后比较每种测度参数的预测结果中真实关键蛋白质的数量。如图2所示,EPMHG算法在六个测度上的预测关键蛋白质数量分别为50、213、373、514、636、723,根据数量比较,该方法在预测数量上均高于其余六种方法。这主要的原因是相比较的六种方法(DC, SC, IC, EC, LAC, NC)是基于PPI网络拓扑结构特征提出的,由于PPI网络本身具有较高的假阳性和假阴性,这使得预测到限制。相比较而言,EPMHG算法是融合了PPI网络拓扑特征、基因表达特征和同源相似性特征,使得蛋白质节点信息更全面,因此预测的数量远高于其余六种方法。
4.2.2. 精确度对比
为了进一步评估EPMHG算法预测关键蛋白质预测的准确度。本小节计算了敏感度(Sensitivity, SN),正确率(Accuracy, ACC),特异性(Specificity, SP),阳性预测值(Positive Predictive Value, PPV),F-测度(F-measure),阴性预测值(Negative Predictive Value, NPV)六个指标对EPMHG算法与其余六种方法进行了比较。六种指标的计算公式表示如下:
(11)
(12)
(13)
(14)
(15)
(16)
其中,TP (true positive)表示关键蛋白质被正确预测为关键蛋白质的数量,FN (false negative)表示关键蛋白质被错误地预测为非关键蛋白质的数量TN (true positive)表示关键蛋白质被正确预测为非关键蛋白质的数量,FN (false negative)表示非关键蛋白质被错误预测为关键蛋白质的数量。
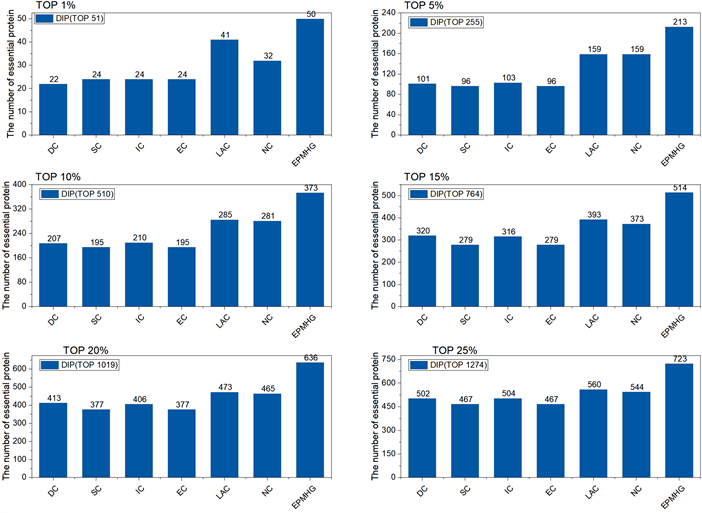
Figure 2. The number of essential protein identifications
图2. 关键蛋白质识别数量图
该部分选择DIP数据集的TOP 25%作为计算六个指标的数据,最终对比结果呈现在表1中。由表1可知,EPMHG算法的准确率ACC为0.775,这超过了相比的其余六种方法。该方法采用了多头注意力机制增强了模型的学习能力,使得模型可以更准确地预测关键蛋白质。

Table 1. Comparison of the six metrics in the DIP dataset
表1. 比较DIP数据集的六种指标
4.2.3. PR曲线仿真
本文也研究了EPMHG算法的Precision-recall (PR)曲线,并将其与其他六种方法进行了比较,选择DIP数据集的TOP 25%作为验证数据。如图3所示,X轴表示 Recall,Y轴表示Precision。Top n个蛋白质的精确度和召回率被定义为:
(17)
(18)
其中,
表示从
个蛋白质里识别出的关键蛋白质数量,
表示从
个蛋白质里识别出的非关键蛋白质数量,
表示DIP数据集的关键蛋白质数量。同时也计算了PR曲线下面积,这有助于更准确地比较各种方法在精确度的优势。从图3的仿真结果可知,EPMHG算法的PR曲线高于其余六种方法,这表明该方法有较高的识别准确率。EPMHG算法引入图注意力神经,该网络的学习策略是识别并重点关注与当前节点更相关或更重要的邻居,进而更准确识别关键蛋白质。
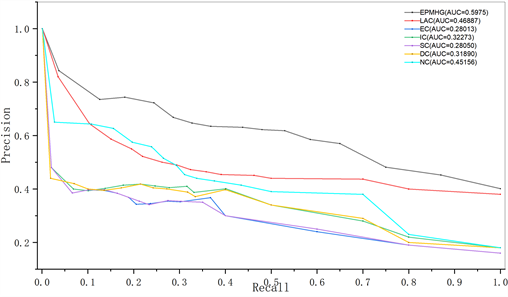
Figure 3. The precision-recall simulation figure
图3. 精度–召回率(PR)仿真图
5. 结论
本文提出了一种结合多头注意力机制来识别关键蛋白质靶点的方法(EPMHG)。通过整合蛋白质相互作用网络的拓扑特征和生物信息构建融合PPI网络,并引入多头注意力机制增强图注意力神经网络模型的学习效果,最终,在DIP酵母蛋白数据集上成功地验证了该方法的识别精度。该方法的提出为蛋白质关键靶点的快速识别提供了一种更为有效的策略,并为未来的疾病预防和治疗提供了新视角。