1. 引言
创伤性脑损伤(traumatic brain injury, TBI)是一种由外力导致的脑组织损伤,包括高空坠物、交通事故、暴力等意外原因引起的脑组织损坏及神经细胞坏死 [1]。近年来,TBI的发生率受交通运输和工业水平飞速发展的影响而进一步上升,TBI作为一种常见的神经外科疾病,已成为导致世界范围内死亡和残疾的主要原因之一,并严重威胁了人群生命健康 [2]。同时,TBI也因其高发生率、高致残率、高死亡率的特点,一直受到国内外学者的密切关注 [3]。
微小RNA(microRNA, miRNA)是一类广泛存在于真核生物中的内源性单链小分子RNA,长度在21~25个碱基之间。当前已被证明,miRNA作为非编码RNA参与基因表达的调控中发挥着关键作用。近年来,有研究通过微阵列分析发现了神经损伤组织中miRNA表达模式的动态变化,并确定了miRNA参与神经损伤和再生过程 [4]。
当前,TBI的临床诊断主要依靠神经系统检查和影像学技术,具有一定的局限性,对于不同程度的脑损伤至今没有诊断的金标准 [5];而miRNA这种参与神经损伤修复的能力使得其成为TBI临床诊断的生物标志物成为可能。于是,本研究旨在通过生物信息学分析,筛选TBI后与差异表达基因相关的miRNA,以期寻找TBI临床诊断的客观指标;并构建gene-miRNA网络,从而对TBI分子水平调节做出整体性预测,为寻找其治疗靶点提供理论基础。
2. 材料与方法
2.1. 材料
通过美国国立生物技术信息中心(NCBI)平台的基因表达综合数据库GEO (https://www.ncbi.nlm.nih.gov/geo/),在大鼠物种下以“创伤性脑损伤(TBI)”为关键词寻找相关数据芯片,分别检索有关miRNA及基因的原始数据。其他选择标准包括:1) 含有假手术与实验组相关对照;2) 含有多组实验样本;3) 样本支持线上分析工具GEO2R。根据选择标准,筛选出涉及大鼠脑组织创伤性损伤手术后有关miRNA和基因的四组数据集。本次实验选取含有miRNA相关表达量变化的两个数据集(GSE59646和GSE115614-GPL22696)和基因表达量变化的两个数据集(GSE115614-GPL15084和 GSE59647-GPL14746)作为后期分析的样本。
2.2. 筛选差异表达基因和miRNA
在本研究中,利用GEO (Gene Expression Omnibus)数据库自带的交互式线上分析工具GEO2R对GSE59646、GSE115614-GPL22696、GSE115614-GPL15084和GSE59647-GPL14746四个数据集进行分析,在miRNA及基因表达数据中比较出实验组与假手术组中存在差异表达的信息,分别对miRNA及基因各两组数据集的差异数据取交集,以P < 0.05,|log2FC| > 1为筛选条件,筛选出差异表达的基因和miRNA。
2.3. 差异表达miRNA的靶基因预测
为了探究差异表达的miRNA与差异基因的靶向关系,本研究通过miRwalk 3.0软件 (http://mirwalk.umm.uni-heidelberg.de/)对差异表达的miRNA进行靶基因预测,筛选标准为P < 0.05,将系统提供的靶基因与差异表达基因进行比较,保留重合的差异表达基因。
2.4. miRNA-Gene靶向调控网络的构建
为了讨论TBI后分子水平调节的相互作用,利用cytoscape v3.7.1软件,通过输入差异表达数据,自动生成初步的miRNA-Gene调控网络图。再通过导入差异表达上下调关系(根据log2FC值得出上调和下调结果),对不同属性的网络节点(Node)进行标注与区分,并将边(Edge)和网络(Network)样式进行调整,绘制一个清晰的miRNA-Gene靶向网络布局图。
2.5. 差异表达基因的基因本体论(GO)功能富集分析
将与差异表达miRNA靶向基因重合的差异表达基因导入在线分析软件DAVID (https://david.ncifcrf.gov/)进行GO功能富集分析,筛选标准为P < 0.05,从生物学过程(BP, Biological Process)、细胞成分(CC, Cellular Component)及分子功能(MF, Molecular Function)三个角度,对这些基因的功能进行描述,从而对TBI分子水平调节做出整体性预测。
3. 结果
3.1. 大鼠创伤性脑损伤后典型差异表达的基因及miRNA
将基因芯片数据库集GSE59646和GSE115614-GPL22696中的四例假手术组作为空白组、六例TBI手术组作为对照组分别进行差异表达分析,分别筛选出375、1120个差异表达miRNA,将两组数据集基因取交集,共筛选出4个差异miRNA,其中4个miRNA表达上调、无miRNA表达下调。运用相同的步骤进行操作,在GSE115614-GPL15084和GSE59647-GPL14746两组差异基因数据集中,取交集后共筛选出608个差异基因,其中411个基因表达上调、197个基因表达下调(见图1)。
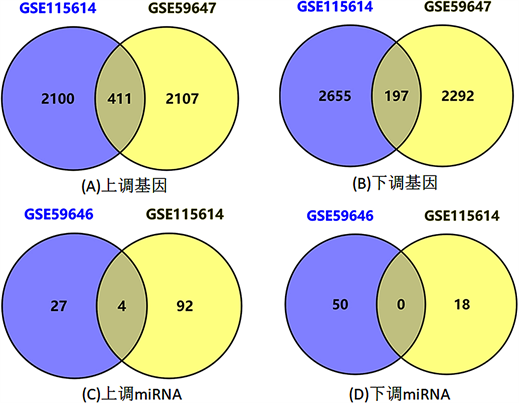
Figure 1. Differentially expressed genes and miRNAs after traumatic brain injury in rats
图1. 大鼠创伤性脑损伤后差异表达的基因及miRNA
3.2. 大鼠创伤性脑损伤后差异表达miRNA的靶基因预测
利用miRwalk软件对筛选miRNA的靶基因进行预测,将靶基因与筛选基因进行比较,得到重合的差异表达基因共95个。表1展示了筛选出的差异表达miRNA及其靶向的差异基因,并标明了其差异表达的上下调情况。

Table 1. Differentially expressed miRNAs and differentially targeted genes
表1. 差异表达miRNA及其靶向的差异基因
3.3. miRNA-Gene靶向调控网络的构建
利用从miRwalk 3.0软件预测出的靶基因,将系统提供的靶基因与差异表达基因进行比较,保留重合的差异表达基因,并使用Cytoscape 3.7.1对获得的两组数据构建了miRNA-Gene靶向调控网络(见图2),其中共加入了4条miRNA和与之对应的95个靶向基因,通过红色来标记基因或miRNA的上调、绿色标记下调。该网络展示了创伤性脑损伤后相关miRNA与其靶基因是如何通过相互作用而进一步产生一系列生物学效应的。本研究试图通过对网络中核心节点的分析,从而对miRNA与其靶基因的调控在整个创伤性脑损伤生物学过程中所产生的影响进行了相关预测和描述,并借此讨论其相关的生物学意义。
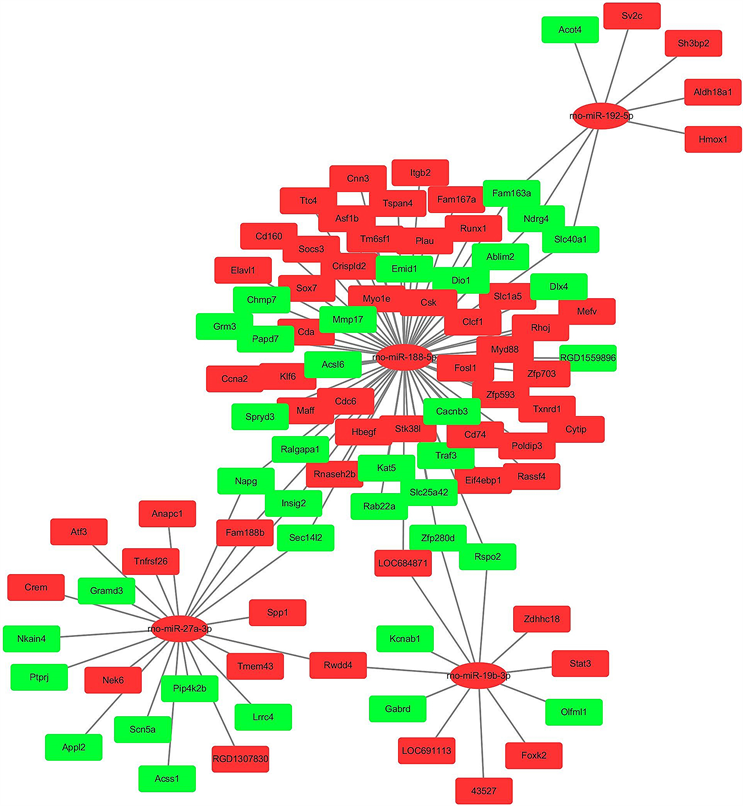
Figure 2. miRNA gene targeting regulatory network
图2. miRNA-Gene靶向调控网络
3.4. 差异表达基因的本体论功能富集分析结果
在本研究中,miRNA的靶向基因通过DAVID软件中基因本体论功能富集分析得到的结果,差异表达基因中CDC6、RNASEH2B、CCNA2、CD74在成纤维细胞正调节(positive regulation of fibroblast proliferation)的生物学进程中起作用,CLCF1、SOCS3、STAT3在JAK-STAT信号转导途径(JAK-STAT cascade)中起作用,PTPRJ、HMOX1、SOX7等7个基因在对细胞增殖的负调节(negative regulation of cell proliferation)中发挥作用。根据P值大小排序,GO分析中生物学过程(BP)富集结果及相关基因数量结果见图3。
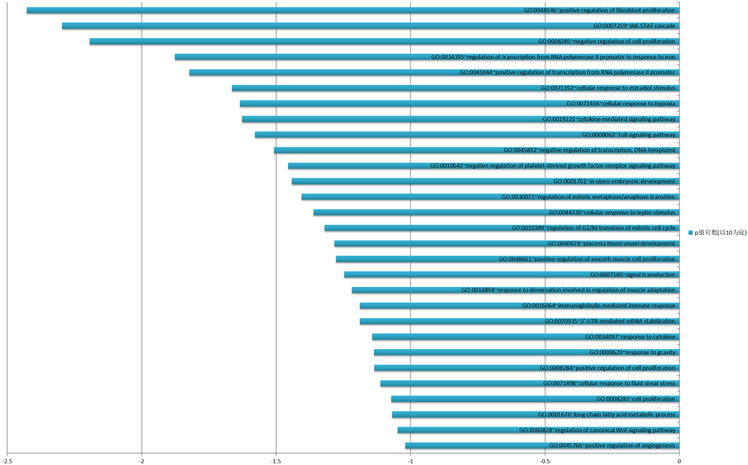
Figure 3. Functional enrichment of differentially expressed gene GO—BP results of biological process
图3. 差异表达基因GO功能富集——生物学过程BP结果
差异表达基因在细胞内(intracellular)、核质(nucleoplasm)、肌动蛋白细胞骨架(actin cytoskeleton)等处发挥作用。根据P值大小排序,GO分析中细胞成分(CC)富集结果及相关基因数量结果见图4。
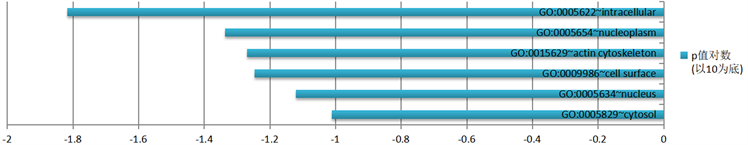
Figure 4. Functional enrichment of differentially expressed gene GO—cell component CC results
图4. 差异表达基因GO功能富集——细胞组分CC结果
差异表达基因在蛋白质激酶结合(protein kinase binding)、抑制转录因子结合(repressing transcription factor binding)、受体结合(receptor binding)等分子功能中起作用。根据P值大小排序,GO分析中分子功能(MF)富集结果及相关基因数量结果见图5。
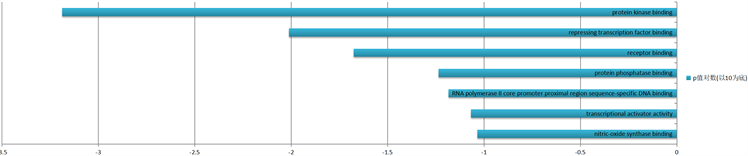
Figure 5. Functional enrichment of differentially expressed gene go—results of molecular function MF
图5. 差异表达基因GO功能富集——分子功能MF结果
4. 讨论
自Ambros和Ruvkun [6] 各自团队发现第一个microRNA开始,microRNA的研究至今可以大致分为三个阶段:第一阶段是从最初的发现到了解它广泛参与生命活动从发生到生长发育的各个时期;第二阶段,从分子水平层面揭示了成熟的miRNA可与其特异性mRNA分子部分互补,从而发挥其下调基因表达的作用,调节蛋白质的表达水平;第三阶段,miRNA分子病理学研究的大量实验证明miRNA与肿瘤及各类疾病有关,由此展开针对诊断标志物及病理机制的研究。
创伤性脑损伤作为神经外科最常见疾病,多数会引起继发性脑损伤,涉及氧化应激、炎症、神经元凋亡坏死、轴突损伤等诸多方面,并以细胞及突触的退化而告终。目前临床上尚没有诊断TBI的分子标志物,过去很长一段时间,许多分子被提出可作为TBI的生物标志物,但由于神经系统的复杂和TBI病情程度的不同,在临床应用上并不理想 [7]。近年来,大量实验证明miRNA与TBI上述生物学活动有关 [8],本研究通过生物信息学分析,实现了利用数据库筛选扩充miRNA调节TBI网络的认识。
结合本研究最终筛选出的95个差异表达基因和4个靶向miRNA,构建完成了miRNA-Gene靶向调控网络,并通过GO功能富集分析,得到了从生物学过程、细胞成分及分子功能三个角度对这些基因进行的功能描述。其中,值得注意的是,多个基因靶向对应miRNA参与了炎症反应、血管生成、以细胞坏死凋亡为主的继发性脑损伤和突出可塑性等生物过程 [9] [10]。
基因表达水平显示,CDC6,RNASEH2B,CCNA2,CD74在TBI组中相比对照组明显上调,并均与miRNA-188-5p存在靶向关系,共同参与成纤维细胞增殖的正向调节,由此推测miRNA-188-5p下调在TBI后可能促进了成纤维细胞的增殖,相关方面还应设计实验加以证明研究。此外,HMOX1,ITGB2,RUNX1在TBI组中同样表达上调,并分别靶向下调了miRNA-192-5p和miRNA-188-5p共同参与对血管生成素的正向调节,近年来,多项实验证明血管生成是脑损伤后神经修复与再生的关键因素,miRNA在其中可以起到调控血管生成的功能 [11]。
在本研究中值得注意的还有MYD88,CD74参与免疫球蛋白介导的免疫反应以及由LRRC4,KLF6,MYD88,SOCS3参与细胞因子介导的信号转导通路,二者在分子机制上同炎症反应均有相关性。此外,有研究表明miRNA-1224可以调节肿瘤坏死因子α (tumornecrosis factor-α, TNF-α),由此说明miRNA-1224可能参与了炎症反应 [12]。另一项研究证明,miRNA-124能诱导小胶质细胞炎症反应显著缓解,可见miRNA-124参与炎症反应的调控。此外miRNA-155,miRNA-414,miRNA-104,miRNA-125,miRNA-146和miRNA-21均参与炎症因子的表达 [13] [14]。因此,有理由推断部分miRNAs参与了中枢神经系统的炎症反应调节。
必须指出,本研究的实验结果可能依旧存在不足,有研究表明,miRNA在执行生物功能时常常表现出时空特异性,在TBI后的不同时间、脑组织不同部位遭到外力创伤,都会对miRNA的表达量产生影响 [15] [16]。因此,对TBI进程中miRNA调控更详尽的机制还有待后续研究。生物信息学分析所给出的4个miRNA是否能够成为TBI的生物标志物,同样需要更多基础研究的实验证明;但总的来说,本研究所得出的miRNA-gene的靶向关系无疑将有助于指导我们研究更复杂的TBI分子机制和miRNA调控机理,为我们开展进一步的实验设计提供帮助。